+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structural insights into the activation mechanism of antimicrobial GBP1: Membrane-bound GBP1 oligomer | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Oligomer / GTPase / Interferon-induced / ANTIMICROBIAL PROTEIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
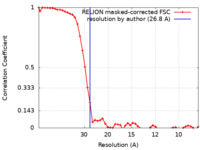
| 手法 | サブトモグラム平均法 / クライオ電子顕微鏡法 / 解像度: 26.8 Å | |||||||||
 データ登録者 データ登録者 | Weismehl M / Chu X / Kutsch M / Lauterjung P / Herrmann C / Kudryashev M / Daumke O | |||||||||
| 資金援助 |  ドイツ, 2件 ドイツ, 2件
| |||||||||
 引用 引用 |  ジャーナル: EMBO J / 年: 2024 ジャーナル: EMBO J / 年: 2024タイトル: Structural insights into the activation mechanism of antimicrobial GBP1. 著者: Marius Weismehl / Xiaofeng Chu / Miriam Kutsch / Paul Lauterjung / Christian Herrmann / Misha Kudryashev / Oliver Daumke /   要旨: The dynamin-related human guanylate-binding protein 1 (GBP1) mediates host defenses against microbial pathogens. Upon GTP binding and hydrolysis, auto-inhibited GBP1 monomers dimerize and assemble ...The dynamin-related human guanylate-binding protein 1 (GBP1) mediates host defenses against microbial pathogens. Upon GTP binding and hydrolysis, auto-inhibited GBP1 monomers dimerize and assemble into soluble and membrane-bound oligomers, which are crucial for innate immune responses. How higher-order GBP1 oligomers are built from dimers, and how assembly is coordinated with nucleotide-dependent conformational changes, has remained elusive. Here, we present cryo-electron microscopy-based structural data of soluble and membrane-bound GBP1 oligomers, which show that GBP1 assembles in an outstretched dimeric conformation. We identify a surface-exposed helix in the large GTPase domain that contributes to the oligomerization interface, and we probe its nucleotide- and dimerization-dependent movements that facilitate the formation of an antimicrobial protein coat on a gram-negative bacterial pathogen. Our results reveal a sophisticated activation mechanism for GBP1, in which nucleotide-dependent structural changes coordinate dimerization, oligomerization, and membrane binding to allow encapsulation of pathogens within an antimicrobial protein coat. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_18806.map.gz emd_18806.map.gz | 1.2 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-18806-v30.xml emd-18806-v30.xml emd-18806.xml emd-18806.xml | 15.8 KB 15.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_18806_fsc.xml emd_18806_fsc.xml | 4.7 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_18806.png emd_18806.png | 92.8 KB | ||
| マスクデータ |  emd_18806_msk_1.map emd_18806_msk_1.map | 8 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-18806.cif.gz emd-18806.cif.gz | 6.1 KB | ||
| その他 |  emd_18806_half_map_1.map.gz emd_18806_half_map_1.map.gz emd_18806_half_map_2.map.gz emd_18806_half_map_2.map.gz | 7.4 MB 7.4 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-18806 http://ftp.pdbj.org/pub/emdb/structures/EMD-18806 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-18806 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-18806 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8r1aMC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_18806.map.gz / 形式: CCP4 / 大きさ: 8 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_18806.map.gz / 形式: CCP4 / 大きさ: 8 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 4.4 Å | ||||||||||||||||||||||||||||||||||||
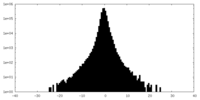
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_18806_msk_1.map emd_18806_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
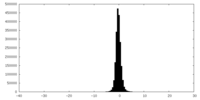
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_18806_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
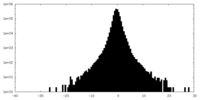
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_18806_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
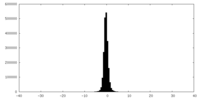
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Membrane-bound oligomer of human guanylate-binding protein 1 (GBP1)
| 全体 | 名称: Membrane-bound oligomer of human guanylate-binding protein 1 (GBP1) |
|---|---|
| 要素 |
|
-超分子 #1: Membrane-bound oligomer of human guanylate-binding protein 1 (GBP1)
| 超分子 | 名称: Membrane-bound oligomer of human guanylate-binding protein 1 (GBP1) タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 / 詳細: in complex with GDP-AlFx |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
-分子 #1: Guanylate binding protein 1
| 分子 | 名称: Guanylate binding protein 1 / タイプ: protein_or_peptide / ID: 1 / コピー数: 6 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 69.26793 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MRGSHHHHHH GSASEIHMTG PMCLIENTNG RLMANPEALK ILSAITQPMV VVAIVGLYRT GKSYLMNKLA GKKKGFSLGS TVQSHTKGI WMWCVPHPKK PGHILVLLDT EGLGDVEKGD NQNDSWIFAL AVLLSSTFVY NSIGTINQQA MDQLYYVTEL T HRIRSKSS ...文字列: MRGSHHHHHH GSASEIHMTG PMCLIENTNG RLMANPEALK ILSAITQPMV VVAIVGLYRT GKSYLMNKLA GKKKGFSLGS TVQSHTKGI WMWCVPHPKK PGHILVLLDT EGLGDVEKGD NQNDSWIFAL AVLLSSTFVY NSIGTINQQA MDQLYYVTEL T HRIRSKSS PDENENEVED SADFVSFFPD FVWTLRDFSL DLEADGQPLT PDEYLTYSLK LKKGTSQKDE TFNLPRLCIR KF FPKKKCF VFDRPVHRRK LAQLEKLQDE ELDPEFVQQV ADFCSYIFSN SKTKTLSGGI QVNGPRLESL VLTYVNAISS GDL PCMENA VLALAQIENS AAVQKAIAHY EQQMGQKVQL PTESLQELLD LHRDSEREAI EVFIRSSFKD VDHLFQKELA AQLE KKRDD FCKQNQEASS DRCSGLLQVI FSPLEEEVKA GIYSKPGGYR LFVQKLQDLK KKYYEEPRKG IQAEEILQTY LKSKE SMTD AILQTDQTLT EKEKEIEVER VKAESAQASA KMLQEMQRKN EQMMEQKERS YQEHLKQLTE KMENDRVQLL KEQERT LAL KLQEQEQLLK EGFQKESRIM KNEIQDLQTK MRRRKACTIS UniProtKB: Guanylate binding protein 1 |
-分子 #2: ALUMINUM FLUORIDE
| 分子 | 名称: ALUMINUM FLUORIDE / タイプ: ligand / ID: 2 / コピー数: 6 / 式: AF3 |
|---|---|
| 分子量 | 理論値: 83.977 Da |
| Chemical component information |  ChemComp-AF3: |
-分子 #3: MAGNESIUM ION
| 分子 | 名称: MAGNESIUM ION / タイプ: ligand / ID: 3 / コピー数: 6 / 式: MG |
|---|---|
| 分子量 | 理論値: 24.305 Da |
-分子 #4: GUANOSINE-5'-DIPHOSPHATE
| 分子 | 名称: GUANOSINE-5'-DIPHOSPHATE / タイプ: ligand / ID: 4 / コピー数: 6 / 式: GDP |
|---|---|
| 分子量 | 理論値: 443.201 Da |
| Chemical component information |  ChemComp-GDP: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | サブトモグラム平均法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.68 mg/mL |
|---|---|
| 緩衝液 | pH: 7.9 詳細: 50 mM Tris-HCl, 150 mM NaCl, 5 mM MgCl2 (supplemented with 200 uM GDP, 300 uM AlCl3, 10 mM NaF) |
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 2.8 e/Å2 詳細: Tilt series were acquired with a Hybrid-STA (Sanchez et al. 2020, Nat Commun): exposure dose of 2.8 e-/A2 for non-zero tilted projection and 14.4 e-/A2 for zero tilted projection |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 5.0 µm / 最小 デフォーカス(公称値): 2.0 µm / 倍率(公称値): 42000 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー





 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)