+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-1861 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Visualising an RNA genome poised for release from its receptor complex. | |||||||||
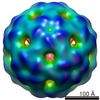
 マップデータ マップデータ | This is a cryo-EM icosahedrally-averaged reconstruction of bacteriophage MS2 unattached to F-pili. | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | MS2 / F-pili | |||||||||
| 生物種 |  Enterobacterio phage MS2 (ファージ) Enterobacterio phage MS2 (ファージ) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 24.0 Å | |||||||||
 データ登録者 データ登録者 | Toropova K / Stockley PG / Ranson NA | |||||||||
 引用 引用 |  ジャーナル: J Mol Biol / 年: 2011 ジャーナル: J Mol Biol / 年: 2011タイトル: Visualising a viral RNA genome poised for release from its receptor complex. 著者: Katerina Toropova / Peter G Stockley / Neil A Ranson /  要旨: We describe the cryo-electron microscopy structure of bacteriophage MS2 bound to its receptor, the bacterial F-pilus. The virus contacts the pilus at a capsid 5-fold vertex, thus locating the surface- ...We describe the cryo-electron microscopy structure of bacteriophage MS2 bound to its receptor, the bacterial F-pilus. The virus contacts the pilus at a capsid 5-fold vertex, thus locating the surface-accessible portion of the single copy of the pilin-binding maturation protein present in virions. This arrangement allows a 5-fold averaged map to be calculated, showing for the first time in any virus-receptor complex the nonuniform distribution of RNA within the capsid. Strikingly, at the vertex that contacts the pilus, a rod of density that may include contributions from both genome and maturation protein sits above a channel that goes through the capsid to the outside. This density is reminiscent of the DNA density observed in the exit channel of double-stranded DNA phages, suggesting that the RNA-maturation protein complex is poised to leave the capsid as the first step of the infection process. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_1861.map.gz emd_1861.map.gz | 10.5 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-1861-v30.xml emd-1861-v30.xml emd-1861.xml emd-1861.xml | 8.4 KB 8.4 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  1861_emdb.jpg 1861_emdb.jpg | 151.7 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-1861 http://ftp.pdbj.org/pub/emdb/structures/EMD-1861 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-1861 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-1861 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_1861_validation.pdf.gz emd_1861_validation.pdf.gz | 220.3 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_1861_full_validation.pdf.gz emd_1861_full_validation.pdf.gz | 219.4 KB | 表示 | |
| XML形式データ |  emd_1861_validation.xml.gz emd_1861_validation.xml.gz | 6.1 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1861 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1861 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1861 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1861 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_1861.map.gz / 形式: CCP4 / 大きさ: 29.8 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_1861.map.gz / 形式: CCP4 / 大きさ: 29.8 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | This is a cryo-EM icosahedrally-averaged reconstruction of bacteriophage MS2 unattached to F-pili. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.96 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Bacteriophage MS2 unattached to F-pili
| 全体 | 名称: Bacteriophage MS2 unattached to F-pili |
|---|---|
| 要素 |
|
-超分子 #1000: Bacteriophage MS2 unattached to F-pili
| 超分子 | 名称: Bacteriophage MS2 unattached to F-pili / タイプ: sample / ID: 1000 / 集合状態: single bacteriophage particle / Number unique components: 1 |
|---|
-超分子 #1: Enterobacterio phage MS2
| 超分子 | 名称: Enterobacterio phage MS2 / タイプ: virus / ID: 1 / Name.synonym: MS2 詳細: MS2 unattached to F-pili (which are also present in the sample). NCBI-ID: 12022 / 生物種: Enterobacterio phage MS2 / ウイルスタイプ: VIRION / ウイルス・単離状態: STRAIN / ウイルス・エンベロープ: No / ウイルス・中空状態: No / Syn species name: MS2 |
|---|---|
| 宿主 | 生物種:  |
| ウイルス殻 | Shell ID: 1 / 直径: 280 Å / T番号(三角分割数): 3 |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.8 / 詳細: 0.1 M Tris-HCl, 0.5 mM EDTA |
|---|---|
| グリッド | 詳細: Lacey carbon copper mesh grid |
| 凍結 | 凍結剤: ETHANE / チャンバー内温度: 22 K / 装置: HOMEMADE PLUNGER 詳細: Vitrification instrument: Double sided custom pneumatic blotter 手法: 1.6s blot |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TECNAI 20 |
|---|---|
| 撮影 | カテゴリ: FILM / フィルム・検出器のモデル: KODAK SO-163 FILM / デジタル化 - スキャナー: OTHER / デジタル化 - サンプリング間隔: 9.88 µm / 実像数: 252 / 平均電子線量: 18 e/Å2 / Od range: 1 / ビット/ピクセル: 16 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 倍率(補正後): 50400 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.0 mm / 倍率(公称値): 50000 |
| 試料ステージ | 試料ホルダー: Side entry / 試料ホルダーモデル: GATAN LIQUID NITROGEN |
- 画像解析
画像解析
| CTF補正 | 詳細: Phase flipping each particle |
|---|---|
| 最終 再構成 | 想定した対称性 - 点群: I (正20面体型対称) / アルゴリズム: OTHER / 解像度のタイプ: BY AUTHOR / 解像度: 24.0 Å / 解像度の算出法: FSC 0.5 CUT-OFF / ソフトウェア - 名称: Spider / 使用した粒子像数: 862 |
| 最終 2次元分類 | クラス数: 43 |
 ムービー
ムービー コントローラー
コントローラー














 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)





















