+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Helical reconstruction of yeast eisosome protein Pil1 bound to membrane composed of lipid mixture -PIP2/+sterol (DOPC, DOPE, DOPS, cholesterol 30:20:20:30) | |||||||||||||||
 マップデータ マップデータ | Helical map Type II -PIP2/ sterol (D1, rise=5.04, twist=-136.50) | |||||||||||||||
 試料 試料 |
| |||||||||||||||
 キーワード キーワード | BAR domain / lipid reconstitution / membrane microdomain / LIPID BINDING PROTEIN | |||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報protein localization to eisosome filament / eisosome filament / eisosome assembly / eisosome / lipid droplet / cell periphery / endocytosis / intracellular protein localization / mitochondrial outer membrane / lipid binding ...protein localization to eisosome filament / eisosome filament / eisosome assembly / eisosome / lipid droplet / cell periphery / endocytosis / intracellular protein localization / mitochondrial outer membrane / lipid binding / mitochondrion / plasma membrane / cytoplasm 類似検索 - 分子機能 | |||||||||||||||
| 生物種 |  | |||||||||||||||
| 手法 | らせん対称体再構成法 / クライオ電子顕微鏡法 / 解像度: 3.35 Å | |||||||||||||||
 データ登録者 データ登録者 | Kefauver JM / Zou L / Desfosses A / Loewith RJ | |||||||||||||||
| 資金援助 | European Union,  スイス, 4件 スイス, 4件
| |||||||||||||||
 引用 引用 |  ジャーナル: Nature / 年: 2024 ジャーナル: Nature / 年: 2024タイトル: Cryo-EM architecture of a near-native stretch-sensitive membrane microdomain. 著者: Jennifer M Kefauver / Markku Hakala / Luoming Zou / Josephine Alba / Javier Espadas / Maria G Tettamanti / Jelena Gajić / Caroline Gabus / Pablo Campomanes / Leandro F Estrozi / Nesli E Sen ...著者: Jennifer M Kefauver / Markku Hakala / Luoming Zou / Josephine Alba / Javier Espadas / Maria G Tettamanti / Jelena Gajić / Caroline Gabus / Pablo Campomanes / Leandro F Estrozi / Nesli E Sen / Stefano Vanni / Aurélien Roux / Ambroise Desfosses / Robbie Loewith /    要旨: Biological membranes are partitioned into functional zones termed membrane microdomains, which contain specific lipids and proteins. The composition and organization of membrane microdomains remain ...Biological membranes are partitioned into functional zones termed membrane microdomains, which contain specific lipids and proteins. The composition and organization of membrane microdomains remain controversial because few techniques are available that allow the visualization of lipids in situ without disrupting their native behaviour. The yeast eisosome, composed of the BAR-domain proteins Pil1 and Lsp1 (hereafter, Pil1/Lsp1), scaffolds a membrane compartment that senses and responds to mechanical stress by flattening and releasing sequestered factors. Here we isolated near-native eisosomes as helical tubules made up of a lattice of Pil1/Lsp1 bound to plasma membrane lipids, and solved their structures by helical reconstruction. Our structures reveal a striking organization of membrane lipids, and, using in vitro reconstitutions and molecular dynamics simulations, we confirmed the positioning of individual PI(4,5)P, phosphatidylserine and sterol molecules sequestered beneath the Pil1/Lsp1 coat. Three-dimensional variability analysis of the native-source eisosomes revealed a dynamic stretching of the Pil1/Lsp1 lattice that affects the sequestration of these lipids. Collectively, our results support a mechanism in which stretching of the Pil1/Lsp1 lattice liberates lipids that would otherwise be anchored by the Pil1/Lsp1 coat, and thus provide mechanistic insight into how eisosome BAR-domain proteins create a mechanosensitive membrane microdomain. | |||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_18308.map.gz emd_18308.map.gz | 944.7 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-18308-v30.xml emd-18308-v30.xml emd-18308.xml emd-18308.xml | 23.6 KB 23.6 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_18308.png emd_18308.png | 206.2 KB | ||
| マスクデータ |  emd_18308_msk_1.map emd_18308_msk_1.map | 1000 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-18308.cif.gz emd-18308.cif.gz | 6.3 KB | ||
| その他 |  emd_18308_additional_1.map.gz emd_18308_additional_1.map.gz emd_18308_additional_2.map.gz emd_18308_additional_2.map.gz emd_18308_additional_3.map.gz emd_18308_additional_3.map.gz emd_18308_half_map_1.map.gz emd_18308_half_map_1.map.gz emd_18308_half_map_2.map.gz emd_18308_half_map_2.map.gz | 494.1 MB 495.1 MB 494.5 MB 928.8 MB 928.8 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-18308 http://ftp.pdbj.org/pub/emdb/structures/EMD-18308 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-18308 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-18308 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_18308_validation.pdf.gz emd_18308_validation.pdf.gz | 1.3 MB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_18308_full_validation.pdf.gz emd_18308_full_validation.pdf.gz | 1.3 MB | 表示 | |
| XML形式データ |  emd_18308_validation.xml.gz emd_18308_validation.xml.gz | 22.1 KB | 表示 | |
| CIF形式データ |  emd_18308_validation.cif.gz emd_18308_validation.cif.gz | 26.2 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-18308 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-18308 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-18308 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-18308 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8qb9MC  8qb7C  8qb8C  8qbbC  8qbdC  8qbeC  8qbfC  8qbgC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_18308.map.gz / 形式: CCP4 / 大きさ: 1000 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_18308.map.gz / 形式: CCP4 / 大きさ: 1000 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Helical map Type II -PIP2/ sterol (D1, rise=5.04, twist=-136.50) | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.83 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_18308_msk_1.map emd_18308_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
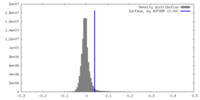
| 密度ヒストグラム |
-追加マップ: Helical map Type I -PIP2/ sterol (D2, rise=9.76, twist=131.91)
| ファイル | emd_18308_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Helical map Type I -PIP2/ sterol (D2, rise=9.76, twist=131.91) | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
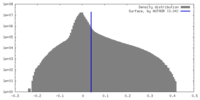
| 密度ヒストグラム |
-追加マップ: Helical map Type II -PIP2/ sterol - unsharpened map
| ファイル | emd_18308_additional_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Helical map Type II -PIP2/ sterol - unsharpened map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: Helical map Type III -PIP2/ sterol (D7, rise=36.54, twist=-14.47)
| ファイル | emd_18308_additional_3.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Helical map Type III -PIP2/ sterol (D7, rise=36.54, twist=-14.47) | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
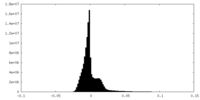
| 密度ヒストグラム |
-ハーフマップ: Helical map Type II -PIP2/ sterol - half map A
| ファイル | emd_18308_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Helical map Type II -PIP2/ sterol - half map A | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
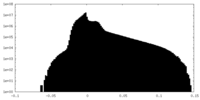
| 密度ヒストグラム |
-ハーフマップ: Helical map Type II -PIP2/ sterol - half map B
| ファイル | emd_18308_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Helical map Type II -PIP2/ sterol - half map B | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Helical assembly of recombinant Pil1 protein tubulating -PIP2/+st...
| 全体 | 名称: Helical assembly of recombinant Pil1 protein tubulating -PIP2/+sterol lipid mixture (DOPC,DOPE,DOPS,cholesterol 30:20:20:30) |
|---|---|
| 要素 |
|
-超分子 #1: Helical assembly of recombinant Pil1 protein tubulating -PIP2/+st...
| 超分子 | 名称: Helical assembly of recombinant Pil1 protein tubulating -PIP2/+sterol lipid mixture (DOPC,DOPE,DOPS,cholesterol 30:20:20:30) タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  |
-分子 #1: Sphingolipid long chain base-responsive protein PIL1
| 分子 | 名称: Sphingolipid long chain base-responsive protein PIL1 タイプ: protein_or_peptide / ID: 1 / コピー数: 14 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 38.393043 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MHRTYSLRNS RAPTASQLQN PPPPPSTTKG RFFGKGGLAY SFRRSAAGAF GPELSRKLSQ LVKIEKNVLR SMELTANERR DAAKQLSIW GLENDDDVSD ITDKLGVLIY EVSELDDQFI DRYDQYRLTL KSIRDIEGSV QPSRDRKDKI TDKIAYLKYK D PQSPKIEV ...文字列: MHRTYSLRNS RAPTASQLQN PPPPPSTTKG RFFGKGGLAY SFRRSAAGAF GPELSRKLSQ LVKIEKNVLR SMELTANERR DAAKQLSIW GLENDDDVSD ITDKLGVLIY EVSELDDQFI DRYDQYRLTL KSIRDIEGSV QPSRDRKDKI TDKIAYLKYK D PQSPKIEV LEQELVRAEA ESLVAEAQLS NITRSKLRAA FNYQFDSIIE HSEKIALIAG YGKALLELLD DSPVTPGETR PA YDGYEAS KQIIIDAESA LNEWTLDSAQ VKPTLSFKQD YEDFEPEEGE EEEEEDGQGR WSEDEQEDGQ IEEPEQEEEG AVE EHEQVG HQQSESLPQQ TTA UniProtKB: Sphingolipid long chain base-responsive protein PIL1 |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | らせん対称体再構成法 |
| 試料の集合状態 | helical array |
- 試料調製
試料調製
| 緩衝液 | pH: 7.4 / 詳細: 20mM HEPES, pH 7.4, 150mM KoAc, 2mM MgAc |
|---|---|
| グリッド | モデル: EMS Lacey Carbon / 材質: COPPER / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: LACEY |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 90 % / チャンバー内温度: 291 K / 装置: LEICA EM GP |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: FEI FALCON IV (4k x 4k) 平均電子線量: 50.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 1.8 µm / 最小 デフォーカス(公称値): 0.6 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 最終 再構成 | 想定した対称性 - らせんパラメータ - Δz: 5.044 Å 想定した対称性 - らせんパラメータ - ΔΦ: -136.5 ° 想定した対称性 - らせんパラメータ - 軸対称性: D1 (2回x1回 2面回転対称) 解像度のタイプ: BY AUTHOR / 解像度: 3.35 Å / 解像度の算出法: FSC 0.143 CUT-OFF / ソフトウェア - 名称: cryoSPARC (ver. 4.1.2) / 使用した粒子像数: 176005 |
|---|---|
| 初期モデル | モデルのタイプ: NONE |
| 最終 角度割当 | タイプ: NOT APPLICABLE / ソフトウェア - 名称: cryoSPARC (ver. 4.1.2) |
 ムービー
ムービー コントローラー
コントローラー











 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)