+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Native eisosome lattice bound to plasma membrane microdomain | |||||||||||||||
 マップデータ マップデータ | Native eisosome lattice - sharpened map | |||||||||||||||
 試料 試料 |
| |||||||||||||||
 キーワード キーワード | BAR domain / plasma membrane microdomain / membrane curvature / native biochemistry / LIPID BINDING PROTEIN | |||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報protein localization to eisosome filament / eisosome filament / eisosome assembly / eisosome / membrane organization / lipid droplet / cell periphery / endocytosis / intracellular protein localization / response to heat ...protein localization to eisosome filament / eisosome filament / eisosome assembly / eisosome / membrane organization / lipid droplet / cell periphery / endocytosis / intracellular protein localization / response to heat / mitochondrial outer membrane / lipid binding / mitochondrion / plasma membrane / cytoplasm 類似検索 - 分子機能 | |||||||||||||||
| 生物種 |  | |||||||||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.2 Å | |||||||||||||||
 データ登録者 データ登録者 | Kefauver JM / Zou L / Loewith RJ / Desfosses A | |||||||||||||||
| 資金援助 | European Union,  スイス, 4件 スイス, 4件
| |||||||||||||||
 引用 引用 | ジャーナル: Acta Crystallogr D Struct Biol / 年: 2018 タイトル: Real-space refinement in PHENIX for cryo-EM and crystallography. 著者: Pavel V Afonine / Billy K Poon / Randy J Read / Oleg V Sobolev / Thomas C Terwilliger / Alexandre Urzhumtsev / Paul D Adams /    要旨: This article describes the implementation of real-space refinement in the phenix.real_space_refine program from the PHENIX suite. The use of a simplified refinement target function enables very fast ...This article describes the implementation of real-space refinement in the phenix.real_space_refine program from the PHENIX suite. The use of a simplified refinement target function enables very fast calculation, which in turn makes it possible to identify optimal data-restraint weights as part of routine refinements with little runtime cost. Refinement of atomic models against low-resolution data benefits from the inclusion of as much additional information as is available. In addition to standard restraints on covalent geometry, phenix.real_space_refine makes use of extra information such as secondary-structure and rotamer-specific restraints, as well as restraints or constraints on internal molecular symmetry. The re-refinement of 385 cryo-EM-derived models available in the Protein Data Bank at resolutions of 6 Å or better shows significant improvement of the models and of the fit of these models to the target maps. | |||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_18307.map.gz emd_18307.map.gz | 59.7 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-18307-v30.xml emd-18307-v30.xml emd-18307.xml emd-18307.xml | 41 KB 41 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_18307.png emd_18307.png | 134.8 KB | ||
| Filedesc metadata |  emd-18307.cif.gz emd-18307.cif.gz | 6.7 KB | ||
| その他 |  emd_18307_additional_1.map.gz emd_18307_additional_1.map.gz emd_18307_additional_10.map.gz emd_18307_additional_10.map.gz emd_18307_additional_11.map.gz emd_18307_additional_11.map.gz emd_18307_additional_2.map.gz emd_18307_additional_2.map.gz emd_18307_additional_3.map.gz emd_18307_additional_3.map.gz emd_18307_additional_4.map.gz emd_18307_additional_4.map.gz emd_18307_additional_5.map.gz emd_18307_additional_5.map.gz emd_18307_additional_6.map.gz emd_18307_additional_6.map.gz emd_18307_additional_7.map.gz emd_18307_additional_7.map.gz emd_18307_additional_8.map.gz emd_18307_additional_8.map.gz emd_18307_additional_9.map.gz emd_18307_additional_9.map.gz emd_18307_half_map_1.map.gz emd_18307_half_map_1.map.gz emd_18307_half_map_2.map.gz emd_18307_half_map_2.map.gz | 31.8 MB 271.1 MB 59.8 MB 267.4 MB 272.9 MB 273.9 MB 274.1 MB 272.7 MB 271.2 MB 272 MB 266.6 MB 59.3 MB 59.3 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-18307 http://ftp.pdbj.org/pub/emdb/structures/EMD-18307 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-18307 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-18307 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_18307_validation.pdf.gz emd_18307_validation.pdf.gz | 864.3 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_18307_full_validation.pdf.gz emd_18307_full_validation.pdf.gz | 863.9 KB | 表示 | |
| XML形式データ |  emd_18307_validation.xml.gz emd_18307_validation.xml.gz | 12.2 KB | 表示 | |
| CIF形式データ |  emd_18307_validation.cif.gz emd_18307_validation.cif.gz | 14.4 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-18307 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-18307 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-18307 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-18307 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8qb7MC  8qb8MC  8qb9C  8qbbC  8qbdC  8qbeC  8qbfC  8qbgC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_18307.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_18307.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Native eisosome lattice - sharpened map | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.327 Å | ||||||||||||||||||||||||||||||||||||
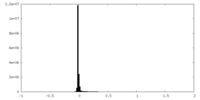
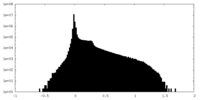
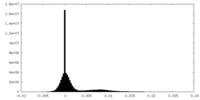
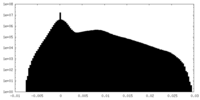
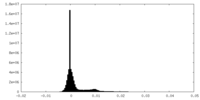
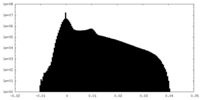
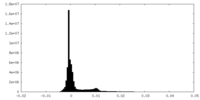
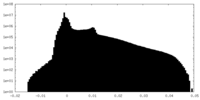
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
+追加マップ: Native eisosome lattice - unsharpened map
+追加マップ: Native eisosome helical map (C2, rise=11.00, twist=54.02)
+追加マップ: Native eisosome lattice - deepEMhancer sharpened map
+追加マップ: Native eisosome helical map (C4, rise=21.27, twist=50.99)
+追加マップ: Native eisosome helical map (C7, rise=37.28, twist=36.72)
+追加マップ: Native eisosome helical map (C1, rise=5.58, twist=53.51)
+追加マップ: Native eisosome helical map (C1, rise=5.13, twist=49.32)
+追加マップ: Native eisosome helical map (C1, rise=5.2, twist=136.56)
+追加マップ: Native eisosome helical map (C2, rise=10.02, twist=47.98)
+追加マップ: Native eisosome helical map (C1, rise=4.89, twist=46.59)
+追加マップ: Native eisosome helical map (C8, rise=37.41, twist=57.83)
+ハーフマップ: Native eisosome lattice - half map B
+ハーフマップ: Native eisosome lattice - half map A
- 試料の構成要素
試料の構成要素
-全体 : Helical lattice of native Pil1/Lsp1 protein bound to plasma membr...
| 全体 | 名称: Helical lattice of native Pil1/Lsp1 protein bound to plasma membrane microdomain |
|---|---|
| 要素 |
|
-超分子 #1: Helical lattice of native Pil1/Lsp1 protein bound to plasma membr...
| 超分子 | 名称: Helical lattice of native Pil1/Lsp1 protein bound to plasma membrane microdomain タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  |
-分子 #1: Sphingolipid long chain base-responsive protein PIL1
| 分子 | 名称: Sphingolipid long chain base-responsive protein PIL1 タイプ: protein_or_peptide / ID: 1 / コピー数: 14 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 38.393043 KDa |
| 配列 | 文字列: MHRTYSLRNS RAPTASQLQN PPPPPSTTKG RFFGKGGLAY SFRRSAAGAF GPELSRKLSQ LVKIEKNVLR SMELTANERR DAAKQLSIW GLENDDDVSD ITDKLGVLIY EVSELDDQFI DRYDQYRLTL KSIRDIEGSV QPSRDRKDKI TDKIAYLKYK D PQSPKIEV ...文字列: MHRTYSLRNS RAPTASQLQN PPPPPSTTKG RFFGKGGLAY SFRRSAAGAF GPELSRKLSQ LVKIEKNVLR SMELTANERR DAAKQLSIW GLENDDDVSD ITDKLGVLIY EVSELDDQFI DRYDQYRLTL KSIRDIEGSV QPSRDRKDKI TDKIAYLKYK D PQSPKIEV LEQELVRAEA ESLVAEAQLS NITRSKLRAA FNYQFDSIIE HSEKIALIAG YGKALLELLD DSPVTPGETR PA YDGYEAS KQIIIDAESA LNEWTLDSAQ VKPTLSFKQD YEDFEPEEGE EEEEEDGQGR WSEDEQEDGQ IEEPEQEEEG AVE EHEQVG HQQSESLPQQ TTA UniProtKB: Sphingolipid long chain base-responsive protein PIL1 |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | helical array |
- 試料調製
試料調製
| 緩衝液 | pH: 7 / 詳細: 50mM PIPES pH 7, 300mM NaCl, 1mM CHAPS, 0.5mM DTT |
|---|---|
| グリッド | モデル: EMS Lacey Carbon / 材質: COPPER / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: LACEY |
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 QUANTUM (4k x 4k) 平均電子線量: 40.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 1.8 µm / 最小 デフォーカス(公称値): 0.8 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期モデル | モデルのタイプ: OTHER / 詳細: Cylindrical map |
|---|---|
| 最終 再構成 | 解像度のタイプ: BY AUTHOR / 解像度: 3.2 Å / 解像度の算出法: FSC 0.143 CUT-OFF / ソフトウェア - 名称: cryoSPARC (ver. 4.1.2) / 使用した粒子像数: 2423944 |
| 初期 角度割当 | タイプ: MAXIMUM LIKELIHOOD / ソフトウェア - 名称: RELION (ver. 3.0.8) |
| 最終 角度割当 | タイプ: MAXIMUM LIKELIHOOD / ソフトウェア - 名称: cryoSPARC (ver. 4.1.2) |
 ムービー
ムービー コントローラー
コントローラー











 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)