+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
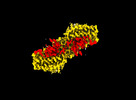
| タイトル | MTHFR + SAH symmetric dis-inhibited state | |||||||||
 マップデータ マップデータ | Main Sharpened | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Dis-inhibited / allosteric / folate / S-adenosylhomocysteine / FLAVOPROTEIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報methylenetetrahydrofolate reductase (NADPH) / response to vitamin B2 / methylenetetrahydrofolate reductase (NADPH) activity / methylenetetrahydrofolate reductase [NAD(P)H] activity / methionine metabolic process / modified amino acid binding / heterochromatin organization / homocysteine metabolic process / S-adenosylmethionine metabolic process / response to folic acid ...methylenetetrahydrofolate reductase (NADPH) / response to vitamin B2 / methylenetetrahydrofolate reductase (NADPH) activity / methylenetetrahydrofolate reductase [NAD(P)H] activity / methionine metabolic process / modified amino acid binding / heterochromatin organization / homocysteine metabolic process / S-adenosylmethionine metabolic process / response to folic acid / methionine biosynthetic process / Metabolism of folate and pterines / tetrahydrofolate interconversion / response to amino acid / FAD binding / response to interleukin-1 / neural tube closure / flavin adenine dinucleotide binding / NADP binding / response to hypoxia / response to xenobiotic stimulus / protein-containing complex binding / cytosol 類似検索 - 分子機能 | |||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
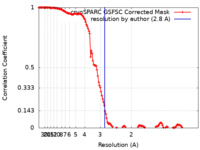
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.8 Å | |||||||||
 データ登録者 データ登録者 | Blomgren LKM / Yue WW / Froese DS / McCorvie TJ | |||||||||
| 資金援助 |  スイス, 1件 スイス, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2024 ジャーナル: Nat Commun / 年: 2024タイトル: Dynamic inter-domain transformations mediate the allosteric regulation of human 5, 10-methylenetetrahydrofolate reductase. 著者: Linnea K M Blomgren / Melanie Huber / Sabrina R Mackinnon / Céline Bürer / Arnaud Baslé / Wyatt W Yue / D Sean Froese / Thomas J McCorvie /   要旨: 5,10-methylenetetrahydrofolate reductase (MTHFR) commits folate-derived one-carbon units to generate the methyl-donor S-adenosyl-L-methionine (SAM). Eukaryotic MTHFR appends to the well-conserved ...5,10-methylenetetrahydrofolate reductase (MTHFR) commits folate-derived one-carbon units to generate the methyl-donor S-adenosyl-L-methionine (SAM). Eukaryotic MTHFR appends to the well-conserved catalytic domain (CD) a unique regulatory domain (RD) that confers feedback inhibition by SAM. Here we determine the cryo-electron microscopy structures of human MTHFR bound to SAM and its demethylated product S-adenosyl-L-homocysteine (SAH). In the active state, with the RD bound to a single SAH, the CD is flexible and exposes its active site for catalysis. However, in the inhibited state the RD pocket is remodelled, exposing a second SAM-binding site that was previously occluded. Dual-SAM bound MTHFR demonstrates a substantially rearranged inter-domain linker that reorients the CD, inserts a loop into the active site, positions Tyr404 to bind the cofactor FAD, and blocks substrate access. Our data therefore explain the long-distance regulatory mechanism of MTHFR inhibition, underpinned by the transition between dual-SAM and single-SAH binding in response to cellular methylation status. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_18298.map.gz emd_18298.map.gz | 229.7 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-18298-v30.xml emd-18298-v30.xml emd-18298.xml emd-18298.xml | 20.7 KB 20.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_18298_fsc.xml emd_18298_fsc.xml | 13.3 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_18298.png emd_18298.png | 35.3 KB | ||
| マスクデータ |  emd_18298_msk_1.map emd_18298_msk_1.map | 244.1 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-18298.cif.gz emd-18298.cif.gz | 6.6 KB | ||
| その他 |  emd_18298_additional_1.map.gz emd_18298_additional_1.map.gz emd_18298_half_map_1.map.gz emd_18298_half_map_1.map.gz emd_18298_half_map_2.map.gz emd_18298_half_map_2.map.gz | 120.9 MB 226.2 MB 226.2 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-18298 http://ftp.pdbj.org/pub/emdb/structures/EMD-18298 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-18298 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-18298 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_18298_validation.pdf.gz emd_18298_validation.pdf.gz | 1.2 MB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_18298_full_validation.pdf.gz emd_18298_full_validation.pdf.gz | 1.2 MB | 表示 | |
| XML形式データ |  emd_18298_validation.xml.gz emd_18298_validation.xml.gz | 22.2 KB | 表示 | |
| CIF形式データ |  emd_18298_validation.cif.gz emd_18298_validation.cif.gz | 28.7 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-18298 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-18298 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-18298 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-18298 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_18298.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_18298.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Main Sharpened | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.574 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_18298_msk_1.map emd_18298_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: Non-sharpened
| ファイル | emd_18298_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Non-sharpened | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_18298_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_18298_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Human 5,10-methylenetetrahydrofolate reductase in complex with S-...
| 全体 | 名称: Human 5,10-methylenetetrahydrofolate reductase in complex with S-Adenosyl-L-homocysteine, regulatory domains |
|---|---|
| 要素 |
|
-超分子 #1: Human 5,10-methylenetetrahydrofolate reductase in complex with S-...
| 超分子 | 名称: Human 5,10-methylenetetrahydrofolate reductase in complex with S-Adenosyl-L-homocysteine, regulatory domains タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 150 KDa |
-分子 #1: Methylenetetrahydrofolate reductase (NADPH)
| 分子 | 名称: Methylenetetrahydrofolate reductase (NADPH) / タイプ: protein_or_peptide / ID: 1 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 75.461195 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MVNEARGNSS LNPCLEGSAS SGSESSKDSS RCSTPGLDPE RHERLREKMR RRLESGDKWF SLEFFPPRTA EGAVNLISRF DRMAAGGPL YIDVTWHPAG DPGSDKETSS MMIASTAVNY CGLETILHMT CCRQRLEEIT GHLHKAKQLG LKNIMALRGD P IGDQWEEE ...文字列: MVNEARGNSS LNPCLEGSAS SGSESSKDSS RCSTPGLDPE RHERLREKMR RRLESGDKWF SLEFFPPRTA EGAVNLISRF DRMAAGGPL YIDVTWHPAG DPGSDKETSS MMIASTAVNY CGLETILHMT CCRQRLEEIT GHLHKAKQLG LKNIMALRGD P IGDQWEEE EGGFNYAVDL VKHIRSEFGD YFDICVAGYP KGHPEAGSFE ADLKHLKEKV SAGADFIITQ LFFEADTFFR FV KACTDMG ITCPIVPGIF PIQGYHSLRQ LVKLSKLEVP QEIKDVIEPI KDNDAAIRNY GIELAVSLCQ ELLASGLVPG LHF YTLNRE MATTEVLKRL GMWTEDPRRP LPWALSAHPK RREEDVRPIF WASRPKSYIY RTQEWDEFPN GRWGNSSSPA FGEL KDYYL FYLKSKSPKE ELLKMWGEEL TSEASVFEVF VLYLSGEPNR NGHKVTCLPW NDEPLAAETS LLKEELLRVN RQGIL TINS QPNINGKPSS DPIVGWGPSG GYVFQKAYLE FFTSRETAEA LLQVLKKYEL RVNYHLVNVK GENITNAPEL QPNAVT WGI FPGREIIQPT VVDPVSFMFW KDEAFALWIE QWGKLYEEES PSRTIIQYIH DNYFLVNLVD NDFPLDNCLW QVVEDTL EL LNRPTQNARE TEAPAENLYF Q UniProtKB: Methylenetetrahydrofolate reductase (NADPH) |
-分子 #2: S-ADENOSYL-L-HOMOCYSTEINE
| 分子 | 名称: S-ADENOSYL-L-HOMOCYSTEINE / タイプ: ligand / ID: 2 / コピー数: 2 / 式: SAH |
|---|---|
| 分子量 | 理論値: 384.411 Da |
| Chemical component information |  ChemComp-SAH: |
-分子 #3: water
| 分子 | 名称: water / タイプ: ligand / ID: 3 / コピー数: 28 / 式: HOH |
|---|---|
| 分子量 | 理論値: 18.015 Da |
| Chemical component information |  ChemComp-HOH: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 2.0 mg/mL |
|---|---|
| 緩衝液 | pH: 7.5 詳細: 20 mM HEPES, pH 7.5, 150 mM NaCl, 0.0025% Tween20, 1 mM S-Adenosyl-L-homocysteine, filter sterilised |
| グリッド | モデル: UltrAuFoil R1.2/1.3 / 材質: GOLD / メッシュ: 300 / 支持フィルム - 材質: GOLD / 支持フィルム - トポロジー: HOLEY ARRAY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 40 sec. |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 295 K / 装置: FEI VITROBOT MARK III |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | TFS GLACIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: FEI FALCON IV (4k x 4k) 撮影したグリッド数: 2 / 実像数: 5606 / 平均露光時間: 5.18 sec. / 平均電子線量: 50.0 e/Å2 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 100.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.1 µm / 最小 デフォーカス(公称値): 0.9 µm / 倍率(公称値): 240000 |
| 試料ステージ | ホルダー冷却材: NITROGEN |
 ムービー
ムービー コントローラー
コントローラー









 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)