+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Composite map of the Gallus gallus 80S rotated ribosome in complex with eEF2 and SERBP1 | |||||||||
 マップデータ マップデータ | Composite map of the Gallus gallus 80S rotated ribosome in complex with eEF2 and SERBP1 | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | translation / complex / eEF2 / SERBP1 / RIBOSOME | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報TNFR1-mediated ceramide production / Major pathway of rRNA processing in the nucleolus and cytosol / Protein hydroxylation / glucose 6-phosphate:phosphate antiporter activity / Translesion synthesis by REV1 / Recognition of DNA damage by PCNA-containing replication complex / Translesion Synthesis by POLH / Activation of NF-kappaB in B cells / Oxygen-dependent proline hydroxylation of Hypoxia-inducible Factor Alpha / Spry regulation of FGF signaling ...TNFR1-mediated ceramide production / Major pathway of rRNA processing in the nucleolus and cytosol / Protein hydroxylation / glucose 6-phosphate:phosphate antiporter activity / Translesion synthesis by REV1 / Recognition of DNA damage by PCNA-containing replication complex / Translesion Synthesis by POLH / Activation of NF-kappaB in B cells / Oxygen-dependent proline hydroxylation of Hypoxia-inducible Factor Alpha / Spry regulation of FGF signaling / Downregulation of ERBB2:ERBB3 signaling / APC/C:Cdc20 mediated degradation of Cyclin B / Autodegradation of Cdh1 by Cdh1:APC/C / SCF-beta-TrCP mediated degradation of Emi1 / APC/C:Cdc20 mediated degradation of Securin / APC/C:Cdh1 mediated degradation of Cdc20 and other APC/C:Cdh1 targeted proteins in late mitosis/early G1 / Cdc20:Phospho-APC/C mediated degradation of Cyclin A / APC-Cdc20 mediated degradation of Nek2A / EGFR downregulation / SCF(Skp2)-mediated degradation of p27/p21 / Degradation of beta-catenin by the destruction complex / TCF dependent signaling in response to WNT / Downstream TCR signaling / NRIF signals cell death from the nucleus / p75NTR recruits signalling complexes / NF-kB is activated and signals survival / Activated NOTCH1 Transmits Signal to the Nucleus / Downregulation of SMAD2/3:SMAD4 transcriptional activity / SMAD2/SMAD3:SMAD4 heterotrimer regulates transcription / Senescence-Associated Secretory Phenotype (SASP) / FCERI mediated NF-kB activation / Regulation of innate immune responses to cytosolic DNA / Autodegradation of the E3 ubiquitin ligase COP1 / RAD18 and ubiquitinated PCNA-mediated recruitment of translesion polymerases / Nucleotide Excision Repair / Deactivation of the beta-catenin transactivating complex / TRAF6 mediated induction of proinflammatory cytokines / TAK1 activates NFkB by phosphorylation and activation of IKKs complex / NFkB activation mediated by RIP1 complexed with activated TLR3 / Activated TAK1 mediates p38 MAP kinase phosphorylation / Activated TAK1 mediates Jun kinases (JNK) phosphorylation and activation / activated TAK1 mediates p38 MAPK activation / JNK (c-Jun kinases) phosphorylation and activation mediated by activated human TAK1 / AUF1 (hnRNP D0) binds and destabilizes mRNA / Degradation of AXIN / Degradation of DVL / Regulation of FZD by ubiquitination / N-glycan trimming in the ER and Calnexin/Calreticulin cycle / Regulation of TNFR1 signaling / TNFR1-induced NF-kappa-B signaling pathway / Hedgehog ligand biogenesis / CLEC7A (Dectin-1) signaling / Degradation of GLI1 by the proteasome / GLI3 is processed to GLI3R by the proteasome / Hedgehog 'on' state / Negative regulation of FGFR1 signaling / Negative regulation of FGFR2 signaling / Negative regulation of FGFR3 signaling / Negative regulation of FGFR4 signaling / Translesion synthesis by POLK / Translesion synthesis by POLI / Termination of translesion DNA synthesis / TNFR2 non-canonical NF-kB pathway / Negative regulation of MAPK pathway / Regulation of necroptotic cell death / MAP3K8 (TPL2)-dependent MAPK1/3 activation / HDR through Homologous Recombination (HRR) / MAPK6/MAPK4 signaling / Josephin domain DUBs / Ovarian tumor domain proteases / Formation of Incision Complex in GG-NER / Gap-filling DNA repair synthesis and ligation in GG-NER / Dual Incision in GG-NER / NFkB and MAPK activation mediated by TRAF6 upon TLR7 or TLR21 stimulation / Formation of TC-NER Pre-Incision Complex / Dual incision in TC-NER / Gap-filling DNA repair synthesis and ligation in TC-NER / Regulation of TP53 Activity through Phosphorylation / Regulation of TP53 Degradation / Regulation of TP53 Activity through Methylation / Negative regulation of MET activity / Assembly of the pre-replicative complex / CDK-mediated phosphorylation and removal of Cdc6 / Ubiquitin-Mediated Degradation of Phosphorylated Cdc25A / Translation initiation complex formation / Formation of the ternary complex, and subsequently, the 43S complex / Ribosomal scanning and start codon recognition / Ubiquitin-dependent degradation of Cyclin D / PTK6 Regulates RTKs and Their Effectors AKT1 and DOK1 / FBXL7 down-regulates AURKA during mitotic entry and in early mitosis / Downregulation of ERBB2 signaling / VLDLR internalisation and degradation / Synthesis of active ubiquitin: roles of E1 and E2 enzymes / E3 ubiquitin ligases ubiquitinate target proteins / RUNX1 regulates transcription of genes involved in differentiation of HSCs / Regulation of RUNX2 expression and activity / Regulation of PTEN localization / Regulation of PTEN stability and activity / Neddylation / ER Quality Control Compartment (ERQC) 類似検索 - 分子機能 | |||||||||
| 生物種 |  | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.4 Å | |||||||||
 データ登録者 データ登録者 | Nurullina L / Jenner L / Myasnikov A / Yusupov M | |||||||||
| 資金援助 |  フランス, 1件 フランス, 1件
| |||||||||
 引用 引用 |  ジャーナル: FEBS Lett / 年: 2024 ジャーナル: FEBS Lett / 年: 2024タイトル: Cryo-EM structure of the inactive ribosome complex accumulated in chick embryo cells in cold-stress conditions. 著者: Liliia Nurullina / Salvatore Terrosu / Alexander G Myasnikov / Lasse Bohl Jenner / Marat Yusupov /    要旨: Here, we present the high-resolution structure of the Gallus gallus 80S ribosome obtained from cold-treated chicken embryos. The translationally inactive ribosome complex contains elongation factor ...Here, we present the high-resolution structure of the Gallus gallus 80S ribosome obtained from cold-treated chicken embryos. The translationally inactive ribosome complex contains elongation factor eEF2 with GDP, SERPINE1 mRNA binding protein 1 (SERBP1) and deacylated tRNA in the P/E position, showing common features with complexes already described in mammals. Modeling of most expansion segments of G. gallus 28S ribosomal RNA allowed us to identify specific features in their structural organization and to describe areas where a marked difference between mammalian and avian ribosomes could shed light on the evolution of the expansion segments. This study provides the first structure of an avian ribosome, establishing a model for future structural and functional studies on the translational machinery in Aves. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_18169.map.gz emd_18169.map.gz | 674.7 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-18169-v30.xml emd-18169-v30.xml emd-18169.xml emd-18169.xml | 13.6 KB 13.6 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_18169.png emd_18169.png | 51.9 KB | ||
| Filedesc metadata |  emd-18169.cif.gz emd-18169.cif.gz | 4.6 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-18169 http://ftp.pdbj.org/pub/emdb/structures/EMD-18169 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-18169 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-18169 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_18169_validation.pdf.gz emd_18169_validation.pdf.gz | 623.3 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_18169_full_validation.pdf.gz emd_18169_full_validation.pdf.gz | 623 KB | 表示 | |
| XML形式データ |  emd_18169_validation.xml.gz emd_18169_validation.xml.gz | 8.8 KB | 表示 | |
| CIF形式データ |  emd_18169_validation.cif.gz emd_18169_validation.cif.gz | 10.2 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-18169 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-18169 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-18169 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-18169 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8q87MC  8q7zC C: 同じ文献を引用 ( M: このマップから作成された原子モデル |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_18169.map.gz / 形式: CCP4 / 大きさ: 744.3 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_18169.map.gz / 形式: CCP4 / 大きさ: 744.3 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Composite map of the Gallus gallus 80S rotated ribosome in complex with eEF2 and SERBP1 | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.803 Å | ||||||||||||||||||||||||||||||||||||
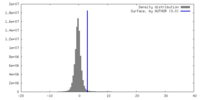
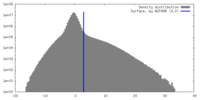
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : 80S Gallus gallus ribosome from chicken embryo
| 全体 | 名称: 80S Gallus gallus ribosome from chicken embryo |
|---|---|
| 要素 |
|
-超分子 #1: 80S Gallus gallus ribosome from chicken embryo
| 超分子 | 名称: 80S Gallus gallus ribosome from chicken embryo / タイプ: complex / ID: 1 / 親要素: 0 詳細: 28S rRNA, 5.8S rRNA, 5S rRNA, 18S rRNA,ribosomal proteins, tRNA, eEF2, SERBP1 |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 4.3 MDa |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 |
|---|---|
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 95 % / チャンバー内温度: 278 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 特殊光学系 | エネルギーフィルター - 名称: TFS Selectris X |
| 撮影 | フィルム・検出器のモデル: FEI FALCON IV (4k x 4k) 撮影したグリッド数: 1 / 実像数: 32439 / 平均電子線量: 40.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 1.5 µm / 最小 デフォーカス(公称値): 0.3 µm / 倍率(公称値): 96000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー




























 X (Sec.)
X (Sec.) Y (Row.)
Y (Row.) Z (Col.)
Z (Col.)