+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of an asymmetric nucleosome (10-N-0) from a Glacios/Falcon3EC | |||||||||
 マップデータ マップデータ | Map from homogeneous refinement in cryosparc. | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Nucleosome / NCP / DNA / Histones / DNA BINDING PROTEIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報structural constituent of chromatin / nucleosome / heterochromatin formation / nucleosome assembly / protein heterodimerization activity / DNA binding / nucleoplasm / nucleus 類似検索 - 分子機能 | |||||||||
| 生物種 | ||||||||||
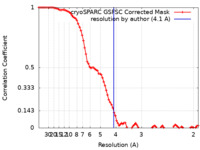
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 4.1 Å | |||||||||
 データ登録者 データ登録者 | Gaullier G | |||||||||
| 資金援助 | 1件
| |||||||||
 引用 引用 |  ジャーナル: Elife / 年: 2021 ジャーナル: Elife / 年: 2021タイトル: Structure and dynamics of the chromatin remodeler ALC1 bound to a PARylated nucleosome. 著者: Luka Bacic / Guillaume Gaullier / Anton Sabantsev / Laura C Lehmann / Klaus Brackmann / Despoina Dimakou / Mario Halic / Graeme Hewitt / Simon J Boulton / Sebastian Deindl /    要旨: The chromatin remodeler ALC1 is recruited to and activated by DNA damage-induced poly(ADP-ribose) (PAR) chains deposited by PARP1/PARP2/HPF1 upon detection of DNA lesions. ALC1 has emerged as a ...The chromatin remodeler ALC1 is recruited to and activated by DNA damage-induced poly(ADP-ribose) (PAR) chains deposited by PARP1/PARP2/HPF1 upon detection of DNA lesions. ALC1 has emerged as a candidate drug target for cancer therapy as its loss confers synthetic lethality in homologous recombination-deficient cells. However, structure-based drug design and molecular analysis of ALC1 have been hindered by the requirement for PARylation and the highly heterogeneous nature of this post-translational modification. Here, we reconstituted an ALC1 and PARylated nucleosome complex modified in vitro using PARP2 and HPF1. This complex was amenable to cryo-EM structure determination without cross-linking, which enabled visualization of several intermediate states of ALC1 from the recognition of the PARylated nucleosome to the tight binding and activation of the remodeler. Functional biochemical assays with PARylated nucleosomes highlight the importance of nucleosomal epitopes for productive remodeling and suggest that ALC1 preferentially slides nucleosomes away from DNA breaks. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_17944.map.gz emd_17944.map.gz | 31.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-17944-v30.xml emd-17944-v30.xml emd-17944.xml emd-17944.xml | 24.6 KB 24.6 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_17944_fsc.xml emd_17944_fsc.xml | 8.5 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_17944.png emd_17944.png | 36.3 KB | ||
| マスクデータ |  emd_17944_msk_1.map emd_17944_msk_1.map emd_17944_msk_2.map emd_17944_msk_2.map | 64 MB 64 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-17944.cif.gz emd-17944.cif.gz | 6.3 KB | ||
| その他 |  emd_17944_additional_1.map.gz emd_17944_additional_1.map.gz emd_17944_half_map_1.map.gz emd_17944_half_map_1.map.gz emd_17944_half_map_2.map.gz emd_17944_half_map_2.map.gz | 57.5 MB 59.5 MB 59.5 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-17944 http://ftp.pdbj.org/pub/emdb/structures/EMD-17944 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-17944 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-17944 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_17944_validation.pdf.gz emd_17944_validation.pdf.gz | 1.1 MB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_17944_full_validation.pdf.gz emd_17944_full_validation.pdf.gz | 1.1 MB | 表示 | |
| XML形式データ |  emd_17944_validation.xml.gz emd_17944_validation.xml.gz | 16.3 KB | 表示 | |
| CIF形式データ |  emd_17944_validation.cif.gz emd_17944_validation.cif.gz | 21.2 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-17944 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-17944 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-17944 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-17944 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  7otqC C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_17944.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_17944.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Map from homogeneous refinement in cryosparc. | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.952 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_17944_msk_1.map emd_17944_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-マスク #2
| ファイル |  emd_17944_msk_2.map emd_17944_msk_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: Map after post-processing with deepEMhancer, using the "tighTarget"...
| ファイル | emd_17944_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Map after post-processing with deepEMhancer, using the "tighTarget" weights. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half-map A from homogeneous refinement in cryosparc.
| ファイル | emd_17944_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half-map A from homogeneous refinement in cryosparc. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half-map B from homogeneous refinement in cryosparc.
| ファイル | emd_17944_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half-map B from homogeneous refinement in cryosparc. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : 601 nucleosome with 10 additional bp
| 全体 | 名称: 601 nucleosome with 10 additional bp |
|---|---|
| 要素 |
|
-超分子 #1: 601 nucleosome with 10 additional bp
| 超分子 | 名称: 601 nucleosome with 10 additional bp / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all 詳細: The histones were recombinantly expressed and purified. The DNA was prepared by PCR. The nucleosomes were assembled by salt gradient dialysis. |
|---|---|
| 分子量 | 理論値: 205.8 KDa |
-分子 #1: Histone H3.2
| 分子 | 名称: Histone H3.2 / タイプ: protein_or_peptide / ID: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種: |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MARTKQTARK STGGKAPRKQ LATKAARKSA PATGGVKKPH RYRPGTVALR EIRRYQKSTE LLIRKLPFQR LVREIAQDFK TDLRFQSSAV MALQEASEAY LVALFEDTNL AAIHAKRVTI MPKDIQLARR IRGERA UniProtKB: Histone H3.2 |
-分子 #2: Histone H4
| 分子 | 名称: Histone H4 / タイプ: protein_or_peptide / ID: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種: |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MSGRGKGGKG LGKGGAKRHR KVLRDNIQGI TKPAIRRLAR RGGVKRISGL IYEETRGVLK VFLENVIRDA VTYTEHAKRK TVTAMDVVYA LKRQGRTLYG FGG UniProtKB: Histone H4 |
-分子 #3: Histone H2A type 1
| 分子 | 名称: Histone H2A type 1 / タイプ: protein_or_peptide / ID: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種: |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MSGRGKQGGK TRAKAKTRSS RAGLQFPVGR VHRLLRKGNY AERVGAGAPV YLAAVLEYLT AEILELAGNA ARDNKKTRII PRHLQLAVRN DEELNKLLGR VTIAQGGVLP NIQSVLLPKK TESSKSAKSK UniProtKB: Histone H2A type 1 |
-分子 #4: Histone H2B 1.1
| 分子 | 名称: Histone H2B 1.1 / タイプ: protein_or_peptide / ID: 4 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種: |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MAKSAPAPKK GSKKAVTKTQ KKDGKKRRKT RKESYAIYVY KVLKQVHPDT GISSKAMSIM NSFVNDVFER IAGEASRLAH YNKRSTITSR EIQTAVRLLL PGELAKHAVS EGTKAVTKYT SAK UniProtKB: Histone H2B 1.1 |
-分子 #5: Widom 601 sequence with 10 additional bp on the strong side
| 分子 | 名称: Widom 601 sequence with 10 additional bp on the strong side タイプ: dna / ID: 5 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 配列 | 文字列: AATCGATGTA TATATCTGAC ACGTGCCTGG AGACTAGGGA GTAATCCCCT TGGCGGTTAA AACGCGGGGG ACAGCGCGTA CGTGCGTTTA AGCGGTGCTA GAGCTGTCTA CGACCAATTG AGCGGCCTCG GCACCGGGAT TCTGATGGTC ACCTAGA |
-分子 #6: Widom 601 sequence with 10 additional bp on the strong side
| 分子 | 名称: Widom 601 sequence with 10 additional bp on the strong side タイプ: dna / ID: 6 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 配列 | 文字列: TCTAGGTGAC CATCAGAATC CCGGTGCCGA GGCCGCTCAA TTGGTCGTAG ACAGCTCTAG CACCGCTTAA ACGCACGTAC GCGCTGTCCC CCGCGTTTTA ACCGCCAAGG GGATTACTCC CTAGTCTCCA GGCACGTGTC AGATATATAC ATCGATT |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 1.17 mg/mL | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 8 / 構成要素:
| |||||||||
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY ARRAY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 120 sec. / 前処理 - 雰囲気: AIR / 前処理 - 気圧: 0.04 kPa / 詳細: Pelco easiGlow operated with a 30 mA current. | |||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV 詳細: Blot time 4s Blot force 0 4 uL of sample were applied on the grid (2 uL on each side of the grid). | |||||||||
| 詳細 | At the time of vitrification, the nucleosome sample used had spent about one year stored at 4C (this increases the propensity of nucleosomes to unwrap upon vitrification). |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | TFS GLACIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: FEI FALCON III (4k x 4k) 検出モード: COUNTING / 撮影したグリッド数: 1 / 実像数: 1537 / 平均電子線量: 40.01 e/Å2 / 詳細: 40 frames per movie |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 0.5 µm |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
 ムービー
ムービー コントローラー
コントローラー








 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)