+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Tilapia Lake Virus polymerase in cRNA pre-initiation state mode A (core only) | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Viral polymerase / VIRAL PROTEIN | |||||||||
| 機能・相同性 | RNA-directed RNA polymerase activity / DNA/RNA polymerase superfamily / Uncharacterized protein / Putative PB1 / RNA-dependent RNA polymerase 機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |  Tilapia lake virus (ウイルス) Tilapia lake virus (ウイルス) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.65 Å | |||||||||
 データ登録者 データ登録者 | Arragain B / Cusack S | |||||||||
| 資金援助 |  フランス, 1件 フランス, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2023 ジャーナル: Nat Commun / 年: 2023タイトル: Structural and functional analysis of the minimal orthomyxovirus-like polymerase of Tilapia Lake Virus from the highly diverged Amnoonviridae family. 著者: Benoit Arragain / Martin Pelosse / Albert Thompson / Stephen Cusack /   要旨: Tilapia Lake Virus (TiLV), a recently discovered pathogen of tilapia fish, belongs to the Amnoonviridae family from the Articulavirales order. Its ten genome segments have characteristic conserved ...Tilapia Lake Virus (TiLV), a recently discovered pathogen of tilapia fish, belongs to the Amnoonviridae family from the Articulavirales order. Its ten genome segments have characteristic conserved ends and encode proteins with no known homologues, apart from the segment 1, which encodes an orthomyxo-like RNA-dependent-RNA polymerase core subunit. Here we show that segments 1-3 encode respectively the PB1, PB2 and PA-like subunits of an active heterotrimeric polymerase that maintains all domains found in the distantly related influenza polymerase, despite an unprecedented overall size reduction of 40%. Multiple high-resolution cryo-EM structures of TiLV polymerase in pre-initiation, initiation and active elongation states, show how it binds the vRNA and cRNA promoters and performs RNA synthesis, with both transcriptase and replicase configurations being characterised. However, the highly truncated endonuclease-like domain appears inactive and the putative cap-binding domain is autoinhibited, emphasising that many functional aspects of TiLV polymerase remain to be elucidated. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_17860.map.gz emd_17860.map.gz | 7.6 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-17860-v30.xml emd-17860-v30.xml emd-17860.xml emd-17860.xml | 20.8 KB 20.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_17860_fsc.xml emd_17860_fsc.xml | 10.7 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_17860.png emd_17860.png | 59.7 KB | ||
| マスクデータ |  emd_17860_msk_1.map emd_17860_msk_1.map | 103 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-17860.cif.gz emd-17860.cif.gz | 6.7 KB | ||
| その他 |  emd_17860_additional_1.map.gz emd_17860_additional_1.map.gz emd_17860_half_map_1.map.gz emd_17860_half_map_1.map.gz emd_17860_half_map_2.map.gz emd_17860_half_map_2.map.gz | 80.8 MB 81 MB 80.8 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-17860 http://ftp.pdbj.org/pub/emdb/structures/EMD-17860 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-17860 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-17860 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_17860_validation.pdf.gz emd_17860_validation.pdf.gz | 832.5 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_17860_full_validation.pdf.gz emd_17860_full_validation.pdf.gz | 832 KB | 表示 | |
| XML形式データ |  emd_17860_validation.xml.gz emd_17860_validation.xml.gz | 17.6 KB | 表示 | |
| CIF形式データ |  emd_17860_validation.cif.gz emd_17860_validation.cif.gz | 23 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-17860 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-17860 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-17860 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-17860 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8psqMC  8psnC  8psoC  8pssC  8psuC  8psxC  8pszC  8pt2C  8pt6C  8pt7C  8pthC  8ptjC  8qz8C M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_17860.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_17860.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.84 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_17860_msk_1.map emd_17860_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
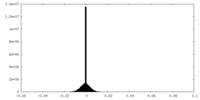
| 密度ヒストグラム |
-追加マップ: #1
| ファイル | emd_17860_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
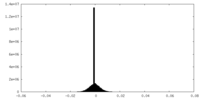
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_17860_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_17860_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Tilapia lake virus polymerase
| 全体 | 名称: Tilapia lake virus polymerase |
|---|---|
| 要素 |
|
-超分子 #1: Tilapia lake virus polymerase
| 超分子 | 名称: Tilapia lake virus polymerase / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#4 |
|---|---|
| 由来(天然) | 生物種:  Tilapia lake virus (ウイルス) Tilapia lake virus (ウイルス) |
-分子 #1: Polymerase acidic protein (PA-like)
| 分子 | 名称: Polymerase acidic protein (PA-like) / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Tilapia lake virus (ウイルス) Tilapia lake virus (ウイルス) |
| 分子量 | 理論値: 47.780508 KDa |
| 組換発現 | 生物種:  Trichoplusia ni (イラクサキンウワバ) Trichoplusia ni (イラクサキンウワバ) |
| 配列 | 文字列: MDSRFAQLTG VFCDDFTYSE GSRRFLSSYS TVERRPGVPV EGDCYDCLKN KWIAFELEGQ PRKFPKATVR CILNNDATYV CSEQEYQQI CKVQFKDYLE IDGVVKVGHK ASYDAELRER LLELPHPKSG PKPRIEWVAP PRLADISKET AELKRQYGFF E CSKFLACG ...文字列: MDSRFAQLTG VFCDDFTYSE GSRRFLSSYS TVERRPGVPV EGDCYDCLKN KWIAFELEGQ PRKFPKATVR CILNNDATYV CSEQEYQQI CKVQFKDYLE IDGVVKVGHK ASYDAELRER LLELPHPKSG PKPRIEWVAP PRLADISKET AELKRQYGFF E CSKFLACG EECGLDQEAR ELILNEYARD REFEFRNGGW IQRYTVASHK PATQKILPLP ASAPLARELL MLIARSTTQA GK VLHSDNT SILAVPVMRD SGKHSKRRPT ASTHHLVVGL SKPGCEHDFE FDGYRAAVHV MHLDPKQSAN IGEQDFVSTR EIY KLDMLE LPPISRKGDL DRASGLETRW DVILLLECLD STRVSQAVAQ HFNRHRLALS VCKDEFRKGY QLASEIRGTI PLSS LYYSL CAVRLRMTVH PFAR UniProtKB: Uncharacterized protein |
-分子 #2: Putative PB1
| 分子 | 名称: Putative PB1 / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Tilapia lake virus (ウイルス) Tilapia lake virus (ウイルス) |
| 分子量 | 理論値: 57.179375 KDa |
| 組換発現 | 生物種:  Trichoplusia ni (イラクサキンウワバ) Trichoplusia ni (イラクサキンウワバ) |
| 配列 | 文字列: MWAFQEGVCK GNLLSGPTSM KAPDSAARES IDRASEIMTG KSYNAVHTGD LSKLPNQGES PLRIVDSDLY SERSCCWVIE KEGRVVCKS TTLTRGMTSL LNTTKCSSPS ELICKVLTVE SLSEKIGDTS VEELLSHGRY FKCALRDQER GKPKSRAIFL S HPFFRLLS ...文字列: MWAFQEGVCK GNLLSGPTSM KAPDSAARES IDRASEIMTG KSYNAVHTGD LSKLPNQGES PLRIVDSDLY SERSCCWVIE KEGRVVCKS TTLTRGMTSL LNTTKCSSPS ELICKVLTVE SLSEKIGDTS VEELLSHGRY FKCALRDQER GKPKSRAIFL S HPFFRLLS SVVETHARSV LSKVSAVYTA TASAEQRAMM AAQVVESRKH VLNGDCTKYN EAIDADTLLK VWDAIGMGSI GV MLAYMVR RKCVLIKDTL VECPGGMLMG MFNATATLAL QGTTDRFLSF SDDFITSFNS PAELREIEDL LFASCHNLSL KKS YISVAS LEINSCTLTR DGDLATGLGC TAGVPFRGPL VTLKQTAAML SGAVDSGVMP FHSAERLFQI KQQECAYRYN NPTY TTRNE DFLPTCLGGK TVISFQSLLT WDCHPFWYQV HPDGPDTIDQ KVLSVLASKT RRRRTRLEAL SDLDPLVPHR LLVSE SDVS KIRAARQAHL KSLGLEQPTN FNYAIYKAVQ PTAGC UniProtKB: Putative PB1 |
-分子 #3: RNA-dependent RNA polymerase
| 分子 | 名称: RNA-dependent RNA polymerase / タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Tilapia lake virus (ウイルス) Tilapia lake virus (ウイルス) |
| 分子量 | 理論値: 53.782141 KDa |
| 組換発現 | 生物種:  Trichoplusia ni (イラクサキンウワバ) Trichoplusia ni (イラクサキンウワバ) |
| 配列 | 文字列: MSQFGKSFKG RTEVTITEYR SHTVKDVHRS LLTADKSLRK SFCFRNALNQ FLDKDLPLLP IRPKLESRVA VKKSKLRSQL SFRPGLTQE EAIDLYNKGY DGDSVSGALQ DRVVNEPVAY SSADNDKFHR GLAALGYTLA DRAFDTCESG FVRAIPTTPC G FICCGPGS ...文字列: MSQFGKSFKG RTEVTITEYR SHTVKDVHRS LLTADKSLRK SFCFRNALNQ FLDKDLPLLP IRPKLESRVA VKKSKLRSQL SFRPGLTQE EAIDLYNKGY DGDSVSGALQ DRVVNEPVAY SSADNDKFHR GLAALGYTLA DRAFDTCESG FVRAIPTTPC G FICCGPGS FKDSLGFVIK IGEFWHMYDG FQHFVAVEDA KFLASKSPSF WLAKRLAKRL NLVPKEDPSV AAAECPCKKV WE ASFARAP TALDPFGGRA FCDQGWVYHR DVGYATANHI SQETLFQQAL SVRNLGPQGS ANVSGSIHTA LDRLRAAYSR GTP ASRSIL QGLANLITPV GENFECDLDK RKLNIKALRS PERYITIEGL VVNLDDVVRG FYLDKAKVTV LSRSKWMGYE DLPQ KPPNG TFYCRKRKAM LLISCSPGTY AKKRKVAVQE DRFKDMRVEN FREVAENMDL NQGSGSENLY FQGHHHHHHH HHH UniProtKB: RNA-dependent RNA polymerase |
-分子 #4: 5' cRNA end - cRNA loop (40-mer)
| 分子 | 名称: 5' cRNA end - cRNA loop (40-mer) / タイプ: rna / ID: 4 / コピー数: 2 |
|---|---|
| 由来(天然) | 生物種:  Tilapia lake virus (ウイルス) Tilapia lake virus (ウイルス) |
| 分子量 | 理論値: 12.82868 KDa |
| 配列 | 文字列: CCAAAUUUUA CUCACAAGUC AGGACGUGAG AAAGAUUUGC |
-分子 #5: ZINC ION
| 分子 | 名称: ZINC ION / タイプ: ligand / ID: 5 / コピー数: 1 / 式: ZN |
|---|---|
| 分子量 | 理論値: 65.409 Da |
-分子 #6: MAGNESIUM ION
| 分子 | 名称: MAGNESIUM ION / タイプ: ligand / ID: 6 / コピー数: 1 / 式: MG |
|---|---|
| 分子量 | 理論値: 24.305 Da |
-分子 #7: water
| 分子 | 名称: water / タイプ: ligand / ID: 7 / コピー数: 1 / 式: HOH |
|---|---|
| 分子量 | 理論値: 18.015 Da |
| Chemical component information |  ChemComp-HOH: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 8 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 平均電子線量: 50.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.0 µm / 最小 デフォーカス(公称値): 0.8 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: AB INITIO MODEL |
|---|---|
| 得られたモデル |  PDB-8psq: |
 ムービー
ムービー コントローラー
コントローラー
















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)