+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Rapid screening cryo-EM map of CAK bound to dinaciclib (grid VC4-1, 1hr collection) | |||||||||
 マップデータ マップデータ | Sharpened cryo-EM map | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Kinase / Inhibitor / Transcription / Cell Cycle / TRANSFERASE | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報RNA polymerase II CTD heptapeptide repeat S5 kinase activity / snRNA transcription by RNA polymerase II / CAK-ERCC2 complex / transcription factor TFIIK complex / transcription factor TFIIH core complex / transcription factor TFIIH holo complex / [RNA-polymerase]-subunit kinase / RNA Polymerase I Transcription Termination / cyclin-dependent protein serine/threonine kinase regulator activity / RNA Pol II CTD phosphorylation and interaction with CE during HIV infection ...RNA polymerase II CTD heptapeptide repeat S5 kinase activity / snRNA transcription by RNA polymerase II / CAK-ERCC2 complex / transcription factor TFIIK complex / transcription factor TFIIH core complex / transcription factor TFIIH holo complex / [RNA-polymerase]-subunit kinase / RNA Polymerase I Transcription Termination / cyclin-dependent protein serine/threonine kinase regulator activity / RNA Pol II CTD phosphorylation and interaction with CE during HIV infection / RNA Pol II CTD phosphorylation and interaction with CE / Formation of the Early Elongation Complex / Formation of the HIV-1 Early Elongation Complex / HIV Transcription Initiation / RNA Polymerase II HIV Promoter Escape / Transcription of the HIV genome / RNA Polymerase II Promoter Escape / RNA Polymerase II Transcription Pre-Initiation And Promoter Opening / RNA Polymerase II Transcription Initiation / RNA Polymerase II Transcription Initiation And Promoter Clearance / mRNA Capping / regulation of G1/S transition of mitotic cell cycle / RNA Polymerase I Transcription Initiation / RNA polymerase II transcribes snRNA genes / ATP-dependent activity, acting on DNA / cyclin-dependent kinase / Tat-mediated elongation of the HIV-1 transcript / cyclin-dependent protein serine/threonine kinase activity / Formation of HIV-1 elongation complex containing HIV-1 Tat / Formation of HIV elongation complex in the absence of HIV Tat / Cyclin E associated events during G1/S transition / Cyclin A/B1/B2 associated events during G2/M transition / Cyclin A:Cdk2-associated events at S phase entry / RNA Polymerase II Transcription Elongation / cyclin-dependent protein kinase holoenzyme complex / Formation of RNA Pol II elongation complex / RNA Polymerase II Pre-transcription Events / RNA polymerase II CTD heptapeptide repeat kinase activity / male germ cell nucleus / TP53 Regulates Transcription of DNA Repair Genes / transcription initiation at RNA polymerase II promoter / RNA Polymerase I Promoter Escape / Transcription-Coupled Nucleotide Excision Repair (TC-NER) / Formation of TC-NER Pre-Incision Complex / Formation of Incision Complex in GG-NER / NoRC negatively regulates rRNA expression / Dual incision in TC-NER / Gap-filling DNA repair synthesis and ligation in TC-NER / Cyclin D associated events in G1 / fibrillar center / kinase activity / RUNX1 regulates transcription of genes involved in differentiation of HSCs / transcription by RNA polymerase II / protein kinase activity / regulation of cell cycle / protein stabilization / cell division / protein serine kinase activity / DNA repair / protein serine/threonine kinase activity / regulation of transcription by RNA polymerase II / perinuclear region of cytoplasm / positive regulation of transcription by RNA polymerase II / nucleoplasm / ATP binding / nucleus / plasma membrane / cytosol / cytoplasm 類似検索 - 分子機能 | |||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.5 Å | |||||||||
 データ登録者 データ登録者 | Cushing VI / Koh AF / Feng J / Jurgaityte K / Bahl AK / Ali S / Kotecha A / Greber BJ | |||||||||
| 資金援助 |  英国, 1件 英国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2024 ジャーナル: Nat Commun / 年: 2024タイトル: High-resolution cryo-EM of the human CDK-activating kinase for structure-based drug design. 著者: Victoria I Cushing / Adrian F Koh / Junjie Feng / Kaste Jurgaityte / Alexander Bondke / Sebastian H B Kroll / Marion Barbazanges / Bodo Scheiper / Ash K Bahl / Anthony G M Barrett / Simak Ali ...著者: Victoria I Cushing / Adrian F Koh / Junjie Feng / Kaste Jurgaityte / Alexander Bondke / Sebastian H B Kroll / Marion Barbazanges / Bodo Scheiper / Ash K Bahl / Anthony G M Barrett / Simak Ali / Abhay Kotecha / Basil J Greber /     要旨: Rational design of next-generation therapeutics can be facilitated by high-resolution structures of drug targets bound to small-molecule inhibitors. However, application of structure-based methods to ...Rational design of next-generation therapeutics can be facilitated by high-resolution structures of drug targets bound to small-molecule inhibitors. However, application of structure-based methods to macromolecules refractory to crystallization has been hampered by the often-limiting resolution and throughput of cryogenic electron microscopy (cryo-EM). Here, we use high-resolution cryo-EM to determine structures of the CDK-activating kinase, a master regulator of cell growth and division, in its free and nucleotide-bound states and in complex with 15 inhibitors at up to 1.8 Å resolution. Our structures provide detailed insight into inhibitor interactions and networks of water molecules in the active site of cyclin-dependent kinase 7 and provide insights into the mechanisms contributing to inhibitor selectivity, thereby providing the basis for rational design of next-generation therapeutics. These results establish a methodological framework for the use of high-resolution cryo-EM in structure-based drug design. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_17476.map.gz emd_17476.map.gz | 14.8 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-17476-v30.xml emd-17476-v30.xml emd-17476.xml emd-17476.xml | 20 KB 20 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_17476.png emd_17476.png | 108.3 KB | ||
| Filedesc metadata |  emd-17476.cif.gz emd-17476.cif.gz | 5.8 KB | ||
| その他 |  emd_17476_half_map_1.map.gz emd_17476_half_map_1.map.gz emd_17476_half_map_2.map.gz emd_17476_half_map_2.map.gz | 14.5 MB 14.5 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-17476 http://ftp.pdbj.org/pub/emdb/structures/EMD-17476 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-17476 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-17476 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_17476_validation.pdf.gz emd_17476_validation.pdf.gz | 1.1 MB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_17476_full_validation.pdf.gz emd_17476_full_validation.pdf.gz | 1.1 MB | 表示 | |
| XML形式データ |  emd_17476_validation.xml.gz emd_17476_validation.xml.gz | 9.6 KB | 表示 | |
| CIF形式データ |  emd_17476_validation.cif.gz emd_17476_validation.cif.gz | 11 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-17476 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-17476 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-17476 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-17476 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8ormC  8p6vC  8p6wC  8p6xC  8p6yC  8p6zC  8p70C  8p71C  8p72C  8p73C  8p74C  8p75C  8p76C  8p77C  8p78C  8p79C  8p7lC  8plzC C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_17476.map.gz / 形式: CCP4 / 大きさ: 15.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_17476.map.gz / 形式: CCP4 / 大きさ: 15.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Sharpened cryo-EM map | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.08 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: Half-map
| ファイル | emd_17476_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half-map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
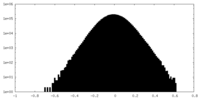
| 密度ヒストグラム |
-ハーフマップ: Half-map
| ファイル | emd_17476_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half-map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
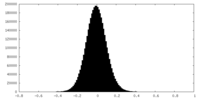
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : CDK-activating kinase
| 全体 | 名称: CDK-activating kinase |
|---|---|
| 要素 |
|
-超分子 #1: CDK-activating kinase
| 超分子 | 名称: CDK-activating kinase / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all 詳細: CAK in complex with non-covalent inhibitor dinaciclib |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 85 KDa |
-分子 #1: MAT1
| 分子 | 名称: MAT1 / タイプ: protein_or_peptide / ID: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 組換発現 | 生物種:  Trichoplusia ni (イラクサキンウワバ) Trichoplusia ni (イラクサキンウワバ) |
| 配列 | 文字列: SNAPVTFSTG IKMGQHISLA PIHKLEEALY EYQPLQIETY GPHVPELEML GRLGYLNHVR AASPQDLAGG YTSSLACHRA LQDAFSGLF WQPS UniProtKB: Isoform 1 of CDK-activating kinase assembly factor MAT1 |
-分子 #2: Cyclin H
| 分子 | 名称: Cyclin H / タイプ: protein_or_peptide / ID: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 組換発現 | 生物種:  Trichoplusia ni (イラクサキンウワバ) Trichoplusia ni (イラクサキンウワバ) |
| 配列 | 文字列: (ACE)MYHNSSQKR HWTFSSEEQL ARLRADANRK FRCKAVANGK VLPNDPVFLE PHEEMTLCKY YEKRLLEFCS VFKPAM PRS VVGTACMYFK RFYLNNSVME YHPRIIMLTC AFLACKVDEF NVSSPQFVGN LRESPLGQEK ALEQILEYEL LLIQQLN FH ...文字列: (ACE)MYHNSSQKR HWTFSSEEQL ARLRADANRK FRCKAVANGK VLPNDPVFLE PHEEMTLCKY YEKRLLEFCS VFKPAM PRS VVGTACMYFK RFYLNNSVME YHPRIIMLTC AFLACKVDEF NVSSPQFVGN LRESPLGQEK ALEQILEYEL LLIQQLN FH LIVHNPYRPF EGFLIDLKTR YPILENPEIL RKTADDFLNR IALTDAYLLY TPSQIALTAI LSSASRAGIT MESYLSES L MLKENRTCLS QLLDIMKSMR NLVKKYEPPR SEEVAVLKQK LERCHSAELA LNVITKKRKG YEDDDYVSKK SKHEEEEWT DDDLVESL UniProtKB: Cyclin-H |
-分子 #3: CDK7
| 分子 | 名称: CDK7 / タイプ: protein_or_peptide / ID: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 組換発現 | 生物種:  Trichoplusia ni (イラクサキンウワバ) Trichoplusia ni (イラクサキンウワバ) |
| 配列 | 文字列: SNAMALDVKS RAKRYEKLDF LGEGQFATVY KARDKNTNQI VAIKKIKLGH RSEAKDGINR TALREIKLLQ ELSHPNIIGL LDAFGHKSN ISLVFDFMET DLEVIIKDNS LVLTPSHIKA YMLMTLQGLE YLHQHWILHR DLKPNNLLLD ENGVLKLADF G LAKSFGSP ...文字列: SNAMALDVKS RAKRYEKLDF LGEGQFATVY KARDKNTNQI VAIKKIKLGH RSEAKDGINR TALREIKLLQ ELSHPNIIGL LDAFGHKSN ISLVFDFMET DLEVIIKDNS LVLTPSHIKA YMLMTLQGLE YLHQHWILHR DLKPNNLLLD ENGVLKLADF G LAKSFGSP NRAYTHQVVT RWYRAPELLF GARMYGVGVD MWAVGCILAE LLLRVPFLPG DSDLDQLTRI FETLGTPTEE QW PDMCSLP DYVTFKSFPG IPLHHIFSAA GDDLLDLIQG LFLFNPCARI TATQALKMKY FSNRPGPTPG CQLPRPNCPV ETL KEQSNP ALAIKRKRTE ALEQGGLPKK LIF UniProtKB: Cyclin-dependent kinase 7 |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.4 mg/mL | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.9 構成要素:
| |||||||||||||||
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: GOLD / メッシュ: 300 / 支持フィルム - 材質: GOLD / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: PLASMA CLEANING / 前処理 - 時間: 50 sec. | |||||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 278 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | TFS GLACIOS |
|---|---|
| 特殊光学系 | エネルギーフィルター - 名称: TFS Selectris X / エネルギーフィルター - スリット幅: 10 eV |
| 撮影 | フィルム・検出器のモデル: TFS FALCON 4i (4k x 4k) 撮影したグリッド数: 1 / 実像数: 466 / 平均電子線量: 70.0 e/Å2 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 20.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 1.2 µm / 最小 デフォーカス(公称値): 0.8 µm / 倍率(公称値): 205000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
 ムービー
ムービー コントローラー
コントローラー












































































 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)