+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-1746 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | P22 F170L C1 polyheads | |||||||||
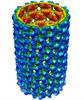
 マップデータ マップデータ | This is a helical reconstruction of a P22 polyhead with F170L coat protein with C1 symmetry | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | cryo-TEM / image reconstruction / helical reconstruction / bacteriophage P22 / virus assembly / coat protein | |||||||||
| 生物種 |  Enterobacteria phage P22 (ファージ) Enterobacteria phage P22 (ファージ) | |||||||||
| 手法 | らせん対称体再構成法 / クライオ電子顕微鏡法 / 解像度: 13.0 Å | |||||||||
 データ登録者 データ登録者 | Parent KN / Sinkovits RS / Suhanovsky MM / Teschke CM / Egelman EH / Baker TS | |||||||||
 引用 引用 |  ジャーナル: Phys Biol / 年: 2010 ジャーナル: Phys Biol / 年: 2010タイトル: Cryo-reconstructions of P22 polyheads suggest that phage assembly is nucleated by trimeric interactions among coat proteins. 著者: Kristin N Parent / Robert S Sinkovits / Margaret M Suhanovsky / Carolyn M Teschke / Edward H Egelman / Timothy S Baker /  要旨: Bacteriophage P22 forms an isometric capsid during normal assembly, yet when the coat protein (CP) is altered at a single site, helical structures (polyheads) also form. The structures of three ...Bacteriophage P22 forms an isometric capsid during normal assembly, yet when the coat protein (CP) is altered at a single site, helical structures (polyheads) also form. The structures of three distinct polyheads obtained from F170L and F170A variants were determined by cryo-reconstruction methods. An understanding of the structures of aberrant assemblies such as polyheads helps to explain how amino acid substitutions affect the CP, and these results can now be put into the context of CP pseudo-atomic models. F170L CP forms two types of polyhead and each has the CP organized as hexons (oligomers of six CPs). These hexons have a skewed structure similar to that in procapsids (precursor capsids formed prior to dsDNA packaging), yet their organization differs completely in polyheads and procapsids. F170A CP forms only one type of polyhead, and though this has hexons organized similarly to hexons in F170L polyheads, the hexons are isometric structures like those found in mature virions. The hexon organization in all three polyheads suggests that nucleation of procapsid assembly occurs via a trimer of CP monomers, and this drives formation of a T = 7, isometric particle. These variants also form procapsids, but they mature quite differently: F170A expands spontaneously at room temperature, whereas F170L requires more energy. The P22 CP structure along with scaffolding protein interactions appear to dictate curvature and geometry in assembled structures and residue 170 significantly influences both assembly and maturation. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_1746.map.gz emd_1746.map.gz | 167.8 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-1746-v30.xml emd-1746-v30.xml emd-1746.xml emd-1746.xml | 10.3 KB 10.3 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  1746.png 1746.png | 570.6 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-1746 http://ftp.pdbj.org/pub/emdb/structures/EMD-1746 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-1746 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-1746 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_1746_validation.pdf.gz emd_1746_validation.pdf.gz | 259.2 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_1746_full_validation.pdf.gz emd_1746_full_validation.pdf.gz | 258.3 KB | 表示 | |
| XML形式データ |  emd_1746_validation.xml.gz emd_1746_validation.xml.gz | 7.2 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1746 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1746 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1746 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1746 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_1746.map.gz / 形式: CCP4 / 大きさ: 173.8 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_1746.map.gz / 形式: CCP4 / 大きさ: 173.8 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | This is a helical reconstruction of a P22 polyhead with F170L coat protein with C1 symmetry | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.883 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : F170L coat protein in C1 helical lattice
| 全体 | 名称: F170L coat protein in C1 helical lattice |
|---|---|
| 要素 |
|
-超分子 #1000: F170L coat protein in C1 helical lattice
| 超分子 | 名称: F170L coat protein in C1 helical lattice / タイプ: sample / ID: 1000 詳細: This sample was generated by concentrating coat protein monomers. The asymmetric unit is a hexon. Number unique components: 1 |
|---|
-超分子 #1: Enterobacteria phage P22
| 超分子 | 名称: Enterobacteria phage P22 / タイプ: virus / ID: 1 / Name.synonym: Coat protein / NCBI-ID: 10754 / 生物種: Enterobacteria phage P22 / データベース: NCBI / ウイルスタイプ: OTHER / ウイルス・単離状態: STRAIN / ウイルス・エンベロープ: No / ウイルス・中空状態: Yes / Syn species name: Coat protein |
|---|---|
| 宿主 | 生物種:  Salmonella enterica subsp. enterica serovar Typhimurium (サルモネラ菌) Salmonella enterica subsp. enterica serovar Typhimurium (サルモネラ菌)別称: BACTERIA(EUBACTERIA) |
| 分子量 | 実験値: 47 MDa / 理論値: 47 MDa |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | らせん対称体再構成法 |
| 試料の集合状態 | filament |
- 試料調製
試料調製
| 濃度 | 35 mg/mL |
|---|---|
| 緩衝液 | pH: 7.6 / 詳細: 20 mM sodium phosphate |
| グリッド | 詳細: Lacey-carbon |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 89 K / 装置: HOMEMADE PLUNGER / 詳細: Vitrification instrument: Manual plunge-freezer / 手法: Blot for 5 seconds before freezing |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI POLARA 300 |
|---|---|
| 温度 | 最低: 90 K / 最高: 90 K / 平均: 90 K |
| アライメント法 | Legacy - 非点収差: At working magnification |
| 日付 | 2008年10月10日 |
| 撮影 | カテゴリ: CCD フィルム・検出器のモデル: GENERIC GATAN (4k x 4k) デジタル化 - サンプリング間隔: 1.883 µm / 実像数: 1141 / 平均電子線量: 19 e/Å2 / Od range: 1.5 / ビット/ピクセル: 16 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 倍率(補正後): 79666 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.3 mm / 最大 デフォーカス(公称値): 3.39 µm / 最小 デフォーカス(公称値): 1.14 µm / 倍率(公称値): 79666 |
| 試料ステージ | 試料ホルダー: Polara Multi Specimen Holder / 試料ホルダーモデル: GATAN LIQUID NITROGEN |
| 実験機器 |  モデル: Tecnai Polara / 画像提供: FEI Company |
- 画像解析
画像解析
| 詳細 | This reconstruction was done with the iterative helical real space reconstruction method |
|---|---|
| 最終 再構成 | 想定した対称性 - らせんパラメータ - Δz: 10.5 Å 想定した対称性 - らせんパラメータ - ΔΦ: 37.4 ° 想定した対称性 - らせんパラメータ - 軸対称性: C1 (非対称) アルゴリズム: OTHER / 解像度のタイプ: BY AUTHOR / 解像度: 13.0 Å / 解像度の算出法: OTHER / ソフトウェア - 名称: IHRSR |
| CTF補正 | 詳細: ROBEM |
 ムービー
ムービー コントローラー
コントローラー









 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)





















