+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | S. cerevisiae consensus-sCMGE on ssDNA after DNA replication initiation | |||||||||||||||
 マップデータ マップデータ | EM map of consensus-sCMGE | |||||||||||||||
 試料 試料 |
| |||||||||||||||
 キーワード キーワード | Saccharomyces cerevisiae / helicase / CMGE / initiation of DNA replication / DNA / DNA unwinding / REPLICATION | |||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報DNA-templated DNA replication maintenance of fidelity / gene conversion / Unwinding of DNA / DNA replication initiation / epsilon DNA polymerase complex / MCM core complex / Assembly of the pre-replicative complex / Switching of origins to a post-replicative state / DNA strand elongation involved in mitotic DNA replication / GINS complex ...DNA-templated DNA replication maintenance of fidelity / gene conversion / Unwinding of DNA / DNA replication initiation / epsilon DNA polymerase complex / MCM core complex / Assembly of the pre-replicative complex / Switching of origins to a post-replicative state / DNA strand elongation involved in mitotic DNA replication / GINS complex / MCM complex binding / mitotic DNA replication preinitiation complex assembly / nuclear DNA replication / premeiotic DNA replication / replication fork protection complex / pre-replicative complex assembly involved in nuclear cell cycle DNA replication / SUMO binding / nucleotide-excision repair, DNA gap filling / Activation of the pre-replicative complex / mitotic DNA replication / CMG complex / nuclear pre-replicative complex / DNA replication proofreading / Termination of translesion DNA synthesis / Activation of ATR in response to replication stress / DNA replication preinitiation complex / single-stranded DNA 3'-5' DNA exonuclease activity / MCM complex / mitotic DNA replication checkpoint signaling / double-strand break repair via break-induced replication / mitotic DNA replication initiation / single-stranded DNA helicase activity / mitotic intra-S DNA damage checkpoint signaling / silent mating-type cassette heterochromatin formation / 加水分解酵素; エステル加水分解酵素; 5'-リン酸モノエステル産生エンドデオキシリボヌクレアーゼ / regulation of DNA-templated DNA replication initiation / mitotic sister chromatid cohesion / DNA strand elongation involved in DNA replication / leading strand elongation / nuclear replication fork / DNA replication origin binding / Dual incision in TC-NER / DNA replication initiation / error-prone translesion synthesis / subtelomeric heterochromatin formation / base-excision repair, gap-filling / DNA helicase activity / replication fork / transcription elongation by RNA polymerase II / helicase activity / base-excision repair / double-strand break repair via nonhomologous end joining / DNA-templated DNA replication / heterochromatin formation / double-strand break repair / mitotic cell cycle / single-stranded DNA binding / 4 iron, 4 sulfur cluster binding / double-stranded DNA binding / DNA helicase / DNA-directed DNA polymerase / DNA-directed DNA polymerase activity / chromosome, telomeric region / DNA replication / nucleotide binding / mRNA binding / DNA damage response / chromatin binding / ATP hydrolysis activity / DNA binding / zinc ion binding / nucleoplasm / ATP binding / nucleus / cytoplasm 類似検索 - 分子機能 | |||||||||||||||
| 生物種 |  | |||||||||||||||
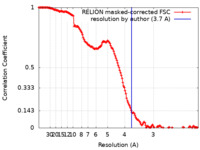
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.7 Å | |||||||||||||||
 データ登録者 データ登録者 | Henrikus SS / Willhoft O | |||||||||||||||
| 資金援助 |  英国, European Union, 英国, European Union,  フランス, 4件 フランス, 4件
| |||||||||||||||
 引用 引用 |  ジャーナル: Nat Struct Mol Biol / 年: 2024 ジャーナル: Nat Struct Mol Biol / 年: 2024タイトル: Unwinding of a eukaryotic origin of replication visualized by cryo-EM. 著者: Sarah S Henrikus / Marta H Gross / Oliver Willhoft / Thomas Pühringer / Jacob S Lewis / Allison W McClure / Julia F Greiwe / Giacomo Palm / Andrea Nans / John F X Diffley / Alessandro Costa /    要旨: To prevent detrimental chromosome re-replication, DNA loading of a double hexamer of the minichromosome maintenance (MCM) replicative helicase is temporally separated from DNA unwinding. Upon S-phase ...To prevent detrimental chromosome re-replication, DNA loading of a double hexamer of the minichromosome maintenance (MCM) replicative helicase is temporally separated from DNA unwinding. Upon S-phase transition in yeast, DNA unwinding is achieved in two steps: limited opening of the double helix and topological separation of the two DNA strands. First, Cdc45, GINS and Polε engage MCM to assemble a double CMGE with two partially separated hexamers that nucleate DNA melting. In the second step, triggered by Mcm10, two CMGEs separate completely, eject the lagging-strand template and cross paths. To understand Mcm10 during helicase activation, we used biochemical reconstitution with cryogenic electron microscopy. We found that Mcm10 splits the double CMGE by engaging the N-terminal homo-dimerization face of MCM. To eject the lagging strand, DNA unwinding is started from the N-terminal side of MCM while the hexamer channel becomes too narrow to harbor duplex DNA. | |||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_17459.map.gz emd_17459.map.gz | 263.3 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-17459-v30.xml emd-17459-v30.xml emd-17459.xml emd-17459.xml | 43.7 KB 43.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_17459_fsc.xml emd_17459_fsc.xml | 14.9 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_17459.png emd_17459.png | 166.7 KB | ||
| Filedesc metadata |  emd-17459.cif.gz emd-17459.cif.gz | 13.3 KB | ||
| その他 |  emd_17459_half_map_1.map.gz emd_17459_half_map_1.map.gz emd_17459_half_map_2.map.gz emd_17459_half_map_2.map.gz | 225.2 MB 225.3 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-17459 http://ftp.pdbj.org/pub/emdb/structures/EMD-17459 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-17459 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-17459 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_17459_validation.pdf.gz emd_17459_validation.pdf.gz | 1.1 MB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_17459_full_validation.pdf.gz emd_17459_full_validation.pdf.gz | 1.1 MB | 表示 | |
| XML形式データ |  emd_17459_validation.xml.gz emd_17459_validation.xml.gz | 22.3 KB | 表示 | |
| CIF形式データ |  emd_17459_validation.cif.gz emd_17459_validation.cif.gz | 29.5 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-17459 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-17459 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-17459 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-17459 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8p63MC  8p5eC  8p62C M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_17459.map.gz / 形式: CCP4 / 大きさ: 282.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_17459.map.gz / 形式: CCP4 / 大きさ: 282.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | EM map of consensus-sCMGE | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.08 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: EM half map 1 of consensus-sCMGE
| ファイル | emd_17459_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | EM half map 1 of consensus-sCMGE | ||||||||||||
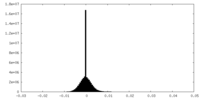
| 投影像・断面図 |
| ||||||||||||
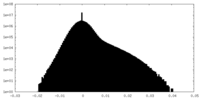
| 密度ヒストグラム |
-ハーフマップ: EM half map 2 of consensus-sCMGE
| ファイル | emd_17459_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | EM half map 2 of consensus-sCMGE | ||||||||||||
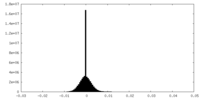
| 投影像・断面図 |
| ||||||||||||
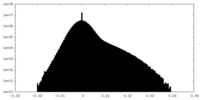
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
+全体 : Complex of Mcm2-7, Cdc45, GINS (Psf1-3, Sld5) DNA polymerase epsi...
+超分子 #1: Complex of Mcm2-7, Cdc45, GINS (Psf1-3, Sld5) DNA polymerase epsi...
+超分子 #2: DNA replication complex GINS containing Psf1, Psf2, Psf3 and Sld5
+超分子 #3: DNA
+超分子 #4: DNA replication licensing factors MCM2-7
+超分子 #5: DNA polymerase epsilon contains catalytic subunit A and non-catal...
+超分子 #6: Cdc45
+分子 #1: DNA replication licensing factor MCM2
+分子 #2: DNA replication licensing factor MCM3
+分子 #3: DNA replication licensing factor MCM4
+分子 #4: Minichromosome maintenance protein 5
+分子 #5: DNA replication licensing factor MCM6
+分子 #6: DNA replication licensing factor MCM7
+分子 #8: DNA replication complex GINS protein PSF3
+分子 #9: DNA replication complex GINS protein SLD5
+分子 #10: Cell division control protein 45
+分子 #11: DNA polymerase epsilon subunit B
+分子 #12: DNA polymerase epsilon catalytic subunit A
+分子 #13: DNA replication complex GINS protein PSF1
+分子 #14: DNA replication complex GINS protein PSF2
+分子 #7: DNA (9-MER)
+分子 #15: ADENOSINE-5'-TRIPHOSPHATE
+分子 #16: ZINC ION
+分子 #17: MAGNESIUM ION
+分子 #18: ADENOSINE-5'-DIPHOSPHATE
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.6 |
|---|---|
| 凍結 | 凍結剤: ETHANE / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 平均電子線量: 1.67 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: SPOT SCAN / 撮影モード: BRIGHT FIELD 最大 デフォーカス(公称値): 3.3000000000000003 µm 最小 デフォーカス(公称値): 1.8 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)