+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | FAD_ox bound dark state structure of PdLCry | |||||||||||||||
 マップデータ マップデータ | structure of FAD_ox bound dark state PdLCry | |||||||||||||||
 試料 試料 |
| |||||||||||||||
 キーワード キーワード | light-sensitive / circalunar clock / FLAVOPROTEIN | |||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報entrainment of circadian clock by photoperiod / FAD binding / circadian regulation of gene expression / negative regulation of DNA-templated transcription / perinuclear region of cytoplasm / DNA binding / nucleus 類似検索 - 分子機能 | |||||||||||||||
| 生物種 |  Platynereis dumerilii (無脊椎動物) Platynereis dumerilii (無脊椎動物) | |||||||||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.57 Å | |||||||||||||||
 データ登録者 データ登録者 | Behrmann E / Behrmann H | |||||||||||||||
| 資金援助 |  ドイツ, 4件 ドイツ, 4件
| |||||||||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2023 ジャーナル: Nat Commun / 年: 2023タイトル: A marine cryptochrome with an inverse photo-oligomerization mechanism. 著者: Hong Ha Vu / Heide Behrmann / Maja Hanić / Gayathri Jeyasankar / Shruthi Krishnan / Dennis Dannecker / Constantin Hammer / Monika Gunkel / Ilia A Solov'yov / Eva Wolf / Elmar Behrmann /  要旨: Cryptochromes (CRYs) are a structurally conserved but functionally diverse family of proteins that can confer unique sensory properties to organisms. In the marine bristle worm Platynereis dumerilii, ...Cryptochromes (CRYs) are a structurally conserved but functionally diverse family of proteins that can confer unique sensory properties to organisms. In the marine bristle worm Platynereis dumerilii, its light receptive cryptochrome L-CRY (PdLCry) allows the animal to discriminate between sunlight and moonlight, an important requirement for synchronizing its lunar cycle-dependent mass spawning. Using cryo-electron microscopy, we show that in the dark, PdLCry adopts a dimer arrangement observed neither in plant nor insect CRYs. Intense illumination disassembles the dimer into monomers. Structural and functional data suggest a mechanistic coupling between the light-sensing flavin adenine dinucleotide chromophore, the dimer interface, and the C-terminal tail helix, with a likely involvement of the phosphate binding loop. Taken together, our work establishes PdLCry as a CRY protein with inverse photo-oligomerization with respect to plant CRYs, and provides molecular insights into how this protein might help discriminating the different light intensities associated with sunlight and moonlight. | |||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_17429.map.gz emd_17429.map.gz | 117.7 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-17429-v30.xml emd-17429-v30.xml emd-17429.xml emd-17429.xml | 24.6 KB 24.6 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_17429_fsc.xml emd_17429_fsc.xml | 10.5 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_17429.png emd_17429.png | 103.2 KB | ||
| Filedesc metadata |  emd-17429.cif.gz emd-17429.cif.gz | 7.1 KB | ||
| その他 |  emd_17429_additional_1.map.gz emd_17429_additional_1.map.gz emd_17429_half_map_1.map.gz emd_17429_half_map_1.map.gz emd_17429_half_map_2.map.gz emd_17429_half_map_2.map.gz | 3.1 MB 115.7 MB 115.7 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-17429 http://ftp.pdbj.org/pub/emdb/structures/EMD-17429 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-17429 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-17429 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_17429_validation.pdf.gz emd_17429_validation.pdf.gz | 812.3 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_17429_full_validation.pdf.gz emd_17429_full_validation.pdf.gz | 811.9 KB | 表示 | |
| XML形式データ |  emd_17429_validation.xml.gz emd_17429_validation.xml.gz | 19.3 KB | 表示 | |
| CIF形式データ |  emd_17429_validation.cif.gz emd_17429_validation.cif.gz | 24.8 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-17429 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-17429 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-17429 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-17429 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8p4xMC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_17429.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_17429.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | structure of FAD_ox bound dark state PdLCry | ||||||||||||||||||||||||||||||||||||
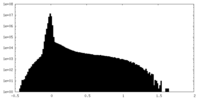
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.862 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-追加マップ: structure of FAD ox bound dark state PdLCry with...
| ファイル | emd_17429_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | structure of FAD_ox bound dark state PdLCry with local resolution filter applied | ||||||||||||
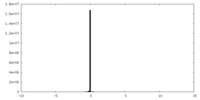
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: structure of FAD ox bound dark state PdLCry - odd particles
| ファイル | emd_17429_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | structure of FAD_ox bound dark state PdLCry - odd particles | ||||||||||||
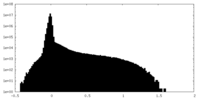
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: structure of FAD ox bound dark state PdLCry - even particles
| ファイル | emd_17429_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | structure of FAD_ox bound dark state PdLCry - even particles | ||||||||||||
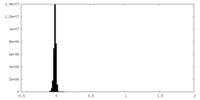
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : dimeric complex of PdLCry in the dark state
| 全体 | 名称: dimeric complex of PdLCry in the dark state |
|---|---|
| 要素 |
|
-超分子 #1: dimeric complex of PdLCry in the dark state
| 超分子 | 名称: dimeric complex of PdLCry in the dark state / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  Platynereis dumerilii (無脊椎動物) Platynereis dumerilii (無脊椎動物) |
| 分子量 | 理論値: 130 KDa |
-分子 #1: Putative light-receptive cryptochrome (Fragment)
| 分子 | 名称: Putative light-receptive cryptochrome (Fragment) / タイプ: protein_or_peptide / ID: 1 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Platynereis dumerilii (無脊椎動物) Platynereis dumerilii (無脊椎動物) |
| 分子量 | 理論値: 65.482855 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MKEKMSAWEV GNGIMEEKTD DWDNKEDNGK EHVSLHWFRH GLRLHDNPAL LKSLEGAKEF YALFIWDGEV AGTKLVSYPR MKFLLECLK DLDDSLKKHG GRLYVVKGPS DVVIKQLIEE WGVTRVTCEI DPEPIWQPRD KAVKDLCATK GVKWFDYNSH L LWDPKAVC ...文字列: MKEKMSAWEV GNGIMEEKTD DWDNKEDNGK EHVSLHWFRH GLRLHDNPAL LKSLEGAKEF YALFIWDGEV AGTKLVSYPR MKFLLECLK DLDDSLKKHG GRLYVVKGPS DVVIKQLIEE WGVTRVTCEI DPEPIWQPRD KAVKDLCATK GVKWFDYNSH L LWDPKAVC DANGGRPPHT YKLFCQVTDL LGKPETPHPD PDFSHVQMPV SDDFDDKFGL PTLKELGCEP ECEEQEKPFN KW QGGETGA LELLETRLMI ERTAYKAGYI MPNQYIPDLV GPPRSMSPHL RFGALSIRKF YWDLHNNYAE VCGGEWLGAL TAQ LVWREY FYCMSYGNPS FDKMEGNPIC LQIPWYKDEE ALEKWKQGQT GFPWIDACMR QLRYEGWMHH VGRHAVACFL TRGD LWISW VDGLEAFYKY MLDGDWSVCA GNWMWVSSSA FENCLQCPQC FSPVLYGMRM DPTGEFTRRY VPQLKNMPLK YLFQP WKAP KEVQEKAGCV IGEDYPSPMV DHKEASSKCR RMMEDVKSII KDPEVWHCTP SDTNEVRKFC WLPEHMTADQ PCLGDL PCI KY UniProtKB: Cryptochrome-1 |
-分子 #2: FLAVIN-ADENINE DINUCLEOTIDE
| 分子 | 名称: FLAVIN-ADENINE DINUCLEOTIDE / タイプ: ligand / ID: 2 / コピー数: 2 / 式: FAD |
|---|---|
| 分子量 | 理論値: 785.55 Da |
| Chemical component information |  ChemComp-FAD: |
-分子 #3: MAGNESIUM ION
| 分子 | 名称: MAGNESIUM ION / タイプ: ligand / ID: 3 / コピー数: 2 / 式: MG |
|---|---|
| 分子量 | 理論値: 24.305 Da |
-分子 #4: water
| 分子 | 名称: water / タイプ: ligand / ID: 4 / コピー数: 84 / 式: HOH |
|---|---|
| 分子量 | 理論値: 18.015 Da |
| Chemical component information |  ChemComp-HOH: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.7 mg/mL | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 8 構成要素:
| |||||||||||||||
| グリッド | モデル: UltrAuFoil R1.2/1.3 / 材質: GOLD / メッシュ: 300 / 前処理 - タイプ: GLOW DISCHARGE | |||||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 293.15 K / 装置: FEI VITROBOT MARK IV 詳細: grids were prepared under far-red light illumination. | |||||||||||||||
| 詳細 | grids were prepared under far-red light illumination (Osram OSLON SSL 120, GF CSSPM1.24) |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| ソフトウェア | 名称: EPU |
| 撮影 | フィルム・検出器のモデル: FEI FALCON III (4k x 4k) 平均電子線量: 30.0 e/Å2 / 詳細: see material+methods |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.0 µm / 最小 デフォーカス(公称値): 0.3 µm / 倍率(公称値): 96000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 初期モデル | Chain - Source name: Other / Chain - Initial model type: integrative model / 詳細: starting model was based on 4jzy |
|---|---|
| ソフトウェア | 名称:  Coot Coot |
| 精密化 | 空間: REAL / プロトコル: OTHER |
| 得られたモデル |  PDB-8p4x: |
 ムービー
ムービー コントローラー
コントローラー






 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)