+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of hantaan orthohantavirus (HTNV) polymerase - Apo core | ||||||||||||
 マップデータ マップデータ | |||||||||||||
 試料 試料 |
| ||||||||||||
 キーワード キーワード | RNA-dependent RNA polymerase / HTNV / Hantaan orthohantavirus / Bunyavirales / Hantaviridae. / VIRAL PROTEIN | ||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報RNA-templated viral transcription / negative stranded viral RNA replication / cap snatching / endonuclease activity / 加水分解酵素; エステル加水分解酵素 / host cell perinuclear region of cytoplasm / RNA-directed RNA polymerase / nucleotide binding / RNA-directed RNA polymerase activity / DNA-templated transcription / metal ion binding 類似検索 - 分子機能 | ||||||||||||
| 生物種 |  Hantaan orthohantavirus (ウイルス) Hantaan orthohantavirus (ウイルス) | ||||||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.78 Å | ||||||||||||
 データ登録者 データ登録者 | Keown JR / Carrique L / Grimes JM | ||||||||||||
| 資金援助 |  英国, 3件 英国, 3件
| ||||||||||||
 引用 引用 |  ジャーナル: PLoS Pathog / 年: 2024 ジャーナル: PLoS Pathog / 年: 2024タイトル: Structural characterization of the full-length Hantaan virus polymerase. 著者: Jeremy R Keown / Loïc Carrique / Benjamin E Nilsson-Payant / Ervin Fodor / Jonathan M Grimes /    要旨: Hantaviridae are a family of segmented negative-sense RNA viruses that contain important human and animal pathogens. Hantaviridae contain a viral RNA-dependent RNA polymerase that replicates and ...Hantaviridae are a family of segmented negative-sense RNA viruses that contain important human and animal pathogens. Hantaviridae contain a viral RNA-dependent RNA polymerase that replicates and transcribes the viral genome. Here we establish the expression and purification of the polymerase from the Old World Hantaan virus and characterise the structure using Cryo-EM. We determine a series of structures at resolutions between 2.7 and 3.3 Å of RNA free polymerase comprising the core, core and endonuclease, and a full-length polymerase. The full-length polymerase structure depicts the location of the cap binding and C-terminal domains which are arranged in a conformation that is incompatible with transcription and in a novel conformation not observed in previous conformations of cap-snatching viral polymerases. We further describe structures with 5' vRNA promoter in the presence and absence of a nucleotide triphosphate. The nucleotide bound structure mimics a replication pre-initiation complex and the nucleotide stabilises the motif E in a conformation distinct from those previously observed. We observe motif E in four distinct conformations including β-sheet, two helical arrangements, and nucleotide primed arrangement. The insights gained here guide future mechanistic studies of both the transcription and replication activities of the hantavirus polymerase and for the development of therapeutic targets. | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_17351.map.gz emd_17351.map.gz | 97 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-17351-v30.xml emd-17351-v30.xml emd-17351.xml emd-17351.xml | 20.2 KB 20.2 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_17351_fsc.xml emd_17351_fsc.xml | 11 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_17351.png emd_17351.png | 98.2 KB | ||
| マスクデータ |  emd_17351_msk_1.map emd_17351_msk_1.map | 103 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-17351.cif.gz emd-17351.cif.gz | 7.5 KB | ||
| その他 |  emd_17351_half_map_1.map.gz emd_17351_half_map_1.map.gz emd_17351_half_map_2.map.gz emd_17351_half_map_2.map.gz | 95.5 MB 95.5 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-17351 http://ftp.pdbj.org/pub/emdb/structures/EMD-17351 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-17351 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-17351 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_17351_validation.pdf.gz emd_17351_validation.pdf.gz | 903 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_17351_full_validation.pdf.gz emd_17351_full_validation.pdf.gz | 902.6 KB | 表示 | |
| XML形式データ |  emd_17351_validation.xml.gz emd_17351_validation.xml.gz | 18.4 KB | 表示 | |
| CIF形式データ |  emd_17351_validation.cif.gz emd_17351_validation.cif.gz | 23.7 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-17351 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-17351 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-17351 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-17351 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_17351.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_17351.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.932 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_17351_msk_1.map emd_17351_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
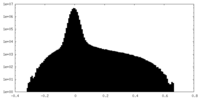
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_17351_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
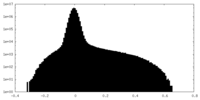
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_17351_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : RNA-dependent RNA polymerase of Hantaan orthohantavirus (HTNV).
| 全体 | 名称: RNA-dependent RNA polymerase of Hantaan orthohantavirus (HTNV). |
|---|---|
| 要素 |
|
-超分子 #1: RNA-dependent RNA polymerase of Hantaan orthohantavirus (HTNV).
| 超分子 | 名称: RNA-dependent RNA polymerase of Hantaan orthohantavirus (HTNV). タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Hantaan orthohantavirus (ウイルス) Hantaan orthohantavirus (ウイルス) |
| 分子量 | 理論値: 247 KDa |
-分子 #1: RNA-directed RNA polymerase L
| 分子 | 名称: RNA-directed RNA polymerase L / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO / EC番号: RNA-directed RNA polymerase |
|---|---|
| 由来(天然) | 生物種:  Hantaan orthohantavirus (ウイルス) Hantaan orthohantavirus (ウイルス) |
| 分子量 | 理論値: 251.806016 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MWSHPQFEKG GGSGGGSGGS SAWSHPQFEK HHHHHHHHEN LYFQGMDKYR EIHNKLKEFS PGTLTAVECI DYLDRLYAVR HDIVDQMIK HDWSDNKDSE EAIGKVLLFA GVPSNIITAL EKKIIPNHPT GKSLKAFFKM TPANYKISGT TIEFVEVTVT A DVDKGIRE ...文字列: MWSHPQFEKG GGSGGGSGGS SAWSHPQFEK HHHHHHHHEN LYFQGMDKYR EIHNKLKEFS PGTLTAVECI DYLDRLYAVR HDIVDQMIK HDWSDNKDSE EAIGKVLLFA GVPSNIITAL EKKIIPNHPT GKSLKAFFKM TPANYKISGT TIEFVEVTVT A DVDKGIRE KKLKYEAGLT YIEQELHKFF LKGEIPQPYK ITFNVVAVRT DGSNITTQWP SRRNDGVVQY MRLVQAEISY VR EHLIKTE ERAALEAMFN LKFNISTHKS QPYYIPDYKG MEPIGANIED LVDYSKDWLS RARNFSFFEV KGTAVFECFN SNE ANHCQR YPMSRKPRNF LLIQCSLITS YKPATTLSDQ IDSRRACSYI LNLIPDTPAS YLIHDMAYRY INLTREDMIN YYAP RIQFK QTQNVREPGT FKLTSSMLRA ESKAMLDLLN NHKSGEKHGA QIESLNIASH IVQSESVSLI TKILSDLELN ITEPS TQEY STTKHTYVDT VLDKFFQNET QKYLIDVLKK TTAWHIGHLI RDITESLIAH SGLKRSKYWS LHSYNNGNVI LFILPS KSL EVAGSFIRFI TVFRIGPGLV DKDNLDTILI DGDSQWGVSK VMSIDLNRLL ALNIAFEKAL IATATWFQYY TEDQGQF PL QYAIRSVFAN HFLLAICQKM KLCAIFDNLR YLIPAVTSLY SGFPSLIEKL FERPFKSSLE VYIYYNIKSL LVALAQNN K ARFYSKVKLL GLTVDQSTVG ASGVYPSFMS RIVYKHYRSL ISEVTTCFFL FEKGLHGNMN EEAKIHLETV EWALKFREK EEKYGESLVE NGYMMWELRA NAELAEQQLY CQDAIELAAI ELNKVLATKS SVVANSILSK NWEEPYFSQT RNISLKGMSG QVQEDGHLS SSVTIIEAIR YLSNSRHNPS LLKLYEETRE QKAMARIVRK YQRTEADRGF FITTLPTRCR LEIIEDYYDA I AKNISEEY ISYGGEKKIL AIQGALEKAL RWASGESFIE LSNHKFIRMK RKLMYVSADA TKWSPGDNSA KFRRFTSMLH NG LPNNKLK NCVIDALKQV YKTDFFMSRK LRNYIDSMES LDPHIKQFLD FFPDGHHGEV KGNWLQGNLN KCSSLFGVAM SLL FKQVWT NLFPELDCFF EFAHHSDDAL FIYGYLEPVD DGTDWFLFVS QQIQAGHLHW FSVNTEMWKS MFNLHEHILL LGSI KISPK KTTVSPTNAE FLSTFFEGCA VSIPFVKILL GSLSDLPGLG YFDDLAAAQS RCVKALDLGA SPQVAQLAVA LCTSK VERL YGTAPGMVNH PAAYLQVKHT DTPIPLGGNG AMSIMELATA GIGMSDKNLL KRALLGYSHK RQKSMLYILG LFKFLM KLS DETFQHERLG QFSFIGKVQW KIFTPKSEFE FADMYTSKFL ELWSSQHVTY DYIIPKGRDN LLIYLVRKLN DPSIVTA MT MQSPLQLRFR MQAKQHMKVC RLDGEWVTFR EVLAAANSFA ENYSATSQDM DLFQTLTSCT FSKEYAWKDF LNGIHCDV I PTKQVQRAKV ARTFTVREKD QIIQNSIPAV IGYKFAVTVE EMSDVLDTAK FPDSLSVDLK TMKDGVYREL GLDISLPDV MKRIAPMLYK SSKSRVVIVQ GNVEGTAEAI CRYWLKSMSL VKTIRVKPHK EVLQAVSIFN RKEDIGQQKD LAALKLCIEV WRWCKANSA PYRDWFQALW FEDKTFSEWL DRFCRVGVPP IDPEIQCAAL MIADIKGDYS VLQLQANRRA YSGKQYDAYC V QTYNEVTK LYEGDLRVTF NFGLDCARLE IFWDKKAYIL ETSITQKHVL KIMMDEVSKE LIKCGMRFNT EQVQGVRHMV LF KTESGFE WGKPNIPCIV YKNCVLRTSL RTTQAINHKF MITIKDDGLR AIAQHDEDSP RFLLAHAFHT IRDIRYQAVD AVS NVWFIH KGVKLYLNPI ISSGLLENFM KNLPAAIPPA AYSLIMNRAK ISVDLFMFND LLKLINPRNT LDLSGLETTG DEFS TVSSM SSRLWSEEMS LVDDDEELDD EFTIDLQDVD FENIDIEADI EHFLQDESSY TGDLLISTEE TESKKMRGIV KILEP VRLI KSWVSRGLSI EKVYSPVNII LMSRYISKTF NLSTKQVSLL DPYDLTELES IVRGWGECVI DQFESLDREA QNMVVN KGI CPEDVIPDSL FSFRHTMVLL RRLFPQDSIS SFY UniProtKB: RNA-directed RNA polymerase L |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.5 mg/mL | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.5 構成要素:
| |||||||||||||||
| グリッド | モデル: Quantifoil R2/1 / 材質: COPPER / メッシュ: 200 / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 45 sec. | |||||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 293 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | TFS KRIOS |
|---|---|
| 特殊光学系 | エネルギーフィルター - 名称: TFS Selectris X / エネルギーフィルター - スリット幅: 10 eV |
| 撮影 | フィルム・検出器のモデル: FEI FALCON IV (4k x 4k) 平均電子線量: 50.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 50.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.6 µm 最小 デフォーカス(公称値): 1.4000000000000001 µm |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 初期モデル | Chain - Source name: AlphaFold / Chain - Initial model type: in silico model |
|---|---|
| 精密化 | 空間: REAL / プロトコル: FLEXIBLE FIT 当てはまり具合の基準: Cross-correlation coefficient |
| 得られたモデル |  PDB-8p1j: |
 ムービー
ムービー コントローラー
コントローラー












 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)