+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Escherichia coli paused disome complex (rotated-PRE-1 | rotated PRE) | |||||||||
 マップデータ マップデータ | Cryo-EM reconstruction of the E. coli disome complex (rotated-PRE-1 | rotated PRE) | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | ribosome / polysome / tranlsation / elongation / pausing / disome / collision / PURE system | |||||||||
| 生物種 |  | |||||||||
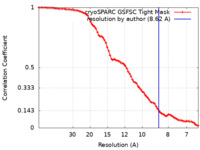
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 8.62 Å | |||||||||
 データ登録者 データ登録者 | Fluegel T / Schacherl M | |||||||||
| 資金援助 |  ドイツ, 1件 ドイツ, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2024 ジャーナル: Nat Commun / 年: 2024タイトル: Transient disome complex formation in native polysomes during ongoing protein synthesis captured by cryo-EM. 著者: Timo Flügel / Magdalena Schacherl / Anett Unbehaun / Birgit Schroeer / Marylena Dabrowski / Jörg Bürger / Thorsten Mielke / Thiemo Sprink / Christoph A Diebolder / Yollete V Guillén ...著者: Timo Flügel / Magdalena Schacherl / Anett Unbehaun / Birgit Schroeer / Marylena Dabrowski / Jörg Bürger / Thorsten Mielke / Thiemo Sprink / Christoph A Diebolder / Yollete V Guillén Schlippe / Christian M T Spahn /  要旨: Structural studies of translating ribosomes traditionally rely on in vitro assembly and stalling of ribosomes in defined states. To comprehensively visualize bacterial translation, we reactivated ex ...Structural studies of translating ribosomes traditionally rely on in vitro assembly and stalling of ribosomes in defined states. To comprehensively visualize bacterial translation, we reactivated ex vivo-derived E. coli polysomes in the PURE in vitro translation system and analyzed the actively elongating polysomes by cryo-EM. We find that 31% of 70S ribosomes assemble into disome complexes that represent eight distinct functional states including decoding and termination intermediates, and a pre-nucleophilic attack state. The functional diversity of disome complexes together with RNase digest experiments suggests that paused disome complexes transiently form during ongoing elongation. Structural analysis revealed five disome interfaces between leading and queueing ribosomes that undergo rearrangements as the leading ribosome traverses through the elongation cycle. Our findings reveal at the molecular level how bL9's CTD obstructs the factor binding site of queueing ribosomes to thwart harmful collisions and illustrate how translation dynamics reshape inter-ribosomal contacts. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_17288.map.gz emd_17288.map.gz | 23.3 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-17288-v30.xml emd-17288-v30.xml emd-17288.xml emd-17288.xml | 22.8 KB 22.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_17288_fsc.xml emd_17288_fsc.xml | 7.7 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_17288.png emd_17288.png | 79.9 KB | ||
| マスクデータ |  emd_17288_msk_1.map emd_17288_msk_1.map | 48.9 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-17288.cif.gz emd-17288.cif.gz | 4.4 KB | ||
| その他 |  emd_17288_half_map_1.map.gz emd_17288_half_map_1.map.gz emd_17288_half_map_2.map.gz emd_17288_half_map_2.map.gz | 45.3 MB 45.3 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-17288 http://ftp.pdbj.org/pub/emdb/structures/EMD-17288 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-17288 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-17288 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_17288_validation.pdf.gz emd_17288_validation.pdf.gz | 1.1 MB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_17288_full_validation.pdf.gz emd_17288_full_validation.pdf.gz | 1.1 MB | 表示 | |
| XML形式データ |  emd_17288_validation.xml.gz emd_17288_validation.xml.gz | 15.7 KB | 表示 | |
| CIF形式データ |  emd_17288_validation.cif.gz emd_17288_validation.cif.gz | 20.4 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-17288 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-17288 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-17288 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-17288 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_17288.map.gz / 形式: CCP4 / 大きさ: 48.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_17288.map.gz / 形式: CCP4 / 大きさ: 48.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Cryo-EM reconstruction of the E. coli disome complex (rotated-PRE-1 | rotated PRE) | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 3.18 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_17288_msk_1.map emd_17288_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
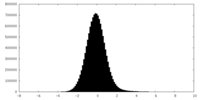
| 密度ヒストグラム |
-ハーフマップ: Cryo-EM reconstruction of the E. coli disome complex...
| ファイル | emd_17288_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Cryo-EM reconstruction of the E. coli disome complex (rotated-PRE-1 | rotated PRE). Half map B. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
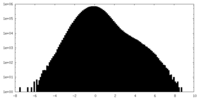
| 密度ヒストグラム |
-ハーフマップ: Cryo-EM reconstruction of the E. coli disome complex...
| ファイル | emd_17288_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Cryo-EM reconstruction of the E. coli disome complex (rotated-PRE-1 | rotated PRE). Half map A. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Escherichia coli paused disome complex
| 全体 | 名称: Escherichia coli paused disome complex |
|---|---|
| 要素 |
|
-超分子 #1: Escherichia coli paused disome complex
| 超分子 | 名称: Escherichia coli paused disome complex / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#60 |
|---|---|
| 由来(天然) | 生物種:  |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.6 |
|---|---|
| グリッド | モデル: Quantifoil R2/2 / 材質: COPPER / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 75 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 平均露光時間: 1.13 sec. / 平均電子線量: 45.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 50.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.0 µm / 最小 デフォーカス(公称値): 0.5 µm / 倍率(公称値): 81000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー




















































 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)