+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-1706 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM map of Lactococcal phage p2 baseplate consisting of ORF 15, 16 and 18. | |||||||||
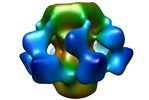
 マップデータ マップデータ | Cryo-EM map of p2 phage baseplate | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Baseplate / phage / P2 / Siphoviridae / orf 15 / orf 16 / orf 18 / RBP / cryo-EM | |||||||||
| 生物種 |  Lactococcus lactis phage p2 (ウイルス) Lactococcus lactis phage p2 (ウイルス) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 26.0 Å | |||||||||
 データ登録者 データ登録者 | Sciara G / Bebeacua C / Bron P / Tremblay D / Ortiz-Lombardia M / Lichiere J / Van Heel M / Campanacci V / Moineau S / Cambillau C | |||||||||
 引用 引用 |  ジャーナル: Proc Natl Acad Sci U S A / 年: 2010 ジャーナル: Proc Natl Acad Sci U S A / 年: 2010タイトル: Structure of lactococcal phage p2 baseplate and its mechanism of activation. 著者: Giuliano Sciara / Cecilia Bebeacua / Patrick Bron / Denise Tremblay / Miguel Ortiz-Lombardia / Julie Lichière / Marin van Heel / Valérie Campanacci / Sylvain Moineau / Christian Cambillau /  要旨: Siphoviridae is the most abundant viral family on earth which infects bacteria as well as archaea. All known siphophages infecting gram+ Lactococcus lactis possess a baseplate at the tip of their ...Siphoviridae is the most abundant viral family on earth which infects bacteria as well as archaea. All known siphophages infecting gram+ Lactococcus lactis possess a baseplate at the tip of their tail involved in host recognition and attachment. Here, we report analysis of the p2 phage baseplate structure by X-ray crystallography and electron microscopy and propose a mechanism for the baseplate activation during attachment to the host cell. This approximately 1 MDa, Escherichia coli-expressed baseplate is composed of three protein species, including six trimers of the receptor-binding protein (RBP). RBPs host-recognition domains point upwards, towards the capsid, in agreement with the electron-microscopy map of the free virion. In the presence of Ca(2+), a cation mandatory for infection, the RBPs rotated 200 degrees downwards, presenting their binding sites to the host, and a channel opens at the bottom of the baseplate for DNA passage. These conformational changes reveal a novel siphophage activation and host-recognition mechanism leading ultimately to DNA ejection. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_1706.map.gz emd_1706.map.gz | 625.4 KB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-1706-v30.xml emd-1706-v30.xml emd-1706.xml emd-1706.xml | 10.1 KB 10.1 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  1706.png 1706.png | 360.1 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-1706 http://ftp.pdbj.org/pub/emdb/structures/EMD-1706 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-1706 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-1706 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_1706_validation.pdf.gz emd_1706_validation.pdf.gz | 195.9 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_1706_full_validation.pdf.gz emd_1706_full_validation.pdf.gz | 195 KB | 表示 | |
| XML形式データ |  emd_1706_validation.xml.gz emd_1706_validation.xml.gz | 5.2 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1706 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1706 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1706 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1706 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_1706.map.gz / 形式: CCP4 / 大きさ: 8.2 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_1706.map.gz / 形式: CCP4 / 大きさ: 8.2 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Cryo-EM map of p2 phage baseplate | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 4.37 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Over-expressed Lactococcal phage p2 baseplate
| 全体 | 名称: Over-expressed Lactococcal phage p2 baseplate |
|---|---|
| 要素 |
|
-超分子 #1000: Over-expressed Lactococcal phage p2 baseplate
| 超分子 | 名称: Over-expressed Lactococcal phage p2 baseplate / タイプ: sample / ID: 1000 集合状態: One homotrimer of ORF16 binds to two hexamers of ORF15 and binds to six homotrimers of ORF18 Number unique components: 1 |
|---|---|
| 分子量 | 実験値: 1 MDa |
-分子 #1: p2 Lactoccocal phage baseplate
| 分子 | 名称: p2 Lactoccocal phage baseplate / タイプ: protein_or_peptide / ID: 1 / Name.synonym: p2 baseplate / 詳細: Complexes was cross-linked by glutaraldehyde / 組換発現: Yes |
|---|---|
| 由来(天然) | 生物種:  Lactococcus lactis phage p2 (ウイルス) Lactococcus lactis phage p2 (ウイルス) |
| 分子量 | 実験値: 1 MDa |
| 組換発現 | 生物種:  |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.07 mg/mL |
|---|---|
| 緩衝液 | pH: 7.5 / 詳細: 10mM HEPES, 150mM NaCl, 0.02% Glutaraldehyde |
| グリッド | 詳細: R2.2 Quantifoil |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 92 % / チャンバー内温度: 103.15 K / 装置: GATAN CRYOPLUNGE 3 / 詳細: Vitrification instrument: CP3 Gatan / 手法: Blot for 1 second before plunging |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | JEOL 2200FS |
|---|---|
| 温度 | 平均: 98.15 K |
| アライメント法 | Legacy - 非点収差: Objective lens astigmatism was corrected at 200,000 times magnification |
| 特殊光学系 | エネルギーフィルター - 名称: Omega エネルギーフィルター - エネルギー下限: 0.0 eV エネルギーフィルター - エネルギー上限: 20.0 eV |
| 撮影 | カテゴリ: FILM / フィルム・検出器のモデル: KODAK SO-163 FILM デジタル化 - スキャナー: NIKON SUPER COOLSCAN 9000 デジタル化 - サンプリング間隔: 10 µm / 実像数: 46 / 平均電子線量: 18 e/Å2 / ビット/ピクセル: 8 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 倍率(補正後): 45710 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.0 mm / 最大 デフォーカス(公称値): 2.6 µm / 最小 デフォーカス(公称値): 1.2 µm / 倍率(公称値): 50000 |
| 試料ステージ | 試料ホルダー: Eucentric / 試料ホルダーモデル: GATAN LIQUID NITROGEN |
- 画像解析
画像解析
| CTF補正 | 詳細: Each particle |
|---|---|
| 最終 再構成 | 想定した対称性 - 点群: C6 (6回回転対称) / 解像度のタイプ: BY AUTHOR / 解像度: 26.0 Å / 解像度の算出法: FSC 0.5 CUT-OFF / ソフトウェア - 名称: IMAGIC V |
| 最終 角度割当 | 詳細: beta 0 degrees, gamma 90 |
| 最終 2次元分類 | クラス数: 263 |
 ムービー
ムービー コントローラー
コントローラー












 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)





















