+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | In-situ structure of the hexameric HEF trimers from influenza C viral particles | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Class 1 fusion protein / membrane fusion / viral assembly / hexameric / VIRUS | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報sialate 9-O-acetylesterase activity / sialate 4-O-acetylesterase activity / sialate O-acetylesterase / viral budding from plasma membrane / endocytosis involved in viral entry into host cell / host cell surface receptor binding / fusion of virus membrane with host plasma membrane / fusion of virus membrane with host endosome membrane / viral envelope / virion attachment to host cell ...sialate 9-O-acetylesterase activity / sialate 4-O-acetylesterase activity / sialate O-acetylesterase / viral budding from plasma membrane / endocytosis involved in viral entry into host cell / host cell surface receptor binding / fusion of virus membrane with host plasma membrane / fusion of virus membrane with host endosome membrane / viral envelope / virion attachment to host cell / host cell plasma membrane / virion membrane / membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |  Influenza C virus (C/Johannesburg/1/66) (インフルエンザウイルス) Influenza C virus (C/Johannesburg/1/66) (インフルエンザウイルス) | |||||||||
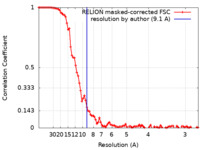
| 手法 | サブトモグラム平均法 / クライオ電子顕微鏡法 / 解像度: 9.1 Å | |||||||||
 データ登録者 データ登録者 | Liu ZB / Rosenthal PB | |||||||||
| 資金援助 |  英国, 1件 英国, 1件
| |||||||||
 引用 引用 |  ジャーナル: To Be Published ジャーナル: To Be Publishedタイトル: Topological defects in the spherical virus crystal 著者: Liu ZB / Rosenthal PB | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_17001.map.gz emd_17001.map.gz | 59.6 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-17001-v30.xml emd-17001-v30.xml emd-17001.xml emd-17001.xml | 17.7 KB 17.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_17001_fsc.xml emd_17001_fsc.xml | 9.2 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_17001.png emd_17001.png | 54.3 KB | ||
| マスクデータ |  emd_17001_msk_1.map emd_17001_msk_1.map | 64 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-17001.cif.gz emd-17001.cif.gz | 5.6 KB | ||
| その他 |  emd_17001_half_map_1.map.gz emd_17001_half_map_1.map.gz emd_17001_half_map_2.map.gz emd_17001_half_map_2.map.gz | 48.3 MB 48.3 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-17001 http://ftp.pdbj.org/pub/emdb/structures/EMD-17001 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-17001 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-17001 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_17001_validation.pdf.gz emd_17001_validation.pdf.gz | 932.1 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_17001_full_validation.pdf.gz emd_17001_full_validation.pdf.gz | 931.7 KB | 表示 | |
| XML形式データ |  emd_17001_validation.xml.gz emd_17001_validation.xml.gz | 16 KB | 表示 | |
| CIF形式データ |  emd_17001_validation.cif.gz emd_17001_validation.cif.gz | 21.3 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-17001 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-17001 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-17001 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-17001 | HTTPS FTP |
-関連構造データ
| 関連構造データ | C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_17001.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_17001.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 1.38 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_17001_msk_1.map emd_17001_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
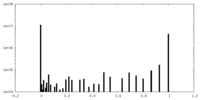
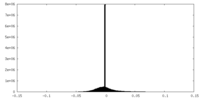
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_17001_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
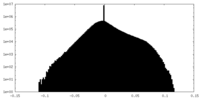
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_17001_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
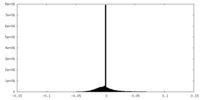
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Influenza C virus (C/Johannesburg/1/66)
| 全体 | 名称:  Influenza C virus (C/Johannesburg/1/66) (インフルエンザウイルス) Influenza C virus (C/Johannesburg/1/66) (インフルエンザウイルス) |
|---|---|
| 要素 |
|
-超分子 #1: Influenza C virus (C/Johannesburg/1/66)
| 超分子 | 名称: Influenza C virus (C/Johannesburg/1/66) / タイプ: virus / ID: 1 / 親要素: 0 / 含まれる分子: all / 詳細: Purified viruses / NCBI-ID: 100673 / 生物種: Influenza C virus (C/Johannesburg/1/66) / ウイルスタイプ: VIRION / ウイルス・単離状態: STRAIN / ウイルス・エンベロープ: Yes / ウイルス・中空状態: No |
|---|---|
| 分子量 | 理論値: 1.24 MDa |
-分子 #1: Haemagglutinin-esterase-fusion 1 glycoprotein
| 分子 | 名称: Haemagglutinin-esterase-fusion 1 glycoprotein / タイプ: protein_or_peptide / ID: 1 / 光学異性体: LEVO / EC番号: sialate O-acetylesterase |
|---|---|
| 由来(天然) | 生物種:  Influenza C virus (C/Johannesburg/1/66) (インフルエンザウイルス) Influenza C virus (C/Johannesburg/1/66) (インフルエンザウイルス) |
| 配列 | 文字列: EKIKICLQKQ VNSSFSLHNG FGGNLYATEE KRMFELVKPK AGASVLNQST WIGFGDSRTD KSNSAFPRSA DVSAKTADKF RFLSGGSLML SMFGPPGKVD YLYQGCGKHK VFYEGVNWSP HAAINCYRKN WTDIKLNFQK NIYELASQSH CMSLVNALDK TIPLQVTAGT ...文字列: EKIKICLQKQ VNSSFSLHNG FGGNLYATEE KRMFELVKPK AGASVLNQST WIGFGDSRTD KSNSAFPRSA DVSAKTADKF RFLSGGSLML SMFGPPGKVD YLYQGCGKHK VFYEGVNWSP HAAINCYRKN WTDIKLNFQK NIYELASQSH CMSLVNALDK TIPLQVTAGT AGNCNNSFLK NPALYTQEVK PSENKCGKEN LAFFTLPTQF GTYECKLHLV ASCYFIYDSK EVYNKRGCDN YFQVIYDSFG KVVGGLDNRV SPYTGNSGDT PTMQCDMLQL KPGRYSVRSS PRFLLMPERS YCFDMKEKGP VTAVQSIWGK GRESDYAVDQ ACLSTPGCML IQKQKPYIGE ADDHHGDQEM RELLSGLDYE ARCISQSGWV NETSPFTEKY LLPPKFGRCP LAAKEESIPK IPDGLLIPTS GTDTTVTKPK SR UniProtKB: Hemagglutinin-esterase-fusion glycoprotein |
-分子 #2: Haemagglutinin-esterase-fusion 2 glycoprotein
| 分子 | 名称: Haemagglutinin-esterase-fusion 2 glycoprotein / タイプ: protein_or_peptide / ID: 2 / 光学異性体: LEVO / EC番号: sialate O-acetylesterase |
|---|---|
| 由来(天然) | 生物種:  Influenza C virus (C/Johannesburg/1/66) (インフルエンザウイルス) Influenza C virus (C/Johannesburg/1/66) (インフルエンザウイルス) |
| 配列 | 文字列: IFGIDDLIIG VLFVAIVETG IGGYLLGSRK ESGGGVTKES AEKGFEKIGN DIQILKSSIN IAIEKLNDRI SHDEQAIRDL TLEIENARSE ALLGELGIIR ALLVGNISIG LQESLWELAS EITNRAGDLA VEVSPGCWII DNNICDQSCQ NFIFKFNETA PVPTIPPLDT KIDLQ UniProtKB: Hemagglutinin-esterase-fusion glycoprotein |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | サブトモグラム平均法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 2 mg/mL | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.4 構成要素:
| ||||||||||||
| グリッド | モデル: Quantifoil R2/2 / 材質: COPPER / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 30 sec. / 前処理 - 雰囲気: AMYLAMINE | ||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK III | ||||||||||||
| 詳細 | Heterogeneous mix of viral particles, viral like particles and cellular vesicles |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 特殊光学系 | エネルギーフィルター - スリット幅: 20 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / デジタル化 - サイズ - 横: 3838 pixel / デジタル化 - サイズ - 縦: 3710 pixel / 実像数: 1 / 平均露光時間: 1.1 sec. / 平均電子線量: 2.1 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 100.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 4.5 µm / 最小 デフォーカス(公称値): 2.0 µm / 倍率(公称値): 64000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
-原子モデル構築 1
| 初期モデル | PDB ID: Chain - Source name: PDB / Chain - Initial model type: experimental model |
|---|
 ムービー
ムービー コントローラー
コントローラー












 Z
Z Y
Y X
X