+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of the Fmoc-Tau-PAM4 Type 3 amyloid fibril | |||||||||
 マップデータ マップデータ | Final postprocessed, sharpened, helical symmetrised map of the Fmoc-TauPAM4 fibril morphology IIa | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | amyloid / tau / helical / cross-beta / fibril / neurodegeneration / Fmoc / PROTEIN FIBRIL | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報plus-end-directed organelle transport along microtubule / histone-dependent DNA binding / negative regulation of establishment of protein localization to mitochondrion / neurofibrillary tangle / microtubule lateral binding / axonal transport / tubulin complex / positive regulation of protein localization to synapse / negative regulation of tubulin deacetylation / phosphatidylinositol bisphosphate binding ...plus-end-directed organelle transport along microtubule / histone-dependent DNA binding / negative regulation of establishment of protein localization to mitochondrion / neurofibrillary tangle / microtubule lateral binding / axonal transport / tubulin complex / positive regulation of protein localization to synapse / negative regulation of tubulin deacetylation / phosphatidylinositol bisphosphate binding / generation of neurons / rRNA metabolic process / axonal transport of mitochondrion / regulation of mitochondrial fission / axon development / regulation of chromosome organization / central nervous system neuron development / intracellular distribution of mitochondria / minor groove of adenine-thymine-rich DNA binding / lipoprotein particle binding / microtubule polymerization / negative regulation of mitochondrial membrane potential / regulation of microtubule polymerization / dynactin binding / apolipoprotein binding / main axon / protein polymerization / axolemma / glial cell projection / Caspase-mediated cleavage of cytoskeletal proteins / regulation of microtubule polymerization or depolymerization / negative regulation of mitochondrial fission / neurofibrillary tangle assembly / positive regulation of axon extension / regulation of cellular response to heat / Activation of AMPK downstream of NMDARs / positive regulation of superoxide anion generation / synapse assembly / regulation of long-term synaptic depression / supramolecular fiber organization / positive regulation of protein localization / cellular response to brain-derived neurotrophic factor stimulus / regulation of calcium-mediated signaling / cytoplasmic microtubule organization / positive regulation of microtubule polymerization / somatodendritic compartment / axon cytoplasm / astrocyte activation / stress granule assembly / phosphatidylinositol binding / nuclear periphery / regulation of microtubule cytoskeleton organization / protein phosphatase 2A binding / cellular response to reactive oxygen species / Hsp90 protein binding / microglial cell activation / synapse organization / cellular response to nerve growth factor stimulus / protein homooligomerization / PKR-mediated signaling / regulation of synaptic plasticity / SH3 domain binding / response to lead ion / microtubule cytoskeleton organization / memory / cytoplasmic ribonucleoprotein granule / neuron projection development / cell-cell signaling / single-stranded DNA binding / protein-folding chaperone binding / cellular response to heat / actin binding / microtubule cytoskeleton / cell body / growth cone / double-stranded DNA binding / protein-macromolecule adaptor activity / microtubule binding / dendritic spine / sequence-specific DNA binding / amyloid fibril formation / microtubule / learning or memory / neuron projection / regulation of autophagy / membrane raft / axon / negative regulation of gene expression / neuronal cell body / dendrite / DNA damage response / protein kinase binding / enzyme binding / mitochondrion / DNA binding / RNA binding / extracellular region / identical protein binding / nucleus / plasma membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
| 手法 | らせん対称体再構成法 / クライオ電子顕微鏡法 / 解像度: 2.7 Å | |||||||||
 データ登録者 データ登録者 | Wilkinson M / Louros N / Tsaka G / Ramakers M / Morelli C / Garcia T / Gallardo RU / D'Haeyer S / Goossens V / Audenaert D ...Wilkinson M / Louros N / Tsaka G / Ramakers M / Morelli C / Garcia T / Gallardo RU / D'Haeyer S / Goossens V / Audenaert D / Thal DR / Ranson NA / Radford SE / Rousseau F / Schymkowitz J | |||||||||
| 資金援助 |  ベルギー, 1件 ベルギー, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2024 ジャーナル: Nat Commun / 年: 2024タイトル: Local structural preferences in shaping tau amyloid polymorphism. 著者: Nikolaos Louros / Martin Wilkinson / Grigoria Tsaka / Meine Ramakers / Chiara Morelli / Teresa Garcia / Rodrigo Gallardo / Sam D'Haeyer / Vera Goossens / Dominique Audenaert / Dietmar Rudolf ...著者: Nikolaos Louros / Martin Wilkinson / Grigoria Tsaka / Meine Ramakers / Chiara Morelli / Teresa Garcia / Rodrigo Gallardo / Sam D'Haeyer / Vera Goossens / Dominique Audenaert / Dietmar Rudolf Thal / Ian R Mackenzie / Rosa Rademakers / Neil A Ranson / Sheena E Radford / Frederic Rousseau / Joost Schymkowitz /    要旨: Tauopathies encompass a group of neurodegenerative disorders characterised by diverse tau amyloid fibril structures. The persistence of polymorphism across tauopathies suggests that distinct ...Tauopathies encompass a group of neurodegenerative disorders characterised by diverse tau amyloid fibril structures. The persistence of polymorphism across tauopathies suggests that distinct pathological conditions dictate the adopted polymorph for each disease. However, the extent to which intrinsic structural tendencies of tau amyloid cores contribute to fibril polymorphism remains uncertain. Using a combination of experimental approaches, we here identify a new amyloidogenic motif, PAM4 (Polymorphic Amyloid Motif of Repeat 4), as a significant contributor to tau polymorphism. Calculation of per-residue contributions to the stability of the fibril cores of different pathologic tau structures suggests that PAM4 plays a central role in preserving structural integrity across amyloid polymorphs. Consistent with this, cryo-EM structural analysis of fibrils formed from a synthetic PAM4 peptide shows that the sequence adopts alternative structures that closely correspond to distinct disease-associated tau strains. Furthermore, in-cell experiments revealed that PAM4 deletion hampers the cellular seeding efficiency of tau aggregates extracted from Alzheimer's disease, corticobasal degeneration, and progressive supranuclear palsy patients, underscoring PAM4's pivotal role in these tauopathies. Together, our results highlight the importance of the intrinsic structural propensity of amyloid core segments to determine the structure of tau in cells, and in propagating amyloid structures in disease. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_16883.map.gz emd_16883.map.gz | 20.8 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-16883-v30.xml emd-16883-v30.xml emd-16883.xml emd-16883.xml | 18.6 KB 18.6 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_16883_fsc.xml emd_16883_fsc.xml | 10.6 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_16883.png emd_16883.png | 78 KB | ||
| Filedesc metadata |  emd-16883.cif.gz emd-16883.cif.gz | 6 KB | ||
| その他 |  emd_16883_half_map_1.map.gz emd_16883_half_map_1.map.gz emd_16883_half_map_2.map.gz emd_16883_half_map_2.map.gz | 80.7 MB 80.7 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-16883 http://ftp.pdbj.org/pub/emdb/structures/EMD-16883 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-16883 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-16883 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_16883_validation.pdf.gz emd_16883_validation.pdf.gz | 818.1 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_16883_full_validation.pdf.gz emd_16883_full_validation.pdf.gz | 817.7 KB | 表示 | |
| XML形式データ |  emd_16883_validation.xml.gz emd_16883_validation.xml.gz | 18 KB | 表示 | |
| CIF形式データ |  emd_16883_validation.cif.gz emd_16883_validation.cif.gz | 23.7 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16883 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16883 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16883 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16883 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_16883.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_16883.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Final postprocessed, sharpened, helical symmetrised map of the Fmoc-TauPAM4 fibril morphology IIa | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.94 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: halfmap2
| ファイル | emd_16883_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | halfmap2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: halfmap1
| ファイル | emd_16883_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | halfmap1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Amyloid fibril form 2a of Tau-PAM4 peptide adducted with the Fmoc...
| 全体 | 名称: Amyloid fibril form 2a of Tau-PAM4 peptide adducted with the Fmoc protection group |
|---|---|
| 要素 |
|
-超分子 #1: Amyloid fibril form 2a of Tau-PAM4 peptide adducted with the Fmoc...
| 超分子 | 名称: Amyloid fibril form 2a of Tau-PAM4 peptide adducted with the Fmoc protection group タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all / 詳細: Synthesised peptide assembled into amyloid fibril |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
-分子 #1: Microtubule-associated protein tau
| 分子 | 名称: Microtubule-associated protein tau / タイプ: protein_or_peptide / ID: 1 詳細: 13-residue peptide of the PAM4 motif of Tau, corresponding to residues 350-362 of the Tau repeat domain. The peptide is C-terminally amidated and should have been N-terminally acetylated but ...詳細: 13-residue peptide of the PAM4 motif of Tau, corresponding to residues 350-362 of the Tau repeat domain. The peptide is C-terminally amidated and should have been N-terminally acetylated but 10% of the peptide (by mass spec) was still adducted to the Fmoc protection group from peptide synthesis. This contaminant species dominated fibril assembly コピー数: 24 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 1.652269 KDa |
| 配列 | 文字列: (VP1)VQSKIGSLD NITH(NH2) UniProtKB: Microtubule-associated protein tau |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | らせん対称体再構成法 |
| 試料の集合状態 | filament |
- 試料調製
試料調製
| 濃度 | 0.6 mg/mL |
|---|---|
| 緩衝液 | pH: 7 詳細: Peptide resuspended in MilliQ water for aggregation reaction |
| グリッド | モデル: EMS Lacey Carbon / 材質: COPPER / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: LACEY / 前処理 - タイプ: PLASMA CLEANING / 前処理 - 時間: 60 sec. |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 90 % / チャンバー内温度: 279 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 特殊光学系 | エネルギーフィルター - 名称: TFS Selectris / エネルギーフィルター - スリット幅: 10 eV |
| 撮影 | フィルム・検出器のモデル: FEI FALCON IV (4k x 4k) デジタル化 - サイズ - 横: 4096 pixel / デジタル化 - サイズ - 縦: 4096 pixel / 撮影したグリッド数: 1 / 実像数: 1957 / 平均露光時間: 5.0 sec. / 平均電子線量: 32.0 e/Å2 詳細: Movies were collected as 1204 EER frames compressed and re-grouped into 35 TIF fractions |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 50.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.4 µm / 最小 デフォーカス(公称値): 1.2 µm / 倍率(公称値): 130000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 詳細 | Initial model built manually in coot, no starting template |
|---|---|
| 精密化 | 空間: REAL / プロトコル: AB INITIO MODEL / 温度因子: 48 当てはまり具合の基準: Cross-correlation coefficient |
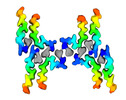
| 得られたモデル |  PDB-8ohp: |
 ムービー
ムービー コントローラー
コントローラー













 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)