+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Human apoferritin | |||||||||
 マップデータ マップデータ | Map of human apoferritin. | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Iron binding protein / METAL BINDING PROTEIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報iron ion sequestering activity / ferritin complex / Scavenging by Class A Receptors / negative regulation of ferroptosis / Golgi Associated Vesicle Biogenesis / ferroxidase / autolysosome / ferroxidase activity / negative regulation of fibroblast proliferation / ferric iron binding ...iron ion sequestering activity / ferritin complex / Scavenging by Class A Receptors / negative regulation of ferroptosis / Golgi Associated Vesicle Biogenesis / ferroxidase / autolysosome / ferroxidase activity / negative regulation of fibroblast proliferation / ferric iron binding / autophagosome / Iron uptake and transport / iron ion transport / ferrous iron binding / tertiary granule lumen / ficolin-1-rich granule lumen / intracellular iron ion homeostasis / immune response / iron ion binding / negative regulation of cell population proliferation / Neutrophil degranulation / extracellular exosome / extracellular region / identical protein binding / nucleus / cytoplasm / cytosol 類似検索 - 分子機能 | |||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
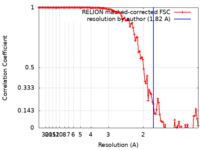
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 1.82 Å | |||||||||
 データ登録者 データ登録者 | Last MGF / Noteborn WEM / Sharp TH | |||||||||
| 資金援助 |  オランダ, 1件 オランダ, 1件
| |||||||||
 引用 引用 |  ジャーナル: J Struct Biol / 年: 2023 ジャーナル: J Struct Biol / 年: 2023タイトル: Super-resolution fluorescence imaging of cryosamples does not limit achievable resolution in cryoEM. 著者: Mart G F Last / Willem E M Noteborn / Lenard M Voortman / Thomas H Sharp /  要旨: Correlated super-resolution cryo-fluorescence and cryo-electron microscopy (cryoEM) has been gaining popularity as a method to investigate biological samples with high resolution and specificity. A ...Correlated super-resolution cryo-fluorescence and cryo-electron microscopy (cryoEM) has been gaining popularity as a method to investigate biological samples with high resolution and specificity. A concern in this combined method (called SR-cryoCLEM), however, is whether and how fluorescence imaging prior to cryoEM acquisition is detrimental to sample integrity. In this report, we investigated the effect of high-dose laser light (405, 488, and 561 nm) irradiation on apoferritin samples prepared for cryoEM with excitation wavelengths commonly used in fluorescence microscopy, and compared these samples to controls that were kept in the dark. We found that laser illumination, of equal duration and intensity as used in cryo-single molecule localization microscopy (cryoSMLM) and in the presence of high concentrations of fluorescent protein, did not affect the achievable resolution in cryoEM, with final reconstructions reaching resolutions of ∼ 1.8 Å regardless of the laser illumination. The finding that super-resolution fluorescence imaging of cryosamples prior to cryoEM data acquisition does not limit the achievable resolution suggests that super-resolution cryo-fluorescence microscopy and in situ structural biology using cryoEM are entirely compatible. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_16784.map.gz emd_16784.map.gz | 15.6 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-16784-v30.xml emd-16784-v30.xml emd-16784.xml emd-16784.xml | 16.7 KB 16.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_16784_fsc.xml emd_16784_fsc.xml | 10.6 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_16784.png emd_16784.png | 202.7 KB | ||
| Filedesc metadata |  emd-16784.cif.gz emd-16784.cif.gz | 5.9 KB | ||
| その他 |  emd_16784_half_map_1.map.gz emd_16784_half_map_1.map.gz emd_16784_half_map_2.map.gz emd_16784_half_map_2.map.gz | 76.4 MB 76.4 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-16784 http://ftp.pdbj.org/pub/emdb/structures/EMD-16784 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-16784 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-16784 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_16784_validation.pdf.gz emd_16784_validation.pdf.gz | 851.5 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_16784_full_validation.pdf.gz emd_16784_full_validation.pdf.gz | 851.1 KB | 表示 | |
| XML形式データ |  emd_16784_validation.xml.gz emd_16784_validation.xml.gz | 18 KB | 表示 | |
| CIF形式データ |  emd_16784_validation.cif.gz emd_16784_validation.cif.gz | 23.6 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16784 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16784 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16784 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16784 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8cpsMC  8cpmC  8cptC  8cpuC  8cpvC  8cpwC  8cpxC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_16784.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_16784.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Map of human apoferritin. | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.656 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: Half map 1
| ファイル | emd_16784_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map 1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half map 2
| ファイル | emd_16784_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map 2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Apoferritin
| 全体 | 名称: Apoferritin |
|---|---|
| 要素 |
|
-超分子 #1: Apoferritin
| 超分子 | 名称: Apoferritin / タイプ: organelle_or_cellular_component / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 484 KDa |
-分子 #1: Ferritin heavy chain, N-terminally processed
| 分子 | 名称: Ferritin heavy chain, N-terminally processed / タイプ: protein_or_peptide / ID: 1 / コピー数: 24 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 20.21857 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: STSQVRQNYH QDSEAAINRQ INLELYASYV YLSMSYYFDR DDVALKNFAK YFLHQSHEER EHAEKLMKLQ NQRGGRIFLQ DIQKPD(CSX)DD WESGLNAMEC ALHLEKNVNQ SLLELHKLAT DKNDPHLCDF IETHYLNEQV KAIKELGDHV TNLRKMG AP ESGLAEYLFD KHTLG UniProtKB: Ferritin heavy chain |
-分子 #2: SODIUM ION
| 分子 | 名称: SODIUM ION / タイプ: ligand / ID: 2 / コピー数: 24 |
|---|---|
| 分子量 | 理論値: 22.99 Da |
-分子 #3: MAGNESIUM ION
| 分子 | 名称: MAGNESIUM ION / タイプ: ligand / ID: 3 / コピー数: 14 / 式: MG |
|---|---|
| 分子量 | 理論値: 24.305 Da |
-分子 #4: water
| 分子 | 名称: water / タイプ: ligand / ID: 4 / コピー数: 1916 / 式: HOH |
|---|---|
| 分子量 | 理論値: 18.015 Da |
| Chemical component information |  ChemComp-HOH: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 4 mg/mL | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 8 構成要素:
| |||||||||
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: GOLD / メッシュ: 300 / 支持フィルム - 材質: GOLD / 支持フィルム - トポロジー: HOLEY / 支持フィルム - Film thickness: 50 / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 30 sec. | |||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 295 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | TFS KRIOS |
|---|---|
| 特殊光学系 | エネルギーフィルター - 名称: GIF Bioquantum / エネルギーフィルター - スリット幅: 20 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 平均電子線量: 50.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 50.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 1.5 µm / 最小 デフォーカス(公称値): 0.5 µm / 倍率(公称値): 130000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー














 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)