+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | In vitro structure of the Nitrosopumilus maritimus S-layer - Six-fold symmetry (C6) | |||||||||
 マップデータ マップデータ | PostProcessed map with B-factor sharpening | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Nmar_1547 S-layer / STRUCTURAL PROTEIN | |||||||||
| 機能・相同性 | membrane / Uncharacterized protein 機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |  Nitrosopumilus maritimus SCM1 (古細菌) Nitrosopumilus maritimus SCM1 (古細菌) | |||||||||
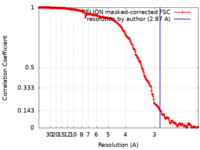
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.87 Å | |||||||||
 データ登録者 データ登録者 | von Kuegelgen A / Bharat T | |||||||||
| 資金援助 |  英国, 2件 英国, 2件
| |||||||||
 引用 引用 |  ジャーナル: Nature / 年: 2024 ジャーナル: Nature / 年: 2024タイトル: Membraneless channels sieve cations in ammonia-oxidizing marine archaea. 著者: Andriko von Kügelgen / C Keith Cassidy / Sofie van Dorst / Lennart L Pagani / Christopher Batters / Zephyr Ford / Jan Löwe / Vikram Alva / Phillip J Stansfeld / Tanmay A M Bharat /    要旨: Nitrosopumilus maritimus is an ammonia-oxidizing archaeon that is crucial to the global nitrogen cycle. A critical step for nitrogen oxidation is the entrapment of ammonium ions from a dilute marine ...Nitrosopumilus maritimus is an ammonia-oxidizing archaeon that is crucial to the global nitrogen cycle. A critical step for nitrogen oxidation is the entrapment of ammonium ions from a dilute marine environment at the cell surface and their subsequent channelling to the cell membrane of N. maritimus. Here we elucidate the structure of the molecular machinery responsible for this process, comprising the surface layer (S-layer), using electron cryotomography and subtomogram averaging from cells. We supplemented our in situ structure of the ammonium-binding S-layer array with a single-particle electron cryomicroscopy structure, revealing detailed features of this immunoglobulin-rich and glycan-decorated S-layer. Biochemical analyses showed strong ammonium binding by the cell surface, which was lost after S-layer disassembly. Sensitive bioinformatic analyses identified similar S-layers in many ammonia-oxidizing archaea, with conserved sequence and structural characteristics. Moreover, molecular simulations and structure determination of ammonium-enriched specimens enabled us to examine the cation-binding properties of the S-layer, revealing how it concentrates ammonium ions on its cell-facing side, effectively acting as a multichannel sieve on the cell membrane. This in situ structural study illuminates the biogeochemically essential process of ammonium binding and channelling, common to many marine microorganisms that are fundamental to the nitrogen cycle. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_16482.map.gz emd_16482.map.gz | 113.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-16482-v30.xml emd-16482-v30.xml emd-16482.xml emd-16482.xml | 26.8 KB 26.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_16482_fsc.xml emd_16482_fsc.xml | 22.4 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_16482.png emd_16482.png | 159.4 KB | ||
| マスクデータ |  emd_16482_msk_1.map emd_16482_msk_1.map | 125 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-16482.cif.gz emd-16482.cif.gz | 8.6 KB | ||
| その他 |  emd_16482_additional_1.map.gz emd_16482_additional_1.map.gz emd_16482_half_map_1.map.gz emd_16482_half_map_1.map.gz emd_16482_half_map_2.map.gz emd_16482_half_map_2.map.gz | 111.3 MB 113.2 MB 113.2 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-16482 http://ftp.pdbj.org/pub/emdb/structures/EMD-16482 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-16482 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-16482 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_16482.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_16482.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | PostProcessed map with B-factor sharpening | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.092 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_16482_msk_1.map emd_16482_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
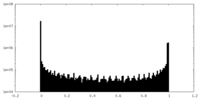
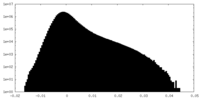
| 密度ヒストグラム |
-追加マップ: Refine3D main map without B-factor sharpening
| ファイル | emd_16482_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Refine3D main map without B-factor sharpening | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
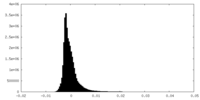
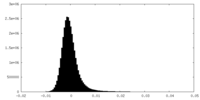
| 密度ヒストグラム |
-ハーフマップ: Half map 1
| ファイル | emd_16482_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map 1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
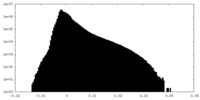
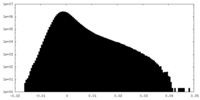
| 密度ヒストグラム |
-ハーフマップ: Half map 2
| ファイル | emd_16482_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map 2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
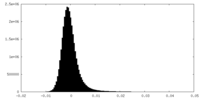
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Nitrosopumilus maritimus S-layer
| 全体 | 名称: Nitrosopumilus maritimus S-layer |
|---|---|
| 要素 |
|
-超分子 #1: Nitrosopumilus maritimus S-layer
| 超分子 | 名称: Nitrosopumilus maritimus S-layer / タイプ: organelle_or_cellular_component / ID: 1 / 親要素: 0 / 含まれる分子: all / 詳細: Nitrosopumilus maritimus S-layer C6 symmetrised |
|---|---|
| 由来(天然) | 生物種:  Nitrosopumilus maritimus SCM1 (古細菌) / 株: SCM1 / 細胞中の位置: extracellular Nitrosopumilus maritimus SCM1 (古細菌) / 株: SCM1 / 細胞中の位置: extracellular |
-分子 #1: Cell surface protein
| 分子 | 名称: Cell surface protein / タイプ: protein_or_peptide / ID: 1 / 詳細: In-vitro isolated S-layer / コピー数: 6 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Nitrosopumilus maritimus SCM1 (古細菌) / 株: SCM1 Nitrosopumilus maritimus SCM1 (古細菌) / 株: SCM1 |
| 分子量 | 理論値: 183.156 KDa |
| 配列 | 文字列: MNNEIGRKIT SLTLMTIMVA GGLTFAIPGV MPEAMAANAN LFVSAENSQF DNYMSGPQVI EVVVIDSDIN DTDEAKGEPD VTVNGKVLR MVQAVDGNWY GYFADRDQAQ IADSTATTAD SGLDFGVFCA SSSGTAALGF STTETDGIAI PITIANATAT G NGTQTGSS ...文字列: MNNEIGRKIT SLTLMTIMVA GGLTFAIPGV MPEAMAANAN LFVSAENSQF DNYMSGPQVI EVVVIDSDIN DTDEAKGEPD VTVNGKVLR MVQAVDGNWY GYFADRDQAQ IADSTATTAD SGLDFGVFCA SSSGTAALGF STTETDGIAI PITIANATAT G NGTQTGSS SGGAITTTCA ANTLDASTAN GTINVVREAK DPVAASGSVS VGQIGLKNGT ANSGPNWPFI QLYELNPTGN VV VQYNKGG GVQSTTLTFD TVDQFAELSL DRTVFPRVSQ VHATITDLWL NIDPTDEDSW TFATNTKNTT SSFNVDTFYQ VFD ENGASG GSALTLRTTL SSLMCEDNCV LTLDVDAQSS GTPVVTIQDN GDSILTQLNA SSNTNANNAS AFGISTETAK LGTG SIPVT ITEQGPNSGV FGTYDESDKS VLKITDNAKR GTSASLDYNE TPQTILVGFS FASIDIQPVT DEWTSGQEIP VVIVD ADQN KNSRADEDLD LNNPDVTLIP ALRTGDPFTI DEGGTPSLIF TNGTNGDDSI FDTGAINNTS AGQVGNFTLN INVTRF SSA TNITSTESID TFSKRLISAQ TANSSANFDV DFAIIDLGSA TLETLKETVV DEDNTAVGFN FFNYDVRSLG ADTVSIA LL NTTGNILPWV NNDTRNVDKN NAILLVSNST NSQAYVDLTN AVSDAVYGST NTDSNVNIGF AMYFTGVGDL AAKEVIVM D FFSFGFTDDG VQSSERFANQ IIRIEAEETG DNTSTFEGSL EYVMVNQINI QDAGTFSGIT PIADDPSFIV IEDLTDEDA PRVNYNDLGA DGVTTPVSDQ EEAPSHSGVV SLNADSYKIA DTVVITVEDL DLNVDSDLID IFTVVSDNSK ATDDAVGSAT TQSLSFGEL GRLLDVTFDD VIWSTPDGAN NTATGNDSDT CSTELSNAGI TDTGLGATGF TLVETGAATG VFVGDFQIPS F WCRVSDTT TTPYTYAGDE ETTTGLDIEV NYVDFRDASG EIVEVGDSAG VRANTGSVSL DRTVYPVPFG TIADSSKAAN AA PNGRSVF PIHATGITST IDSTEELPTG DLTIHVRIND PDFDENPAGE DAMDQDNALK ISVIRGSDSV VLGYAGASER TGK IDVGGN NGTISNIRSF GEMDEIAPDA GIFELDVNIK FTDGPASAQC NSHDTLYTAL DGTTGKADTN RFDDGAASGQ EYCI LQGDI LQVEYTDPAD ASGDANTVTD SATFDLRNGV LQSDKSVYII GSDMILTLIE PDFDLDNDSA ETYDLDLIEW DSDAA TTTM GNKGVTGAAA AFDPEPTDFR ETGDSTGIFQ IVIEIPESLS NDKLERGEEI ILEYTDWGPS GSDYVGDEDE DVNLTI YTS NFGATVELDQ KVYSWTDKVY ITIVAPDHNF DSDLVDEIGE TDSDPIKVST RGFDLDNYKL VETGTDTGIF TGEVILT GF TAHDADGDGN TGDATGTTSG SGPTDGLLAT DDDDGLTVSF EFSEDETIVG SALIRWNIGE VQWLEASYPA SGTGVVRV I DPDMNLDPEA VDNFEVDVWS DSDAGGIDLT VTETNEATGI FEGTVFFTTL DESSGHRLRV SEGDTVTAEY EDNTLPDPY TTADELDITA TSLIGTVVPP LERAPAANLR TVDAFGNSLD SVSVDQQVQI SADLANGQDR EQSFAYLVQI QDANGVTVSL AWITGSLSS GQSFSPALSW IPTEAGTYTA TAFVWESVDN PTALSPPVST TVNVS UniProtKB: Uncharacterized protein |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | 2D array |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 構成要素:
詳細: 50 mM HEPES/NaOH pH=7.5, 500 mM NaCl, 50 mM MgCl2, 10 mM CaCl2 | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| グリッド | モデル: Quantifoil R2/2 / 材質: COPPER/RHODIUM / メッシュ: 200 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY ARRAY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 20 sec. / 前処理 - 雰囲気: AIR / 詳細: 15 mA | |||||||||||||||
| 凍結 | 凍結剤: NITROGEN / チャンバー内湿度: 100 % / チャンバー内温度: 283.15 K / 装置: FEI VITROBOT MARK IV 詳細: absorption for 60 sec and blotted for 5 sec with blot force -10. | |||||||||||||||
| 詳細 | In vitro isolate S-layer cell envelopes |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 温度 | 最低: 70.0 K / 最高: 70.0 K |
| 特殊光学系 | 球面収差補正装置: not used / 色収差補正装置: not used / エネルギーフィルター - 名称: GIF Quantum LS / エネルギーフィルター - スリット幅: 20 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) デジタル化 - サイズ - 横: 5760 pixel / デジタル化 - サイズ - 縦: 4092 pixel / 撮影したグリッド数: 3 / 実像数: 12557 / 平均露光時間: 4.2 sec. / 平均電子線量: 48.5 e/Å2 詳細: collected over three sessions, two with no stage tilt and one session with stage tilted by 33 degrees (alpha tilt) |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 70.0 µm / 最大 デフォーカス(補正後): 5.0 µm / 最小 デフォーカス(補正後): 2.0 µm / 倍率(補正後): 81000 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 5.0 µm / 最小 デフォーカス(公称値): 2.0 µm / 倍率(公称値): 81000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: RECIPROCAL / プロトコル: AB INITIO MODEL / 温度因子: 59.2 / 当てはまり具合の基準: Best Fit |
|---|---|
| 得られたモデル |  PDB-8c8k: |
 ムービー
ムービー コントローラー
コントローラー















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)