+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of the yeast SPT-Orm1-Monomer complex | |||||||||
 マップデータ マップデータ | yeast-SPOTS-Orm1-Monomer | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Serine-Palmitoyl-Transferase / SPT / Orm-Protein / TRANSFERASE | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報negative regulation of sphingolipid biosynthetic process / positive regulation of sphingolipid biosynthetic process / 3-keto-sphinganine metabolic process / serine palmitoyltransferase complex / intracellular sphingolipid homeostasis / serine C-palmitoyltransferase activity / serine C-palmitoyltransferase / ceramide metabolic process / sphingosine biosynthetic process / sphingolipid biosynthetic process ...negative regulation of sphingolipid biosynthetic process / positive regulation of sphingolipid biosynthetic process / 3-keto-sphinganine metabolic process / serine palmitoyltransferase complex / intracellular sphingolipid homeostasis / serine C-palmitoyltransferase activity / serine C-palmitoyltransferase / ceramide metabolic process / sphingosine biosynthetic process / sphingolipid biosynthetic process / ceramide biosynthetic process / response to unfolded protein / Neutrophil degranulation / enzyme activator activity / pyridoxal phosphate binding / endoplasmic reticulum membrane / endoplasmic reticulum / membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |  | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.4 Å | |||||||||
 データ登録者 データ登録者 | Schaefer J / Koerner C / Parey K / Januliene D / Moeller A / Froehlich F | |||||||||
| 資金援助 |  ドイツ, 2件 ドイツ, 2件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2023 ジャーナル: Nat Commun / 年: 2023タイトル: Structure of the ceramide-bound SPOTS complex. 著者: Jan-Hannes Schäfer / Carolin Körner / Bianca M Esch / Sergej Limar / Kristian Parey / Stefan Walter / Dovile Januliene / Arne Moeller / Florian Fröhlich /  要旨: Sphingolipids are structural membrane components that also function in cellular stress responses. The serine palmitoyltransferase (SPT) catalyzes the rate-limiting step in sphingolipid biogenesis. ...Sphingolipids are structural membrane components that also function in cellular stress responses. The serine palmitoyltransferase (SPT) catalyzes the rate-limiting step in sphingolipid biogenesis. Its activity is tightly regulated through multiple binding partners, including Tsc3, Orm proteins, ceramides, and the phosphatidylinositol-4-phosphate (PI4P) phosphatase Sac1. The structural organization and regulatory mechanisms of this complex are not yet understood. Here, we report the high-resolution cryo-EM structures of the yeast SPT in complex with Tsc3 and Orm1 (SPOT) as dimers and monomers and a monomeric complex further carrying Sac1 (SPOTS). In all complexes, the tight interaction of the downstream metabolite ceramide and Orm1 reveals the ceramide-dependent inhibition. Additionally, observation of ceramide and ergosterol binding suggests a co-regulation of sphingolipid biogenesis and sterol metabolism within the SPOTS complex. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_16467.map.gz emd_16467.map.gz | 290.4 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-16467-v30.xml emd-16467-v30.xml emd-16467.xml emd-16467.xml | 21.8 KB 21.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
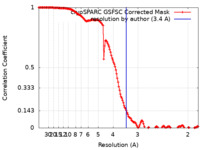
| FSC (解像度算出) |  emd_16467_fsc.xml emd_16467_fsc.xml | 14.3 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_16467.png emd_16467.png | 53.1 KB | ||
| Filedesc metadata |  emd-16467.cif.gz emd-16467.cif.gz | 7 KB | ||
| その他 |  emd_16467_half_map_1.map.gz emd_16467_half_map_1.map.gz emd_16467_half_map_2.map.gz emd_16467_half_map_2.map.gz | 285.7 MB 285.7 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-16467 http://ftp.pdbj.org/pub/emdb/structures/EMD-16467 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-16467 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-16467 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_16467_validation.pdf.gz emd_16467_validation.pdf.gz | 1.2 MB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_16467_full_validation.pdf.gz emd_16467_full_validation.pdf.gz | 1.2 MB | 表示 | |
| XML形式データ |  emd_16467_validation.xml.gz emd_16467_validation.xml.gz | 22.5 KB | 表示 | |
| CIF形式データ |  emd_16467_validation.cif.gz emd_16467_validation.cif.gz | 28.3 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16467 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16467 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16467 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16467 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8c80MC  8c81C  8c82C M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_16467.map.gz / 形式: CCP4 / 大きさ: 307.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_16467.map.gz / 形式: CCP4 / 大きさ: 307.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | yeast-SPOTS-Orm1-Monomer | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.924 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: halfmap-A yeast-SPOTS-Orm1-Monomer
| ファイル | emd_16467_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | halfmap-A yeast-SPOTS-Orm1-Monomer | ||||||||||||
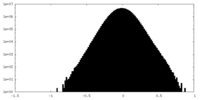
| 投影像・断面図 |
| ||||||||||||
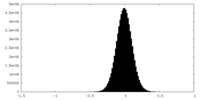
| 密度ヒストグラム |
-ハーフマップ: halfmap-B yeast-SPOTS-Orm1-Monomer
| ファイル | emd_16467_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | halfmap-B yeast-SPOTS-Orm1-Monomer | ||||||||||||
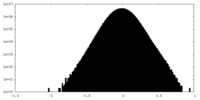
| 投影像・断面図 |
| ||||||||||||
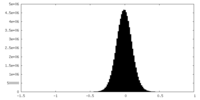
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Heterotetrameric complex of Orm1 with Lcb1, Lcb2 and Tsc3
| 全体 | 名称: Heterotetrameric complex of Orm1 with Lcb1, Lcb2 and Tsc3 |
|---|---|
| 要素 |
|
-超分子 #1: Heterotetrameric complex of Orm1 with Lcb1, Lcb2 and Tsc3
| 超分子 | 名称: Heterotetrameric complex of Orm1 with Lcb1, Lcb2 and Tsc3 タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#4 |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 160 KDa |
-分子 #1: Protein ORM1
| 分子 | 名称: Protein ORM1 / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 25.221674 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MTELDYQGTA EAASTSYSRN QTDLKPFPSA GSASSSIKTT EPVKDHRRRR AAAIISHVEP ETFEDENDQQ LLPNMNATWV DQRGAWIIH VVIIILLKLF YNLFPGVTTE WSWTLTNMTY VIGSYVMFHL IKGTPFDFNG GAYDNLTMWE QIDDETLYTP S RKFLISVP ...文字列: MTELDYQGTA EAASTSYSRN QTDLKPFPSA GSASSSIKTT EPVKDHRRRR AAAIISHVEP ETFEDENDQQ LLPNMNATWV DQRGAWIIH VVIIILLKLF YNLFPGVTTE WSWTLTNMTY VIGSYVMFHL IKGTPFDFNG GAYDNLTMWE QIDDETLYTP S RKFLISVP IALFLVSTHY AHYDLKLFSW NCFLTTFGAV VPKLPVTHRL RISIPGITGR AQIS UniProtKB: Protein ORM1 |
-分子 #2: Serine palmitoyltransferase 1
| 分子 | 名称: Serine palmitoyltransferase 1 / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO / EC番号: serine C-palmitoyltransferase |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 64.98552 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MAHIPEVLPD YKDHDGDYKD HDIDYKDDDD KKSIPIPAFI VTTSSYLWYY FNLVLTQIPG GQFIVSYIKK SHHDDPYRTT VEIGLILYG IIYYLSKPQQ KKSLQAQKPN LSPQEIDALI EDWEPEPLVD PSATDEQSWR VAKTPVTMEM PIQNHITITR N NLQEKYTN ...文字列: MAHIPEVLPD YKDHDGDYKD HDIDYKDDDD KKSIPIPAFI VTTSSYLWYY FNLVLTQIPG GQFIVSYIKK SHHDDPYRTT VEIGLILYG IIYYLSKPQQ KKSLQAQKPN LSPQEIDALI EDWEPEPLVD PSATDEQSWR VAKTPVTMEM PIQNHITITR N NLQEKYTN VFNLASNNFL QLSATEPVKE VVKTTIKNYG VGACGPAGFY GNQDVHYTLE YDLAQFFGTQ GSVLYGQDFC AA PSVLPAF TKRGDVIVAD DQVSLPVQNA LQLSRSTVYY FNHNDMNSLE CLLNELTEQE KLEKLPAIPR KFIVTEGIFH NSG DLAPLP ELTKLKNKYK FRLFVDETFS IGVLGATGRG LSEHFNMDRA TAIDITVGSM ATALGSTGGF VLGDSVMCLH QRIG SNAYC FSACLPAYTV TSVSKVLKLM DSNNDAVQTL QKLSKSLHDS FASDDSLRSY VIVTSSPVSA VLHLQLTPAY RSRKF GYTC EQLFETMSAL QKKSQTNKFI EPYEEEEKFL QSIVDHALIN YNVLITRNTI VLKQETLPIV PSLKICCNAA MSPEEL KNA CESVKQSILA CCQESNK UniProtKB: Serine palmitoyltransferase 1 |
-分子 #3: Serine palmitoyltransferase 2
| 分子 | 名称: Serine palmitoyltransferase 2 / タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO / EC番号: serine C-palmitoyltransferase |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 63.189707 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MSTPANYTRV PLCEPEELPD DIQKENEYGT LDSPGHLYQV KSRHGKPLPE PVVDTPPYYI SLLTYLNYLI LIILGHVHDF LGMTFQKNK HLDLLEHDGL APWFSNFESF YVRRIKMRID DCFSRPTTGV PGRFIRCIDR ISHNINEYFT YSGAVYPCMN L SSYNYLGF ...文字列: MSTPANYTRV PLCEPEELPD DIQKENEYGT LDSPGHLYQV KSRHGKPLPE PVVDTPPYYI SLLTYLNYLI LIILGHVHDF LGMTFQKNK HLDLLEHDGL APWFSNFESF YVRRIKMRID DCFSRPTTGV PGRFIRCIDR ISHNINEYFT YSGAVYPCMN L SSYNYLGF AQSKGQCTDA ALESVDKYSI QSGGPRAQIG TTDLHIKAEK LVARFIGKED ALVFSMGYGT NANLFNAFLD KK CLVISDE LNHTSIRTGV RLSGAAVRTF KHGDMVGLEK LIREQIVLGQ PKTNRPWKKI LICAEGLFSM EGTLCNLPKL VEL KKKYKC YLFIDEAHSI GAMGPTGRGV CEIFGVDPKD VDILMGTFTK SFGAAGGYIA ADQWIIDRLR LDLTTVSYSE SMPA PVLAQ TISSLQTISG EICPGQGTER LQRIAFNSRY LRLALQRLGF IVYGVADSPV IPLLLYCPSK MPAFSRMMLQ RRIAV VVVA YPATPLIESR VRFCMSASLT KEDIDYLLRH VSEVGDKLNL KSNSGKSSYD GKRQRWDIEE VIRRTPEDCK DDKYFV N UniProtKB: Serine palmitoyltransferase 2 |
-分子 #4: Serine palmitoyltransferase-regulating protein TSC3
| 分子 | 名称: Serine palmitoyltransferase-regulating protein TSC3 / タイプ: protein_or_peptide / ID: 4 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 9.590233 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MTQHKSSMVY IPTTKEAKRR NGKSEGILNT IEEVVEKLYW TYYIHLPFYL MASFDSFFLH VFFLTIFSLS FFGILKYCFL UniProtKB: Serine palmitoyltransferase-regulating protein TSC3 |
-分子 #5: ERGOSTEROL
| 分子 | 名称: ERGOSTEROL / タイプ: ligand / ID: 5 / コピー数: 3 / 式: ERG |
|---|---|
| 分子量 | 理論値: 396.648 Da |
| Chemical component information |  ChemComp-ERG: |
-分子 #6: N-[(2S,3S,4R)-1,3,4-trihydroxyoctadecan-2-yl]hexacosanamide
| 分子 | 名称: N-[(2S,3S,4R)-1,3,4-trihydroxyoctadecan-2-yl]hexacosanamide タイプ: ligand / ID: 6 / コピー数: 1 / 式: Z8A |
|---|---|
| 分子量 | 理論値: 696.182 Da |
| Chemical component information |  ChemComp-Z8A: |
-分子 #7: PYRIDOXAL-5'-PHOSPHATE
| 分子 | 名称: PYRIDOXAL-5'-PHOSPHATE / タイプ: ligand / ID: 7 / コピー数: 1 / 式: PLP |
|---|---|
| 分子量 | 理論値: 247.142 Da |
| Chemical component information |  ChemComp-PLP: |
-分子 #8: 2-{[(4-O-alpha-D-glucopyranosyl-alpha-D-glucopyranosyl)oxy]methyl...
| 分子 | 名称: 2-{[(4-O-alpha-D-glucopyranosyl-alpha-D-glucopyranosyl)oxy]methyl}-4-{[(3beta,9beta,14beta,17beta,25R)-spirost-5-en-3-yl]oxy}butyl 4-O-alpha-D-glucopyranosyl-alpha-D-glucopyranoside タイプ: ligand / ID: 8 / コピー数: 1 / 式: Q7G |
|---|---|
| 分子量 | 理論値: 1.165315 KDa |
| Chemical component information |  ChemComp-Q7G: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 10.0 mg/mL |
|---|---|
| 緩衝液 | pH: 6.8 |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277.15 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | TFS GLACIOS |
|---|---|
| 特殊光学系 | エネルギーフィルター - 名称: TFS Selectris / エネルギーフィルター - スリット幅: 10 eV |
| 撮影 | フィルム・検出器のモデル: FEI FALCON IV (4k x 4k) 実像数: 13604 / 平均電子線量: 50.0 e/Å2 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.0 µm / 最小 デフォーカス(公称値): 0.8 µm / 倍率(公称値): 130000 |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: AB INITIO MODEL / 温度因子: 79 |
|---|---|
| 得られたモデル |  PDB-8c80: |
 ムービー
ムービー コントローラー
コントローラー

















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)