+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Pontibacter korlensis curli subunit CsgA | ||||||||||||
 マップデータ マップデータ | |||||||||||||
 試料 試料 |
| ||||||||||||
 キーワード キーワード | bacterial functional amyloid / PROTEIN FIBRIL | ||||||||||||
| 機能・相同性 | Curlin associated / Curlin associated repeat / pilus / cell adhesion / Curlin associated repeat-containing protein 機能・相同性情報 機能・相同性情報 | ||||||||||||
| 生物種 |  Pontibacter korlensis (バクテリア) Pontibacter korlensis (バクテリア) | ||||||||||||
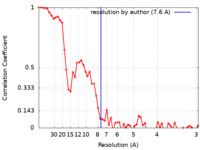
| 手法 | らせん対称体再構成法 / クライオ電子顕微鏡法 / 解像度: 7.6 Å | ||||||||||||
 データ登録者 データ登録者 | Remaut H / Sleutel M / Pradhan B | ||||||||||||
| 資金援助 |  ベルギー, European Union, 3件 ベルギー, European Union, 3件
| ||||||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2023 ジャーナル: Nat Commun / 年: 2023タイトル: Structural analysis and architectural principles of the bacterial amyloid curli. 著者: Mike Sleutel / Brajabandhu Pradhan / Alexander N Volkov / Han Remaut /  要旨: Two decades have passed since the initial proposition that amyloids are not only (toxic) byproducts of an unintended aggregation cascade, but that they can also be produced by an organism to serve a ...Two decades have passed since the initial proposition that amyloids are not only (toxic) byproducts of an unintended aggregation cascade, but that they can also be produced by an organism to serve a defined biological function. That revolutionary idea was borne out of the realization that a large fraction of the extracellular matrix that holds Gram-negative cells into a persistent biofilm is composed of protein fibers (curli; tafi) with cross-β architecture, nucleation-dependent polymerization kinetics and classic amyloid tinctorial properties. The list of proteins shown to form so-called functional amyloid fibers in vivo has greatly expanded over the years, but detailed structural insights have not followed at a similar pace in part due to the associated experimental barriers. Here we combine extensive AlphaFold2 modelling and cryo-electron transmission microscopy to propose an atomic model of curli protofibrils, and their higher modes of organization. We uncover an unexpected structural diversity of curli building blocks and fibril architectures. Our results allow for a rationalization of the extreme physico-chemical robustness of curli, as well as earlier observations of inter-species curli promiscuity, and should facilitate further engineering efforts to expand the repertoire of curli-based functional materials. | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_16431.map.gz emd_16431.map.gz | 9.8 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-16431-v30.xml emd-16431-v30.xml emd-16431.xml emd-16431.xml | 15.9 KB 15.9 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_16431_fsc.xml emd_16431_fsc.xml | 7 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_16431.png emd_16431.png | 42 KB | ||
| Filedesc metadata |  emd-16431.cif.gz emd-16431.cif.gz | 5.7 KB | ||
| その他 |  emd_16431_half_map_1.map.gz emd_16431_half_map_1.map.gz emd_16431_half_map_2.map.gz emd_16431_half_map_2.map.gz | 9.8 MB 9.8 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-16431 http://ftp.pdbj.org/pub/emdb/structures/EMD-16431 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-16431 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-16431 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_16431_validation.pdf.gz emd_16431_validation.pdf.gz | 916 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_16431_full_validation.pdf.gz emd_16431_full_validation.pdf.gz | 915.5 KB | 表示 | |
| XML形式データ |  emd_16431_validation.xml.gz emd_16431_validation.xml.gz | 10.7 KB | 表示 | |
| CIF形式データ |  emd_16431_validation.cif.gz emd_16431_validation.cif.gz | 14.7 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16431 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16431 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16431 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16431 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8c50MC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_16431.map.gz / 形式: CCP4 / 大きさ: 12.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_16431.map.gz / 形式: CCP4 / 大きさ: 12.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.485 Å | ||||||||||||||||||||||||||||||||||||
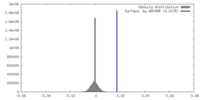
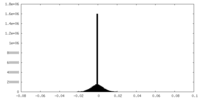
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #2
| ファイル | emd_16431_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
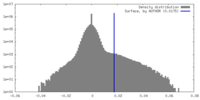
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_16431_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
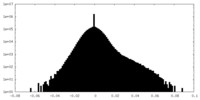
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : curli fiber of Pontibacteri korlensis CsgA
| 全体 | 名称: curli fiber of Pontibacteri korlensis CsgA |
|---|---|
| 要素 |
|
-超分子 #1: curli fiber of Pontibacteri korlensis CsgA
| 超分子 | 名称: curli fiber of Pontibacteri korlensis CsgA / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all 詳細: Sample is generated by in vitro polymerisation of Pontibacter korlensis Csga subunits |
|---|---|
| 由来(天然) | 生物種:  Pontibacter korlensis (バクテリア) Pontibacter korlensis (バクテリア) |
-分子 #1: Curlin associated repeat-containing protein
| 分子 | 名称: Curlin associated repeat-containing protein / タイプ: protein_or_peptide / ID: 1 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Pontibacter korlensis (バクテリア) Pontibacter korlensis (バクテリア) |
| 分子量 | 理論値: 38.033785 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: QGTTSSATTS QTGNTNTAVI DQVGGLNNSA EASQAGDGNV ATVTQAEGMD NAVYIDQVGL TNTATVLQEG GLDNDADVDQ DGDLNVAYI WQNLGEDNDA DIDQDGTLND AAIEQDGGED NDADIDQDGS ENAAYVGQTG GEDNDADIDQ DGTFNNAYIG Q FGGEDNEA ...文字列: QGTTSSATTS QTGNTNTAVI DQVGGLNNSA EASQAGDGNV ATVTQAEGMD NAVYIDQVGL TNTATVLQEG GLDNDADVDQ DGDLNVAYI WQNLGEDNDA DIDQDGTLND AAIEQDGGED NDADIDQDGS ENAAYVGQTG GEDNDADIDQ DGTFNNAYIG Q FGGEDNEA DLDQDGDANY AAILQDGGED NDADIDQDGT NNWAETNQIQ GNDNDVEVDQ DGSDNIAEVW QMHGEDNEAN VD QDGDLNN AYILQEGGMN NLADVHQNGV SNTAEIYQYG GMDNEAYLIQ DGDMHTGVIT QSGDGANYAE LNQMGLMNTG SIT QDGMGN SAITSQGGSM NMSTVTQSGT GNISLVNQHG GHHHHHH UniProtKB: Curlin associated repeat-containing protein |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | らせん対称体再構成法 |
| 試料の集合状態 | filament |
- 試料調製
試料調製
| 緩衝液 | pH: 6 / 詳細: 15 mM MES pH 6.0 |
|---|---|
| グリッド | モデル: Quantifoil R2/1 / 材質: COPPER / メッシュ: 300 / 支持フィルム - 材質: GRAPHENE OXIDE / 前処理 - タイプ: GLOW DISCHARGE |
| 凍結 | 凍結剤: ETHANE / 装置: GATAN CRYOPLUNGE 3 |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | JEOL CRYO ARM 300 |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 撮影したグリッド数: 1 / 実像数: 4455 / 平均電子線量: 64.66 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: OTHER / 撮影モード: OTHER / 最大 デフォーカス(公称値): 3.5 µm / 最小 デフォーカス(公称値): 0.8 µm / 倍率(公称値): 60000 |
| 試料ステージ | 試料ホルダーモデル: JEOL CRYOSPECPORTER / ホルダー冷却材: NITROGEN |
 ムービー
ムービー コントローラー
コントローラー




 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)