+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of the peroxisomal Pex1/Pex6 ATPase complex bound to a substrate in twin seam state | |||||||||||||||
 マップデータ マップデータ | Map generated with SPHIRE Postrefine | |||||||||||||||
 試料 試料 |
| |||||||||||||||
 キーワード キーワード | AAA ATPase / peroxisomes / peroxisome biogenesis / peroxisome biogenesis disorders / Zellweger Syndrome / TRANSLOCASE | |||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報protein import into peroxisome matrix, receptor recycling / protein import into peroxisome matrix / transporter complex / protein transporter activity / peroxisomal membrane / ATPase complex / protein unfolding / 加水分解酵素; 酸無水物に作用; 酸無水物に作用・細胞または細胞小器官の運動に関与 / peroxisome / ATP hydrolysis activity ...protein import into peroxisome matrix, receptor recycling / protein import into peroxisome matrix / transporter complex / protein transporter activity / peroxisomal membrane / ATPase complex / protein unfolding / 加水分解酵素; 酸無水物に作用; 酸無水物に作用・細胞または細胞小器官の運動に関与 / peroxisome / ATP hydrolysis activity / ATP binding / cytosol 類似検索 - 分子機能 | |||||||||||||||
| 生物種 |  | |||||||||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 4.7 Å | |||||||||||||||
 データ登録者 データ登録者 | Ruettermann M / Koci M / Lill P / Geladas ED / Kaschani F / Klink BU / Erdmann R / Gatsogiannis C | |||||||||||||||
| 資金援助 |  ドイツ, 4件 ドイツ, 4件
| |||||||||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2023 ジャーナル: Nat Commun / 年: 2023タイトル: Structure of the peroxisomal Pex1/Pex6 ATPase complex bound to a substrate. 著者: Maximilian Rüttermann / Michelle Koci / Pascal Lill / Ermis Dionysios Geladas / Farnusch Kaschani / Björn Udo Klink / Ralf Erdmann / Christos Gatsogiannis /  要旨: The double-ring AAA+ ATPase Pex1/Pex6 is required for peroxisomal receptor recycling and is essential for peroxisome formation. Pex1/Pex6 mutations cause severe peroxisome associated developmental ...The double-ring AAA+ ATPase Pex1/Pex6 is required for peroxisomal receptor recycling and is essential for peroxisome formation. Pex1/Pex6 mutations cause severe peroxisome associated developmental disorders. Despite its pathophysiological importance, mechanistic details of the heterohexamer are not yet available. Here, we report cryoEM structures of Pex1/Pex6 from Saccharomyces cerevisiae, with an endogenous protein substrate trapped in the central pore of the catalytically active second ring (D2). Pairs of Pex1/Pex6(D2) subdomains engage the substrate via a staircase of pore-1 loops with distinct properties. The first ring (D1) is catalytically inactive but undergoes significant conformational changes resulting in alternate widening and narrowing of its pore. These events are fueled by ATP hydrolysis in the D2 ring and disengagement of a "twin-seam" Pex1/Pex6(D2) heterodimer from the staircase. Mechanical forces are propagated in a unique manner along Pex1/Pex6 interfaces that are not available in homo-oligomeric AAA-ATPases. Our structural analysis reveals the mechanisms of how Pex1 and Pex6 coordinate to achieve substrate translocation. | |||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_16373.map.gz emd_16373.map.gz | 35.4 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-16373-v30.xml emd-16373-v30.xml emd-16373.xml emd-16373.xml | 27.3 KB 27.3 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_16373.png emd_16373.png | 47.9 KB | ||
| Filedesc metadata |  emd-16373.cif.gz emd-16373.cif.gz | 7.9 KB | ||
| その他 |  emd_16373_additional_1.map.gz emd_16373_additional_1.map.gz emd_16373_additional_2.map.gz emd_16373_additional_2.map.gz emd_16373_half_map_1.map.gz emd_16373_half_map_1.map.gz emd_16373_half_map_2.map.gz emd_16373_half_map_2.map.gz | 63.9 MB 203.8 MB 36.5 MB 36.5 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-16373 http://ftp.pdbj.org/pub/emdb/structures/EMD-16373 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-16373 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-16373 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_16373_validation.pdf.gz emd_16373_validation.pdf.gz | 710.9 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_16373_full_validation.pdf.gz emd_16373_full_validation.pdf.gz | 710.4 KB | 表示 | |
| XML形式データ |  emd_16373_validation.xml.gz emd_16373_validation.xml.gz | 15.7 KB | 表示 | |
| CIF形式データ |  emd_16373_validation.cif.gz emd_16373_validation.cif.gz | 18.5 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16373 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16373 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16373 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16373 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8c0wMC  8c0vC C: 同じ文献を引用 ( M: このマップから作成された原子モデル |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_16373.map.gz / 形式: CCP4 / 大きさ: 216 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_16373.map.gz / 形式: CCP4 / 大きさ: 216 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Map generated with SPHIRE Postrefine | ||||||||||||||||||||||||||||||||||||
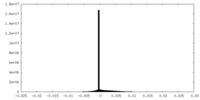
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.68 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-追加マップ: Density-modified map.
| ファイル | emd_16373_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Density-modified map. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
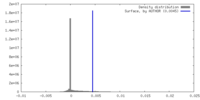
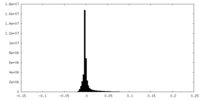
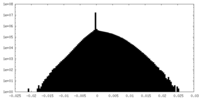
| 密度ヒストグラム |
-追加マップ: Cryosparc NU-refined map.
| ファイル | emd_16373_additional_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Cryosparc NU-refined map. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
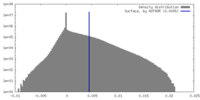
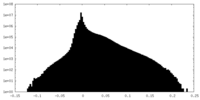
| 密度ヒストグラム |
-ハーフマップ: Half maps from Relion 3D reconstruction. Input for SPHIRE Postrefine.
| ファイル | emd_16373_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half maps from Relion 3D reconstruction. Input for SPHIRE Postrefine. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
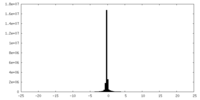
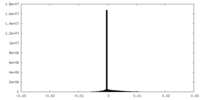
| 密度ヒストグラム |
-ハーフマップ: Half maps from Relion 3D reconstruction. Input for SPHIRE Postrefine.
| ファイル | emd_16373_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half maps from Relion 3D reconstruction. Input for SPHIRE Postrefine. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
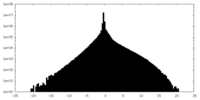
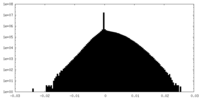
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Peroxisomal AAA ATPase complex Pex1/Pex6("twin seam" state)
| 全体 | 名称: Peroxisomal AAA ATPase complex Pex1/Pex6("twin seam" state) |
|---|---|
| 要素 |
|
-超分子 #1: Peroxisomal AAA ATPase complex Pex1/Pex6("twin seam" state)
| 超分子 | 名称: Peroxisomal AAA ATPase complex Pex1/Pex6("twin seam" state) タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#3 |
|---|---|
| 分子量 | 理論値: 774 KDa |
-超分子 #2: Peroxisomal AAA ATPase complex Pex1/Pex6("twin seam" state)
| 超分子 | 名称: Peroxisomal AAA ATPase complex Pex1/Pex6("twin seam" state) タイプ: complex / ID: 2 / 親要素: 1 / 含まれる分子: #1-#2 |
|---|---|
| 由来(天然) | 生物種:  |
-超分子 #3: Unknown peptide
| 超分子 | 名称: Unknown peptide / タイプ: complex / ID: 3 / 親要素: 1 / 含まれる分子: #3 |
|---|---|
| 由来(天然) | 生物種:  |
-分子 #1: Peroxisomal ATPase PEX6
| 分子 | 名称: Peroxisomal ATPase PEX6 / タイプ: protein_or_peptide / ID: 1 / コピー数: 3 / 光学異性体: LEVO EC番号: 加水分解酵素; 酸無水物に作用; 酸無水物に作用・細胞または細胞小器官の運動に関与 |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 115.687094 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MKASLTFSLS GIYAPCSISR DIYLEYGDKK AECLYGTIRL PQYGPGCTPG KIVHCVLDDS LPFCSIVVPS KLFGFMPTQP TMDFCYFEP ILDNVVPVLD SVTFLINEQL YSKLMDLPQE MQQIQFLHYK YNINSMETVV HSRDILTSGL CQILNCSPFP Q GLVDFTET ...文字列: MKASLTFSLS GIYAPCSISR DIYLEYGDKK AECLYGTIRL PQYGPGCTPG KIVHCVLDDS LPFCSIVVPS KLFGFMPTQP TMDFCYFEP ILDNVVPVLD SVTFLINEQL YSKLMDLPQE MQQIQFLHYK YNINSMETVV HSRDILTSGL CQILNCSPFP Q GLVDFTET QLILVNDTEQ KLSALKYANE DEEYALPKIG TNSALSIDLE SLPCTISRDL LRPAPHINDD NSIYAFTDAE TL LRLDVTS GSFITVSNMG CVRLVKLFVL LLPNGFKKRT IYAPPKIIAS FPDCSVVTIS KSNIGHTDIP IANQVFISRV GGW LQSQKC FQNIILTTLK KFFSESKRIL CQNDLIPIAF DSSMADLNIA EENDESDDED ELGQYYKNDS LVWFFVTSAE LDCF SKDNS HFIIDPNRTK LITTNITNRR PLPLSRSNLQ RYYGFAETFY YDLHIFPYVR QLVNILETSF NCSQRGITLN ASVLL HSTT NNVGKATMVR FASKYLGIHL LEIDCLSLTS NSRQLDSTSK IIGYIRAKCE NVLPYASPAV IFLAHLDSIL LDVNAN QDP EAIKLQKSIN FEMSKLLDDF TFKFPGTTFV GSVNNIDNVP SSFRSHMRFE ILVPVPSEAQ RLRIFQWYLS SHELNRD VQ QKVPVSYMDN ISFSSLSSYS AGLTPLDIKS IVETARMTAT ARFYQESKKC GWLPQSILIT QEDLSKATSK ARNEFSVS I GAPQIPNVTW DDIGGIDFVK GEILDTIDMP LKHPELFTSG MKKRSGILFY GPPGTGKTLM AKAIATNFSL NFFSVKGPE LLNMYIGESE ANVRRVFQKA REAKPCVIFF DQIDSVAPKR GNQGDSGGVM DRIVSQLLAE LDGMSTDADG VFVIGATNRP DLLDEALLR PGRFDKLLYL GIPDTDTKQL NILEALTRKF VLDNDVKLIE LAKLCPFNYT GADFYALCSD AMLNAMSRIA R MVEKKVSQ HNELTGENIS TRRWFDKIAT KEDTKVVVKM EDFLKAQEQL TPSVSRAELN HYEAVRANFE GA UniProtKB: Peroxisomal ATPase PEX6 |
-分子 #2: Peroxisomal ATPase PEX1
| 分子 | 名称: Peroxisomal ATPase PEX1 / タイプ: protein_or_peptide / ID: 2 / コピー数: 3 / 光学異性体: LEVO EC番号: 加水分解酵素; 酸無水物に作用; 酸無水物に作用・細胞または細胞小器官の運動に関与 |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 92.758891 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: TILKNGAIQL LKKVILRSTV CKMDFPKDNL FVVYISDGAQ LPSQKGYASI VKCSLRQSKK SDSDNKSVGI PSKKIGVFIK CDSQIPENH IALSSHLWDA FFTHPMNGAK IKLEFLQMNQ ANIISGRNAT VNIKYFGKDV PTKSGDQYSK LLGGSLLTNN L ILPTEQII ...文字列: TILKNGAIQL LKKVILRSTV CKMDFPKDNL FVVYISDGAQ LPSQKGYASI VKCSLRQSKK SDSDNKSVGI PSKKIGVFIK CDSQIPENH IALSSHLWDA FFTHPMNGAK IKLEFLQMNQ ANIISGRNAT VNIKYFGKDV PTKSGDQYSK LLGGSLLTNN L ILPTEQII IEIKKGESEQ QLCNLNEISN ESVQWKVTQM GKEEVKDIIE RHLPKHYHVK ETGEVSRTSK DEDDFITVNS IK KEMVNYL TSPIIATPAI ILDGKQGIGK TRLLKELINE VEKDHHIFVK YADCETLHET SNLDKTQKLI MEWCSFCYWY GPS LIVLDN VEALFGKPQA NDGDPSNNGQ WDNASKLLNF FINQVTKIFN KDNKRIRVLF SGKQKTQINP LLFDKHFVSE TWSL RAPDK HARAKLLEYF FSKNQIMKLN RDLQFSDLSL ETEGFSPLDL EIFTEKIFYD LQLERDCDNV VTRELFSKSL SAFTP SALR GVKLTKETNI KWGDIGALAN AKDVLLETLE WPTKYEPIFV NCPLRLRSGI LLYGYPGCGK TLLASAVAQQ CGLNFI SVK GPEILNKFIG ASEQNIRELF ERAQSVKPCI LFFDEFDSIA PKRGHDSTGV TDRVVNQLLT QMDGAEGLDG VYILAAT SR PDLIDSALLR PGRLDKSVIC NIPTESERLD ILQAIVNSKD KDTGQKKFAL EKNADLKLIA EKTAGFSGAD LQGLCYNA Y LKSVHRWLSA ADQSEVVPGN DNIEYFSINE HGRREENRLR LKTLLQQDVV HETKTSTSAA SELTAVVTIN DLLEACQET KPSISTSELV KLRGIYDRFQ KDR UniProtKB: Peroxisomal ATPase PEX1 |
-分子 #3: unknown peptide
| 分子 | 名称: unknown peptide / タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 783.958 Da |
| 配列 | 文字列: (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) |
-分子 #4: ADENOSINE-5'-TRIPHOSPHATE
| 分子 | 名称: ADENOSINE-5'-TRIPHOSPHATE / タイプ: ligand / ID: 4 / コピー数: 10 / 式: ATP |
|---|---|
| 分子量 | 理論値: 507.181 Da |
| Chemical component information |  ChemComp-ATP: |
-分子 #5: MAGNESIUM ION
| 分子 | 名称: MAGNESIUM ION / タイプ: ligand / ID: 5 / コピー数: 10 / 式: MG |
|---|---|
| 分子量 | 理論値: 24.305 Da |
-分子 #6: ADENOSINE-5'-DIPHOSPHATE
| 分子 | 名称: ADENOSINE-5'-DIPHOSPHATE / タイプ: ligand / ID: 6 / コピー数: 2 / 式: ADP |
|---|---|
| 分子量 | 理論値: 427.201 Da |
| Chemical component information |  ChemComp-ADP: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.4 |
|---|---|
| グリッド | モデル: Quantifoil / 材質: COPPER / 前処理 - タイプ: GLOW DISCHARGE |
| 凍結 | 凍結剤: ETHANE / 装置: FEI VITROBOT MARK II 詳細: Grids were blotted for 3.5 sec and plunge-frozen after 1 sec drain time at 100% humidity. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 特殊光学系 | エネルギーフィルター - 名称: GIF Bioquantum |
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 実像数: 16763 / 平均電子線量: 60.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: SPOT SCAN / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.4 µm / 最小 デフォーカス(公称値): 0.6 µm / 倍率(公称値): 105000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: AB INITIO MODEL |
|---|---|
| 得られたモデル |  PDB-8c0w: |
 ムービー
ムービー コントローラー
コントローラー








 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)