+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | In situ structure of the Caulobacter crescentus S-layer | |||||||||
 マップデータ マップデータ | PostProcessed map with B-factor sharpening | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | RsaA S-layer sub-tomogram averaging Caulobacter / STRUCTURAL PROTEIN | |||||||||
| 機能・相同性 | RsaA N-terminal domain / RTX calcium-binding nonapeptide repeat / RTX calcium-binding nonapeptide repeat (4 copies) / Serralysin-like metalloprotease, C-terminal / calcium ion binding / S-layer protein rsaA 機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |  Caulobacter vibrioides NA1000 (バクテリア) Caulobacter vibrioides NA1000 (バクテリア) | |||||||||
| 手法 | サブトモグラム平均法 / クライオ電子顕微鏡法 / 解像度: 3.5 Å | |||||||||
 データ登録者 データ登録者 | von Kuegelgen A / Bharat T | |||||||||
| 資金援助 |  英国, 2件 英国, 2件
| |||||||||
 引用 引用 |  ジャーナル: Elife / 年: 2022 ジャーナル: Elife / 年: 2022タイトル: A Bayesian approach to single-particle electron cryo-tomography in RELION-4.0. 著者: Jasenko Zivanov / Joaquín Otón / Zunlong Ke / Andriko von Kügelgen / Euan Pyle / Kun Qu / Dustin Morado / Daniel Castaño-Díez / Giulia Zanetti / Tanmay A M Bharat / John A G Briggs / Sjors H W Scheres /     要旨: We present a new approach for macromolecular structure determination from multiple particles in electron cryo-tomography (cryo-ET) data sets. Whereas existing subtomogram averaging approaches are ...We present a new approach for macromolecular structure determination from multiple particles in electron cryo-tomography (cryo-ET) data sets. Whereas existing subtomogram averaging approaches are based on 3D data models, we propose to optimise a regularised likelihood target that approximates a function of the 2D experimental images. In addition, analogous to Bayesian polishing and contrast transfer function (CTF) refinement in single-particle analysis, we describe the approaches that exploit the increased signal-to-noise ratio in the averaged structure to optimise tilt-series alignments, beam-induced motions of the particles throughout the tilt-series acquisition, defoci of the individual particles, as well as higher-order optical aberrations of the microscope. Implementation of our approaches in the open-source software package RELION aims to facilitate their general use, particularly for those researchers who are already familiar with its single-particle analysis tools. We illustrate for three applications that our approaches allow structure determination from cryo-ET data to resolutions sufficient for de novo atomic modelling. #1:  ジャーナル: BioRxiv / 年: 2022 ジャーナル: BioRxiv / 年: 2022タイトル: A Bayesian approach to single-particle electron cryo-tomography in RELION-4.0 著者: Zivanov J / Oton J / Ke Z / von Kuegelgen A / Pyle E / Qu K / Morado D / Castano-Diez D / Zanetti G / Bharat TAM / Briggs JAG / Scheres SHW | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_16183.map.gz emd_16183.map.gz | 13.7 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-16183-v30.xml emd-16183-v30.xml emd-16183.xml emd-16183.xml | 21.8 KB 21.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_16183.png emd_16183.png | 175.8 KB | ||
| マスクデータ |  emd_16183_msk_1.map emd_16183_msk_1.map | 15.6 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-16183.cif.gz emd-16183.cif.gz | 7.1 KB | ||
| その他 |  emd_16183_half_map_1.map.gz emd_16183_half_map_1.map.gz emd_16183_half_map_2.map.gz emd_16183_half_map_2.map.gz | 7.1 MB 7.1 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-16183 http://ftp.pdbj.org/pub/emdb/structures/EMD-16183 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-16183 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-16183 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_16183_validation.pdf.gz emd_16183_validation.pdf.gz | 957.6 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_16183_full_validation.pdf.gz emd_16183_full_validation.pdf.gz | 957.2 KB | 表示 | |
| XML形式データ |  emd_16183_validation.xml.gz emd_16183_validation.xml.gz | 9.7 KB | 表示 | |
| CIF形式データ |  emd_16183_validation.cif.gz emd_16183_validation.cif.gz | 11.2 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16183 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16183 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16183 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16183 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_16183.map.gz / 形式: CCP4 / 大きさ: 15.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_16183.map.gz / 形式: CCP4 / 大きさ: 15.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | PostProcessed map with B-factor sharpening | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.35 Å | ||||||||||||||||||||||||||||||||||||
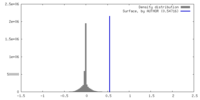
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_16183_msk_1.map emd_16183_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
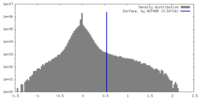
| 密度ヒストグラム |
-ハーフマップ: Half map 1
| ファイル | emd_16183_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map 1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
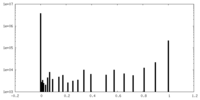
| 密度ヒストグラム |
-ハーフマップ: Half map 2
| ファイル | emd_16183_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map 2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
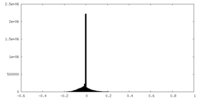
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Caulobacter crescentus S-layer
| 全体 | 名称: Caulobacter crescentus S-layer |
|---|---|
| 要素 |
|
-超分子 #1: Caulobacter crescentus S-layer
| 超分子 | 名称: Caulobacter crescentus S-layer / タイプ: organelle_or_cellular_component / ID: 1 / 親要素: 0 / 含まれる分子: #1 / 詳細: Caulobacter crescentus S-layer |
|---|---|
| 由来(天然) | 生物種:  Caulobacter vibrioides NA1000 (バクテリア) / 株: YB2811 / 細胞中の位置: extra-cellular Caulobacter vibrioides NA1000 (バクテリア) / 株: YB2811 / 細胞中の位置: extra-cellular |
-分子 #1: S-layer protein rsaA
| 分子 | 名称: S-layer protein rsaA / タイプ: protein_or_peptide / ID: 1 / 詳細: In-situ S-layer / コピー数: 6 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Caulobacter vibrioides NA1000 (バクテリア) / 株: NA1000 / CB15N Caulobacter vibrioides NA1000 (バクテリア) / 株: NA1000 / CB15N |
| 分子量 | 理論値: 98.153906 KDa |
| 組換発現 | 生物種:  Caulobacter vibrioides NA1000 (バクテリア) Caulobacter vibrioides NA1000 (バクテリア) |
| 配列 | 文字列: MAYTTAQLVT AYTNANLGKA PDAATTLTLD AYATQTQTGG LSDAAALTNT LKLVNSTTAV AIQTYQFFTG VAPSAAGLDF LVDSTTNTN DLNDAYYSKF AQENRFINFS INLATGAGAG ATAFAAAYTG VSYAQTVATA YDKIIGNAVA TAAGVDVAAA V AFLSRQAN ...文字列: MAYTTAQLVT AYTNANLGKA PDAATTLTLD AYATQTQTGG LSDAAALTNT LKLVNSTTAV AIQTYQFFTG VAPSAAGLDF LVDSTTNTN DLNDAYYSKF AQENRFINFS INLATGAGAG ATAFAAAYTG VSYAQTVATA YDKIIGNAVA TAAGVDVAAA V AFLSRQAN IDYLTAFVRA NTPFTAAADI DLAVKAALIG TILNAATVSG IGGYATATAA MINDLSDGAL STDNAAGVNL FT AYPSSGV SGSTLSLTTG TDTLTGTANN DTFVAGEVAG AATLTVGDTL SGGAGTDVLN WVQAAAVTAL PTGVTISGIE TMN VTSGAA ITLNTSSGVT GLTALNTNTS GAAQTVTAGA GQNLTATTAA QAANNVAVDG GANVTVASTG VTSGTTTVGA NSAA SGTVS VSVANSSTTT TGAIAVTGGT AVTVAQTAGN AVNTTLTQAD VTVTGNSSTT AVTVTQTAAA TAGATVAGRV NGAVT ITDS AAASATTAGK IATVTLGSFG AATIDSSALT TVNLSGTGTS LGIGRGALTA TPTANTLTLN VNGLTTTGAI TDSEAA ADD GFTTINIAGS TASSTIASLV AADATTLNIS GDARVTITSH TAAALTGITV TNSVGATLGA ELATGLVFTG GAGADSI LL GATTKAIVMG AGDDTVTVSS ATLGAGGSVN GGDGTDVLVA NVNGSSFSAD PAFGGFETLR VAGAAAQGSH NANGFTAL Q LGATAGATTF TNVAVNVGLT VLAAPTGTTT VTLANATGTS DVFNLTLSSS AALAAGTVAL AGVETVNIAA TDTNTTAHV DTLTLQATSA KSIVVTGNAG LNLTNTGNTA VTSFDASAVT GTGSAVTFVS ANTTVGEVVT IRGGAGADSL TGSATANDTI IGGAGADTL VYTGGTDTFT GGTGADIFDI NAIGTSTAFV TITDAAVGDK LDLVGISTNG AIADGAFGAA VTLGAAATLA Q YLDAAAAG DGSGTSVAKW FQFGGDTYVV VDSSAGATFV SGADAVIKLT GLVTLTTSAF ATEVLTLA UniProtKB: S-layer protein rsaA |
-分子 #3: CALCIUM ION
| 分子 | 名称: CALCIUM ION / タイプ: ligand / ID: 3 / コピー数: 18 / 式: CA |
|---|---|
| 分子量 | 理論値: 40.078 Da |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | サブトモグラム平均法 |
| 試料の集合状態 | cell |
- 試料調製
試料調製
| 緩衝液 | pH: 7 / 詳細: PYE medium |
|---|---|
| グリッド | モデル: Quantifoil R2/2 / 材質: COPPER/RHODIUM / メッシュ: 200 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY ARRAY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 20 sec. / 前処理 - 雰囲気: AIR / 詳細: 15 mA |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 283.15 K / 装置: FEI VITROBOT MARK IV / 詳細: 1.5 s blot. |
| 詳細 | Caulobacter crescentus stalk |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 温度 | 最低: 70.0 K / 最高: 70.0 K |
| 特殊光学系 | 球面収差補正装置: not used / 色収差補正装置: not used / エネルギーフィルター - 名称: GIF Quantum LS / エネルギーフィルター - スリット幅: 20 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: SUPER-RESOLUTION / デジタル化 - 画像ごとのフレーム数: 1-10 / 実像数: 1 / 平均露光時間: 1.0 sec. / 平均電子線量: 3.4 e/Å2 / 詳細: Dose symmetric tilt scheme (Hagen et al, JSB) |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 70.0 µm / 最大 デフォーカス(補正後): 5.0 µm / 最小 デフォーカス(補正後): 2.0 µm / 倍率(補正後): 105000 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 5.0 µm / 最小 デフォーカス(公称値): 2.0 µm / 倍率(公称値): 105000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 最終 再構成 | 使用したクラス数: 3 / 想定した対称性 - 点群: C6 (6回回転対称) / アルゴリズム: FOURIER SPACE / 解像度のタイプ: BY AUTHOR / 解像度: 3.5 Å / 解像度の算出法: FSC 0.143 CUT-OFF / ソフトウェア - 名称: RELION / 使用したサブトモグラム数: 42990 |
|---|---|
| 抽出 | トモグラム数: 110 / 使用した粒子像数: 51866 / 参照モデル: EMDF / 手法: EMD-10388 / ソフトウェア - 名称: RELION (ver. 4.0.0) / 詳細: RELION subtomogram averaging |
| 最終 3次元分類 | クラス数: 6 / 平均メンバー数/クラス: 8645 / ソフトウェア - 名称: RELION (ver. 4.0.0) |
| 最終 角度割当 | タイプ: MAXIMUM LIKELIHOOD / ソフトウェア - 名称: RELION (ver. 4.0.0) / 詳細: RELION 4.0.0 |
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT |
|---|---|
| 得られたモデル |  PDB-8bqe: |
 ムービー
ムービー コントローラー
コントローラー








 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)