+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | ||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | SPA of Trypsin untreated Rotavirus TLP spike | ||||||||||||||||||
 マップデータ マップデータ | |||||||||||||||||||
 試料 試料 |
| ||||||||||||||||||
 キーワード キーワード | Rotavirus / SPA / VIRAL PROTEIN | ||||||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報viral intermediate capsid / host cell endoplasmic reticulum lumen / host cell rough endoplasmic reticulum / T=2 icosahedral viral capsid / permeabilization of host organelle membrane involved in viral entry into host cell / T=13 icosahedral viral capsid / viral inner capsid / host cytoskeleton / viral outer capsid / host cell endoplasmic reticulum-Golgi intermediate compartment ...viral intermediate capsid / host cell endoplasmic reticulum lumen / host cell rough endoplasmic reticulum / T=2 icosahedral viral capsid / permeabilization of host organelle membrane involved in viral entry into host cell / T=13 icosahedral viral capsid / viral inner capsid / host cytoskeleton / viral outer capsid / host cell endoplasmic reticulum-Golgi intermediate compartment / viral nucleocapsid / host cell surface receptor binding / fusion of virus membrane with host plasma membrane / viral envelope / virion attachment to host cell / host cell plasma membrane / structural molecule activity / RNA binding / metal ion binding / membrane 類似検索 - 分子機能 | ||||||||||||||||||
| 生物種 |  Rotavirus A (ロタウイルス A) Rotavirus A (ロタウイルス A) | ||||||||||||||||||
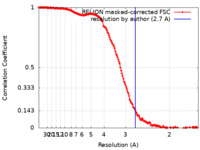
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.7 Å | ||||||||||||||||||
 データ登録者 データ登録者 | Shah PNM / Stuart DI | ||||||||||||||||||
| 資金援助 |  英国, 5件 英国, 5件
| ||||||||||||||||||
 引用 引用 |  ジャーナル: Cell Host Microbe / 年: 2023 ジャーナル: Cell Host Microbe / 年: 2023タイトル: Characterization of the rotavirus assembly pathway in situ using cryoelectron tomography. 著者: Pranav N M Shah / James B Gilchrist / Björn O Forsberg / Alister Burt / Andrew Howe / Shyamal Mosalaganti / William Wan / Julika Radecke / Yuriy Chaban / Geoff Sutton / David I Stuart / Mark Boyce /    要旨: Rotavirus assembly is a complex process that involves the stepwise acquisition of protein layers in distinct intracellular locations to form the fully assembled particle. Understanding and ...Rotavirus assembly is a complex process that involves the stepwise acquisition of protein layers in distinct intracellular locations to form the fully assembled particle. Understanding and visualization of the assembly process has been hampered by the inaccessibility of unstable intermediates. We characterize the assembly pathway of group A rotaviruses observed in situ within cryo-preserved infected cells through the use of cryoelectron tomography of cellular lamellae. Our findings demonstrate that the viral polymerase VP1 recruits viral genomes during particle assembly, as revealed by infecting with a conditionally lethal mutant. Additionally, pharmacological inhibition to arrest the transiently enveloped stage uncovered a unique conformation of the VP4 spike. Subtomogram averaging provided atomic models of four intermediate states, including a pre-packaging single-layered intermediate, the double-layered particle, the transiently enveloped double-layered particle, and the fully assembled triple-layered virus particle. In summary, these complementary approaches enable us to elucidate the discrete steps involved in forming an intracellular rotavirus particle. | ||||||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_16146.map.gz emd_16146.map.gz | 317.7 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-16146-v30.xml emd-16146-v30.xml emd-16146.xml emd-16146.xml | 21.9 KB 21.9 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_16146_fsc.xml emd_16146_fsc.xml | 15.9 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_16146.png emd_16146.png | 174.4 KB | ||
| Filedesc metadata |  emd-16146.cif.gz emd-16146.cif.gz | 7.3 KB | ||
| その他 |  emd_16146_half_map_1.map.gz emd_16146_half_map_1.map.gz emd_16146_half_map_2.map.gz emd_16146_half_map_2.map.gz | 273.8 MB 273.9 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-16146 http://ftp.pdbj.org/pub/emdb/structures/EMD-16146 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-16146 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-16146 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8bp8MC  8co6C  8coaC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_16146.map.gz / 形式: CCP4 / 大きさ: 343 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_16146.map.gz / 形式: CCP4 / 大きさ: 343 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.82 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #2
| ファイル | emd_16146_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_16146_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Rotavirus A
| 全体 | 名称:  Rotavirus A (ロタウイルス A) Rotavirus A (ロタウイルス A) |
|---|---|
| 要素 |
|
-超分子 #1: Rotavirus A
| 超分子 | 名称: Rotavirus A / タイプ: virus / ID: 1 / 親要素: 0 / 含まれる分子: #1-#4 / NCBI-ID: 28875 / 生物種: Rotavirus A / ウイルスタイプ: VIRION / ウイルス・単離状態: STRAIN / ウイルス・エンベロープ: No / ウイルス・中空状態: No |
|---|
-分子 #1: Outer capsid protein VP4
| 分子 | 名称: Outer capsid protein VP4 / タイプ: protein_or_peptide / ID: 1 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Rotavirus A (ロタウイルス A) Rotavirus A (ロタウイルス A) |
| 分子量 | 理論値: 86.767953 KDa |
| 組換発現 | 生物種:  Chlorocebus aethiops aethiops (ミドリザル) Chlorocebus aethiops aethiops (ミドリザル) |
| 配列 | 文字列: MASLIYRQLL TNSYTVDLSD EIQEIGSTKS QNVTINPGPF AQTGYAPVNW GPGEINDSTT VGPLLDGPYQ PTTFNPPVDY WMLLAPTTP GVIVEGTNNT DRWLATILIE PNVQSENRTY TIFGIQEQLT VSNTSQDQWK FIDVVKTTAN GSIGQYGPLL S SPKLYAVM ...文字列: MASLIYRQLL TNSYTVDLSD EIQEIGSTKS QNVTINPGPF AQTGYAPVNW GPGEINDSTT VGPLLDGPYQ PTTFNPPVDY WMLLAPTTP GVIVEGTNNT DRWLATILIE PNVQSENRTY TIFGIQEQLT VSNTSQDQWK FIDVVKTTAN GSIGQYGPLL S SPKLYAVM KHNEKLYTYE GQTPNATTAH YSTTNYDSVN MTAFCDFYII PRSEESKCTE YINNGLPPIQ NTRNVVPLSL TA RDVIHYR AQANEDIVIS KTSLWKEMQY NRDITIRFKF ANTIIKSGGL GYKWSEISFK PANYQYTYTR DGEEVTAHTT CSV NGMNDF SFNGGYLPTD FVVSKFEVIK ENSYVYIDYW DDSQAFRNVV YVRSLAANLN SVMCTGGSYN FSLPVGQWPV LTGG AVSLH SAGVTLSTQF TDFVSLNSLR FRFRLAVEEP HFKLTRTRLD RLYGLPAADP NNGKEYYEIA GRFSLISLVP SNDDY QTPI ANSVTVRQDL ERQLGELREE FNALSQEIAM SQLIDLALLP LDMFSMFSGI KSTIDAAKSM ATNVMKKFKK SGLANS VST LTDSLSDAAS SISRGSSIRS IGSSASAWTD VSTQITDISS SVSSVSTQTS TISRRLRLKE MATQTEGMNF DDISAAV LK TKIDKSTQIS PNTIPDIVTE ASEKFIPNRA YRVINNDDVF EAGIDGKFFA YKVDTFEEIP FDVQKFADLV TDSPVISA I IDFKTLKNLN DNYGISRQQA FNLLRSDPRV LREFINQDNP IIRNRIEQLI MQCRL UniProtKB: Outer capsid protein VP4 |
-分子 #2: Outer capsid glycoprotein VP7
| 分子 | 名称: Outer capsid glycoprotein VP7 / タイプ: protein_or_peptide / ID: 2 / コピー数: 13 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Rotavirus A (ロタウイルス A) Rotavirus A (ロタウイルス A) |
| 分子量 | 理論値: 37.230664 KDa |
| 組換発現 | 生物種:  Chlorocebus aethiops aethiops (ミドリザル) Chlorocebus aethiops aethiops (ミドリザル) |
| 配列 | 文字列: MYGIEYTTVL TFLISIILLN YILKSLTRIM DFIIYRFLFI IVILSPFLRA QNYGINLPIT GSMDIAYANS TQEEPFLTST LCLYYPTEA ATEINDNSWK DTLSQLFLTK GWPTGSVYFK EYTNIASFSV DPQLYCDYNV VLMKYDATLQ LDMSELADLI L NEWLCNPM ...文字列: MYGIEYTTVL TFLISIILLN YILKSLTRIM DFIIYRFLFI IVILSPFLRA QNYGINLPIT GSMDIAYANS TQEEPFLTST LCLYYPTEA ATEINDNSWK DTLSQLFLTK GWPTGSVYFK EYTNIASFSV DPQLYCDYNV VLMKYDATLQ LDMSELADLI L NEWLCNPM DITLYYYQQT DEANKWISMG SSCTIKVCPL NTQTLGIGCL TTDATTFEEV ATAEKLVITD VVDGVNHKLD VT TATCTIR NCKKLGPREN VAVIQVGGSD ILDITADPTT APQTERMMRI NWKKWWQVFY TVVDYVDQII QVMSKRSRSL NSA AFYYRV UniProtKB: Outer capsid glycoprotein VP7 |
-分子 #3: Intermediate capsid protein VP6
| 分子 | 名称: Intermediate capsid protein VP6 / タイプ: protein_or_peptide / ID: 3 / コピー数: 13 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Rotavirus A (ロタウイルス A) Rotavirus A (ロタウイルス A) |
| 分子量 | 理論値: 44.910738 KDa |
| 組換発現 | 生物種:  Chlorocebus aethiops aethiops (ミドリザル) Chlorocebus aethiops aethiops (ミドリザル) |
| 配列 | 文字列: MDVLYSLSKT LKDARDKIVE GTLYSNVSDL IQQFNQMIIT MNGNEFQTGG IGNLPIRNWN FNFGLLGTTL LNLDANYVET ARNTIDYFV DFVDNVCMDE MVRESQRNGI APQSDSLRKL SAIKFKRINF DNSSEYIENW NLQNRRQRTG FTFHKPNIFP Y SASFTLNR ...文字列: MDVLYSLSKT LKDARDKIVE GTLYSNVSDL IQQFNQMIIT MNGNEFQTGG IGNLPIRNWN FNFGLLGTTL LNLDANYVET ARNTIDYFV DFVDNVCMDE MVRESQRNGI APQSDSLRKL SAIKFKRINF DNSSEYIENW NLQNRRQRTG FTFHKPNIFP Y SASFTLNR SQPAHDNLMG TMWLNAGSEI QVAGFDYSCA INAPANIQQF EHIVPLRRVL TTATITLLPD AERFSFPRVI NS ADGATTW FFNPVILRPN NVEVEFLLNG QIINTYQARF GTIVARNFDT IRLSFQLMRP PNMTPAVAVL FPNAQPFEHH ATV GLTLRI ESAVCESVLA DASETLLANV TSVRQEYAIP VGPVFPPGMN WTDLITNYSP SREDNLQRVF TVASIRSMLI K UniProtKB: Intermediate capsid protein VP6 |
-分子 #4: Inner capsid protein VP2
| 分子 | 名称: Inner capsid protein VP2 / タイプ: protein_or_peptide / ID: 4 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Rotavirus A (ロタウイルス A) Rotavirus A (ロタウイルス A) |
| 分子量 | 理論値: 102.412961 KDa |
| 組換発現 | 生物種:  Chlorocebus aethiops aethiops (ミドリザル) Chlorocebus aethiops aethiops (ミドリザル) |
| 配列 | 文字列: MAYRKRGARR ETNLKQDERM QEKEDSKNIN NDSPKSQLSE KVLSKKEEII TDNQEEVKIS DEVKKSNKEE SKQLLEVLKT KEEHQKEVQ YEILQKTIPT FEPKESILKK LEDIKPEQAK AQTKLFRIFE PRQKPIYIAN GEKERRNRIY WKLKKDTLPD G DYDVREYF ...文字列: MAYRKRGARR ETNLKQDERM QEKEDSKNIN NDSPKSQLSE KVLSKKEEII TDNQEEVKIS DEVKKSNKEE SKQLLEVLKT KEEHQKEVQ YEILQKTIPT FEPKESILKK LEDIKPEQAK AQTKLFRIFE PRQKPIYIAN GEKERRNRIY WKLKKDTLPD G DYDVREYF LNLYDQVLTE MPDYLLLKDM AVENKNSRDA GKVVDSETAS ICDAIFQDEE TEGAVARFIA EMRQRVQADR NV VNYPSIL HPIDYAFNEY FLQHQLVEPL NNDIIFNYIP ERIRNDVNYI LNMDRNLPST ARYIRPNLLQ DRLNLHDNFE SLW DTITTS NYILARSVVP DLKELVSTEA QIQKMSQDLQ LEALTIQSET QFLTGINSQA ANDCFKTLIA AMLSQRTMSL DFVT TNYMS LISGMWLLTV VPNDMFIRES LVACQLAIVN TIIYVAFGMQ RMHYRNGDPQ TPFQIAEQQI QNFQVANWLH FVNNN YFRQ VVIDGVLNQV LNDNIRNGHV INQLMEALMQ LSRQQFPTMP VDYKRSIQRG ILLLSNRLGQ LVDLTRLLAY SYETLM ACV TMNMQHVQTL TTEKLQLTSV TSLCMLIGNA TVIPSPQTLF HYYNVNVNFH SNYNERINDA VAIITAANRL NLYQKKM KA IVEDFLKRLH IFDVAVAPDD QMYRLRDRLR LLPVEVRRLD IFNLILMAMD QIERASDKIA QGVIIAYRDM QLERDEMY G YVNIARNLDG FQQINLEELM RTGSYAQITN MLLNNQPVAL VGALPFVTDS SVISLIAILD ATVFAQIVKL RKVDTLKPI LYKINSDSND FYLVANYDWV PISTTKVYKQ VPVQFDFRNS MHMLTSNLTF TVYSDLLAFV SADTVEPINA VAFDNMRIMN EL UniProtKB: Inner capsid protein VP2 |
-分子 #5: CALCIUM ION
| 分子 | 名称: CALCIUM ION / タイプ: ligand / ID: 5 / コピー数: 39 / 式: CA |
|---|---|
| 分子量 | 理論値: 40.078 Da |
-分子 #6: ZINC ION
| 分子 | 名称: ZINC ION / タイプ: ligand / ID: 6 / コピー数: 5 / 式: ZN |
|---|---|
| 分子量 | 理論値: 65.409 Da |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 7 mg/mL |
|---|---|
| 緩衝液 | pH: 7.5 |
| グリッド | モデル: Quantifoil R2/1 / 材質: COPPER / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE |
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | TFS KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / 平均電子線量: 39.8 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: OTHER / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 3.0 µm / 最小 デフォーカス(公称値): 0.5 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー
















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)