+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | hexameric ct-sc 5S RNP monomer | ||||||||||||
 マップデータ マップデータ | Hexameric ct-sc 5S RNP monomer (Local filtered map) (containing Rpf2, Rrs1 and Syo1 from C. thermophilum and uL18/L5, uL5/L11 and 5S rRNA from S. cerevisiae) | ||||||||||||
 試料 試料 |
| ||||||||||||
 キーワード キーワード | 5S RNP / Ribosome biogenesis / Symportin 1 / Rpf2-Rrs1 / RNA BINDING PROTEIN | ||||||||||||
| 生物種 |  Thermochaetoides thermophila DSM 1495 (菌類) Thermochaetoides thermophila DSM 1495 (菌類) | ||||||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 4.1 Å | ||||||||||||
 データ登録者 データ登録者 | Castillo N / Thoms M / Flemming D / Hammaren HM / Buschauer R / Ameismeier M / Bassler J / Beck M / Beckmann R / Hurt E | ||||||||||||
| 資金援助 |  ドイツ, European Union, 3件 ドイツ, European Union, 3件
| ||||||||||||
 引用 引用 |  ジャーナル: Nat Struct Mol Biol / 年: 2023 ジャーナル: Nat Struct Mol Biol / 年: 2023タイトル: Structure of nascent 5S RNPs at the crossroad between ribosome assembly and MDM2-p53 pathways. 著者: Nestor Miguel Castillo Duque de Estrada / Matthias Thoms / Dirk Flemming / Henrik M Hammaren / Robert Buschauer / Michael Ameismeier / Jochen Baßler / Martin Beck / Roland Beckmann / Ed Hurt /  要旨: The 5S ribonucleoprotein (RNP) is assembled from its three components (5S rRNA, Rpl5/uL18 and Rpl11/uL5) before being incorporated into the pre-60S subunit. However, when ribosome synthesis is ...The 5S ribonucleoprotein (RNP) is assembled from its three components (5S rRNA, Rpl5/uL18 and Rpl11/uL5) before being incorporated into the pre-60S subunit. However, when ribosome synthesis is disturbed, a free 5S RNP can enter the MDM2-p53 pathway to regulate cell cycle and apoptotic signaling. Here we reconstitute and determine the cryo-electron microscopy structure of the conserved hexameric 5S RNP with fungal or human factors. This reveals how the nascent 5S rRNA associates with the initial nuclear import complex Syo1-uL18-uL5 and, upon further recruitment of the nucleolar factors Rpf2 and Rrs1, develops into the 5S RNP precursor that can assemble into the pre-ribosome. In addition, we elucidate the structure of another 5S RNP intermediate, carrying the human ubiquitin ligase Mdm2, which unravels how this enzyme can be sequestered from its target substrate p53. Our data provide molecular insight into how the 5S RNP can mediate between ribosome biogenesis and cell proliferation. | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_16037.map.gz emd_16037.map.gz | 3 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-16037-v30.xml emd-16037-v30.xml emd-16037.xml emd-16037.xml | 17.5 KB 17.5 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_16037.png emd_16037.png | 64.1 KB | ||
| その他 |  emd_16037_additional_1.map.gz emd_16037_additional_1.map.gz emd_16037_additional_2.map.gz emd_16037_additional_2.map.gz emd_16037_half_map_1.map.gz emd_16037_half_map_1.map.gz emd_16037_half_map_2.map.gz emd_16037_half_map_2.map.gz | 51.4 MB 97.3 MB 95.7 MB 95.8 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-16037 http://ftp.pdbj.org/pub/emdb/structures/EMD-16037 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-16037 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-16037 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_16037_validation.pdf.gz emd_16037_validation.pdf.gz | 642.3 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_16037_full_validation.pdf.gz emd_16037_full_validation.pdf.gz | 641.9 KB | 表示 | |
| XML形式データ |  emd_16037_validation.xml.gz emd_16037_validation.xml.gz | 13.4 KB | 表示 | |
| CIF形式データ |  emd_16037_validation.cif.gz emd_16037_validation.cif.gz | 15.9 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16037 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16037 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16037 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16037 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_16037.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_16037.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Hexameric ct-sc 5S RNP monomer (Local filtered map) (containing Rpf2, Rrs1 and Syo1 from C. thermophilum and uL18/L5, uL5/L11 and 5S rRNA from S. cerevisiae) | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.059 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-追加マップ: Hexameric ct-sc 5S RNP monomer (Non-uniform Refinement)
| ファイル | emd_16037_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Hexameric ct-sc 5S RNP monomer (Non-uniform Refinement) | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
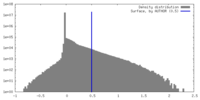
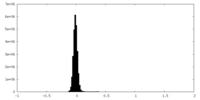
| 密度ヒストグラム |
-追加マップ: Hexameric ct-sc 5S RNP monomer (Non-uniform Refinement, sharpened...
| ファイル | emd_16037_additional_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Hexameric ct-sc 5S RNP monomer (Non-uniform Refinement, sharpened map) | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
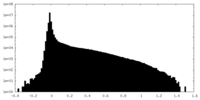
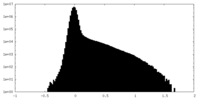
| 密度ヒストグラム |
-ハーフマップ: Hexameric ct-sc 5S RNP monomer (half map A...
| ファイル | emd_16037_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Hexameric ct-sc 5S RNP monomer (half map A from the non-uniform Refinement) | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
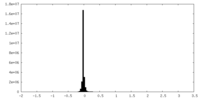
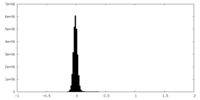
| 密度ヒストグラム |
-ハーフマップ: Hexameric ct-sc 5S RNP monomer (half map B...
| ファイル | emd_16037_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Hexameric ct-sc 5S RNP monomer (half map B from the non-uniform Refinement) | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
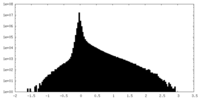
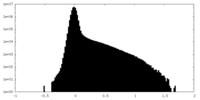
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : hexameric ct-sc 5S RNP monomer
| 全体 | 名称: hexameric ct-sc 5S RNP monomer |
|---|---|
| 要素 |
|
-超分子 #1: hexameric ct-sc 5S RNP monomer
| 超分子 | 名称: hexameric ct-sc 5S RNP monomer / タイプ: complex / ID: 1 / 親要素: 0 |
|---|---|
| 由来(天然) | 生物種:  Thermochaetoides thermophila DSM 1495 (菌類) Thermochaetoides thermophila DSM 1495 (菌類) |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 |
|---|---|
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: GOLD / 前処理 - タイプ: GLOW DISCHARGE |
| 凍結 | 凍結剤: ETHANE / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 平均電子線量: 46.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: SPOT SCAN / 撮影モード: BRIGHT FIELD 最大 デフォーカス(公称値): 2.8000000000000003 µm 最小 デフォーカス(公称値): 0.5 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期モデル | モデルのタイプ: INSILICO MODEL |
|---|---|
| 最終 再構成 | 解像度のタイプ: BY AUTHOR / 解像度: 4.1 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 使用した粒子像数: 56245 |
| 初期 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
| 最終 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
 ムービー
ムービー コントローラー
コントローラー










 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)