+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | CryoEM structure of nucleotide-free GroEL-Rubisco. | |||||||||
 マップデータ マップデータ | CryoEM structure of GroEL bound to non-native Rubisco. | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | GroEL / Rubisco / Chaperone / complex | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報GroEL-GroES complex / chaperonin ATPase / virion assembly / : / isomerase activity / ATP-dependent protein folding chaperone / response to radiation / unfolded protein binding / protein folding / response to heat ...GroEL-GroES complex / chaperonin ATPase / virion assembly / : / isomerase activity / ATP-dependent protein folding chaperone / response to radiation / unfolded protein binding / protein folding / response to heat / protein refolding / magnesium ion binding / ATP hydrolysis activity / ATP binding / identical protein binding / membrane / cytosol 類似検索 - 分子機能 | |||||||||
| 生物種 |  | |||||||||
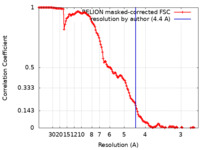
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 4.4 Å | |||||||||
 データ登録者 データ登録者 | Gardner S / Saibil HR | |||||||||
| 資金援助 |  英国, 1件 英国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Proc Natl Acad Sci U S A / 年: 2023 ジャーナル: Proc Natl Acad Sci U S A / 年: 2023タイトル: Structural basis of substrate progression through the bacterial chaperonin cycle. 著者: Scott Gardner / Michele C Darrow / Natalya Lukoyanova / Konstantinos Thalassinos / Helen R Saibil /  要旨: The bacterial chaperonin GroEL-GroES promotes protein folding through ATP-regulated cycles of substrate protein binding, encapsulation, and release. Here, we have used cryoEM to determine structures ...The bacterial chaperonin GroEL-GroES promotes protein folding through ATP-regulated cycles of substrate protein binding, encapsulation, and release. Here, we have used cryoEM to determine structures of GroEL, GroEL-ADP·BeF, and GroEL-ADP·AlF-GroES all complexed with the model substrate Rubisco. Our structures provide a series of snapshots that show how the conformation and interactions of non-native Rubisco change as it proceeds through the GroEL-GroES reaction cycle. We observe specific charged and hydrophobic GroEL residues forming strong initial contacts with non-native Rubisco. Binding of ATP or ADP·BeF to GroEL-Rubisco results in the formation of an intermediate GroEL complex displaying striking asymmetry in the ATP/ADP·BeF-bound ring. In this ring, four GroEL subunits bind Rubisco and the other three are in the GroES-accepting conformation, suggesting how GroEL can recruit GroES without releasing bound substrate. Our cryoEM structures of stalled GroEL-ADP·AlF-Rubisco-GroES complexes show Rubisco folding intermediates interacting with GroEL-GroES via different sets of residues. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_15939.map.gz emd_15939.map.gz | 120.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-15939-v30.xml emd-15939-v30.xml emd-15939.xml emd-15939.xml | 16.2 KB 16.2 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_15939_fsc.xml emd_15939_fsc.xml | 13.6 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_15939.png emd_15939.png | 108.6 KB | ||
| マスクデータ |  emd_15939_msk_1.map emd_15939_msk_1.map | 216 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-15939.cif.gz emd-15939.cif.gz | 5.7 KB | ||
| その他 |  emd_15939_half_map_1.map.gz emd_15939_half_map_1.map.gz emd_15939_half_map_2.map.gz emd_15939_half_map_2.map.gz | 172.5 MB 172.3 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-15939 http://ftp.pdbj.org/pub/emdb/structures/EMD-15939 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-15939 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-15939 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_15939_validation.pdf.gz emd_15939_validation.pdf.gz | 959.9 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_15939_full_validation.pdf.gz emd_15939_full_validation.pdf.gz | 959.5 KB | 表示 | |
| XML形式データ |  emd_15939_validation.xml.gz emd_15939_validation.xml.gz | 20.9 KB | 表示 | |
| CIF形式データ |  emd_15939_validation.cif.gz emd_15939_validation.cif.gz | 27.6 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-15939 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-15939 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-15939 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-15939 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8ba7MC  8ba8C  8ba9C M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_15939.map.gz / 形式: CCP4 / 大きさ: 216 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_15939.map.gz / 形式: CCP4 / 大きさ: 216 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | CryoEM structure of GroEL bound to non-native Rubisco. | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.34 Å | ||||||||||||||||||||||||||||||||||||
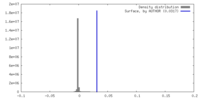
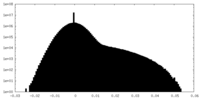
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_15939_msk_1.map emd_15939_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
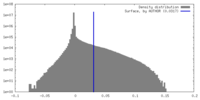
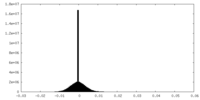
| 密度ヒストグラム |
-ハーフマップ: CryoEM structure of GroEL bound to non-native Rubisco.
| ファイル | emd_15939_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | CryoEM structure of GroEL bound to non-native Rubisco. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
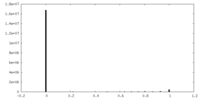
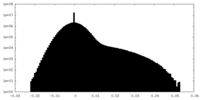
| 密度ヒストグラム |
-ハーフマップ: CryoEM structure of GroEL bound to non-native Rubisco.
| ファイル | emd_15939_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | CryoEM structure of GroEL bound to non-native Rubisco. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
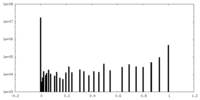
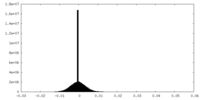
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : GroEL
| 全体 | 名称: GroEL |
|---|---|
| 要素 |
|
-超分子 #1: GroEL
| 超分子 | 名称: GroEL / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 802 KDa |
-分子 #1: Chaperonin GroEL
| 分子 | 名称: Chaperonin GroEL / タイプ: protein_or_peptide / ID: 1 / コピー数: 14 / 光学異性体: LEVO / EC番号: chaperonin ATPase |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 57.260504 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: AAKDVKFGND ARVKMLRGVN VLADAVKVTL GPKGRNVVLD KSFGAPTITK DGVSVAREIE LEDKFENMGA QMVKEVASKA NDAAGDGTT TATVLAQAII TEGLKAVAAG MNPMDLKRGI DKAVTAAVEE LKALSVPCSD SKAIAQVGTI SANSDETVGK L IAEAMDKV ...文字列: AAKDVKFGND ARVKMLRGVN VLADAVKVTL GPKGRNVVLD KSFGAPTITK DGVSVAREIE LEDKFENMGA QMVKEVASKA NDAAGDGTT TATVLAQAII TEGLKAVAAG MNPMDLKRGI DKAVTAAVEE LKALSVPCSD SKAIAQVGTI SANSDETVGK L IAEAMDKV GKEGVITVED GTGLQDELDV VEGMQFDRGY LSPYFINKPE TGAVELESPF ILLADKKISN IREMLPVLEA VA KAGKPLL IIAEDVEGEA LATLVVNTMR GIVKVAAVKA PGFGDRRKAM LQDIATLTGG TVISEEIGME LEKATLEDLG QAK RVVINK DTTTIIDGVG EEAAIQGRVA QIRQQIEEAT SDYDREKLQE RVAKLAGGVA VIKVGAATEV EMKEKKARVE DALH ATRAA VEEGVVAGGG VALIRVASKL ADLRGQNEDQ NVGIKVALRA MEAPLRQIVL NCGEEPSVVA NTVKGGDGNY GYNAA TEEY GNMIDMGILD PTKVTRSALQ YAASVAGLMI TTECMVTDLP KNDAADLGAA GGMGGMGGMG GMM UniProtKB: Chaperonin GroEL |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 2.7 mg/mL |
|---|---|
| 緩衝液 | pH: 7.5 |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 80 % / チャンバー内温度: 293 K / 装置: SPOTITON 詳細: The grid was prepared using a chameleon (SPT Labtech).. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 BASE (4k x 4k) 撮影したグリッド数: 2 / 平均電子線量: 40.2 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 70.0 µm / 照射モード: SPOT SCAN / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 3.0 µm 最小 デフォーカス(公称値): 1.4000000000000001 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー











 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)