+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Open conformation of the complex of DNA ligase I on PCNA and DNA in the presence of ATP | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | DNA / Replication / Complex / Ligase / PCNA / Ligation / Okazaki fragment maturation | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報Okazaki fragment processing involved in mitotic DNA replication / DNA ligase activity / DNA ligase (ATP) / DNA ligase (ATP) activity / positive regulation of deoxyribonuclease activity / dinucleotide insertion or deletion binding / PCNA-p21 complex / mitotic telomere maintenance via semi-conservative replication / Regulation of MITF-M-dependent genes involved in DNA replication, damage repair and senescence / purine-specific mismatch base pair DNA N-glycosylase activity ...Okazaki fragment processing involved in mitotic DNA replication / DNA ligase activity / DNA ligase (ATP) / DNA ligase (ATP) activity / positive regulation of deoxyribonuclease activity / dinucleotide insertion or deletion binding / PCNA-p21 complex / mitotic telomere maintenance via semi-conservative replication / Regulation of MITF-M-dependent genes involved in DNA replication, damage repair and senescence / purine-specific mismatch base pair DNA N-glycosylase activity / nuclear lamina / positive regulation of DNA-directed DNA polymerase activity / Polymerase switching / MutLalpha complex binding / Telomere C-strand (Lagging Strand) Synthesis / Processive synthesis on the lagging strand / PCNA complex / lagging strand elongation / Removal of the Flap Intermediate / Processive synthesis on the C-strand of the telomere / Polymerase switching on the C-strand of the telomere / Mismatch repair (MMR) directed by MSH2:MSH3 (MutSbeta) / Mismatch repair (MMR) directed by MSH2:MSH6 (MutSalpha) / Removal of the Flap Intermediate from the C-strand / Transcription of E2F targets under negative control by DREAM complex / replisome / response to L-glutamate / DNA biosynthetic process / response to dexamethasone / histone acetyltransferase binding / DNA polymerase processivity factor activity / leading strand elongation / G1/S-Specific Transcription / Early Phase of HIV Life Cycle / nuclear replication fork / replication fork processing / SUMOylation of DNA replication proteins / POLB-Dependent Long Patch Base Excision Repair / anatomical structure morphogenesis / PCNA-Dependent Long Patch Base Excision Repair / response to cadmium ion / translesion synthesis / mismatch repair / estrous cycle / cyclin-dependent protein kinase holoenzyme complex / Translesion synthesis by REV1 / Translesion synthesis by POLK / base-excision repair, gap-filling / DNA polymerase binding / Translesion synthesis by POLI / Gap-filling DNA repair synthesis and ligation in GG-NER / epithelial cell differentiation / liver regeneration / positive regulation of DNA repair / TP53 Regulates Transcription of Genes Involved in G2 Cell Cycle Arrest / Termination of translesion DNA synthesis / positive regulation of DNA replication / replication fork / Recognition of DNA damage by PCNA-containing replication complex / nuclear estrogen receptor binding / Translesion Synthesis by POLH / male germ cell nucleus / HDR through Homologous Recombination (HRR) / Dual Incision in GG-NER / base-excision repair / receptor tyrosine kinase binding / Dual incision in TC-NER / Gap-filling DNA repair synthesis and ligation in TC-NER / cellular response to hydrogen peroxide / cellular response to xenobiotic stimulus / cellular response to UV / E3 ubiquitin ligases ubiquitinate target proteins / response to estradiol / heart development / chromatin organization / DNA recombination / damaged DNA binding / chromosome, telomeric region / nuclear body / cell division / DNA repair / intracellular membrane-bounded organelle / centrosome / chromatin binding / chromatin / protein-containing complex binding / enzyme binding / negative regulation of transcription by RNA polymerase II / DNA binding / extracellular exosome / nucleoplasm / ATP binding / metal ion binding / identical protein binding / nucleus 類似検索 - 分子機能 | |||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 4.2 Å | |||||||||
 データ登録者 データ登録者 | Blair K / Tehseen M / Raducanu VS / Shahid T / Lancey C / Cruehet R / Hamdan S / De Biasio A | |||||||||
| 資金援助 |  英国, 1件 英国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2022 ジャーナル: Nat Commun / 年: 2022タイトル: Mechanism of human Lig1 regulation by PCNA in Okazaki fragment sealing. 著者: Kerry Blair / Muhammad Tehseen / Vlad-Stefan Raducanu / Taha Shahid / Claudia Lancey / Fahad Rashid / Ramon Crehuet / Samir M Hamdan / Alfredo De Biasio /    要旨: During lagging strand synthesis, DNA Ligase 1 (Lig1) cooperates with the sliding clamp PCNA to seal the nicks between Okazaki fragments generated by Pol δ and Flap endonuclease 1 (FEN1). We present ...During lagging strand synthesis, DNA Ligase 1 (Lig1) cooperates with the sliding clamp PCNA to seal the nicks between Okazaki fragments generated by Pol δ and Flap endonuclease 1 (FEN1). We present several cryo-EM structures combined with functional assays, showing that human Lig1 recruits PCNA to nicked DNA using two PCNA-interacting motifs (PIPs) located at its disordered N-terminus (PIP) and DNA binding domain (PIP). Once Lig1 and PCNA assemble as two-stack rings encircling DNA, PIP is released from PCNA and only PIP is required for ligation to facilitate the substrate handoff from FEN1. Consistently, we observed that PCNA forms a defined complex with FEN1 and nicked DNA, and it recruits Lig1 to an unoccupied monomer creating a toolbelt that drives the transfer of DNA to Lig1. Collectively, our results provide a structural model on how PCNA regulates FEN1 and Lig1 during Okazaki fragments maturation. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_15921.map.gz emd_15921.map.gz | 3.8 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-15921-v30.xml emd-15921-v30.xml emd-15921.xml emd-15921.xml | 26.5 KB 26.5 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_15921_fsc.xml emd_15921_fsc.xml | 7.9 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_15921.png emd_15921.png | 80.5 KB | ||
| Filedesc metadata |  emd-15921.cif.gz emd-15921.cif.gz | 6.8 KB | ||
| その他 |  emd_15921_additional_1.map.gz emd_15921_additional_1.map.gz emd_15921_additional_2.map.gz emd_15921_additional_2.map.gz emd_15921_half_map_1.map.gz emd_15921_half_map_1.map.gz emd_15921_half_map_2.map.gz emd_15921_half_map_2.map.gz | 508.3 KB 3.3 MB 31.4 MB 31.4 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-15921 http://ftp.pdbj.org/pub/emdb/structures/EMD-15921 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-15921 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-15921 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_15921_validation.pdf.gz emd_15921_validation.pdf.gz | 774.8 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_15921_full_validation.pdf.gz emd_15921_full_validation.pdf.gz | 774.4 KB | 表示 | |
| XML形式データ |  emd_15921_validation.xml.gz emd_15921_validation.xml.gz | 13.4 KB | 表示 | |
| CIF形式データ |  emd_15921_validation.cif.gz emd_15921_validation.cif.gz | 19.3 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-15921 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-15921 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-15921 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-15921 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8b8tMC  7qnzC  7qo1C C: 同じ文献を引用 ( M: このマップから作成された原子モデル |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_15921.map.gz / 形式: CCP4 / 大きさ: 40.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_15921.map.gz / 形式: CCP4 / 大きさ: 40.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.086 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-追加マップ: #2
| ファイル | emd_15921_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
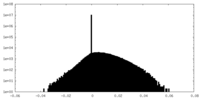
| 密度ヒストグラム |
-追加マップ: #1
| ファイル | emd_15921_additional_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
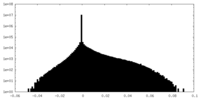
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_15921_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_15921_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : complex of DNA ligase I on PCNA and DNA in the presence of ATP
| 全体 | 名称: complex of DNA ligase I on PCNA and DNA in the presence of ATP |
|---|---|
| 要素 |
|
-超分子 #1: complex of DNA ligase I on PCNA and DNA in the presence of ATP
| 超分子 | 名称: complex of DNA ligase I on PCNA and DNA in the presence of ATP タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
-分子 #1: DNA ligase 1
| 分子 | 名称: DNA ligase 1 / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO / EC番号: DNA ligase (ATP) |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 29.720342 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: DPSGYNPAKN NYHPVEDACW KPGQKVPYLA VARTFEKIEE VSARLRMVET LSNLLRSVVA LSPPDLLPVL YLSLNHLGPP QQGLELGVG DGVLLKAVAQ ATGRQLESVR AEAAEKGDVG LVAENSRSTQ RLMLPPPPLT ASGVFSKFRD IARLTGSAST A KKIDIIKG ...文字列: DPSGYNPAKN NYHPVEDACW KPGQKVPYLA VARTFEKIEE VSARLRMVET LSNLLRSVVA LSPPDLLPVL YLSLNHLGPP QQGLELGVG DGVLLKAVAQ ATGRQLESVR AEAAEKGDVG LVAENSRSTQ RLMLPPPPLT ASGVFSKFRD IARLTGSAST A KKIDIIKG LFVACRHSEA RFIARSLSGR LRLGLAEQSV LAALSQAVSL TPPGQEFPPA MVDAGKGKTA EARKTWLEEQ GM ILKQTFC EVPDLDRIIP VLLEHGLERL PEHCKLS UniProtKB: DNA ligase 1 |
-分子 #2: Proliferating cell nuclear antigen
| 分子 | 名称: Proliferating cell nuclear antigen / タイプ: protein_or_peptide / ID: 2 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 28.441504 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: GPHMFEARLV QGSILKKVLE ALKDLINEAC WDISSSGVNL QSMDSSHVSL VQLTLRSEGF DTYRCDRNLA MGVNLTSMSK ILKCAGNED IITLRAEDNA DTLALVFEAP NQEKVSDYEM KLMDLDVEQL GIPEQEYSCV VKMPSGEFAR ICRDLSHIGD A VVISCAKD ...文字列: GPHMFEARLV QGSILKKVLE ALKDLINEAC WDISSSGVNL QSMDSSHVSL VQLTLRSEGF DTYRCDRNLA MGVNLTSMSK ILKCAGNED IITLRAEDNA DTLALVFEAP NQEKVSDYEM KLMDLDVEQL GIPEQEYSCV VKMPSGEFAR ICRDLSHIGD A VVISCAKD GVKFSASGEL GNGNIKLSQT SNVDKEEEAV TIEMNEPVQL TFALRYLNFF TKATPLSSTV TLSMSADVPL VV EYKIADM GHLKYYLAPK I UniProtKB: Proliferating cell nuclear antigen |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 構成要素:
| ||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 温度 | 最低: 77.0 K / 最高: 77.0 K |
| 特殊光学系 | エネルギーフィルター - 名称: GIF Bioquantum / エネルギーフィルター - スリット幅: 20 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) デジタル化 - サイズ - 横: 5760 pixel / デジタル化 - サイズ - 縦: 4092 pixel / 撮影したグリッド数: 1 / 平均露光時間: 2.0 sec. / 平均電子線量: 18.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 50.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 1.0 µm / 倍率(公称値): 105000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | プロトコル: RIGID BODY FIT |
|---|---|
| 得られたモデル |  PDB-8b8t: |
 ムービー
ムービー コントローラー
コントローラー



























 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)