+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Rosellinia necatrix megabirnavirus 1-W779 full capsid with Crown protein | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | viruses / dsRNA / capsid / cryo-EM / fungus / Megabirnaviridae / mycoviruses / VIRUS | |||||||||
| 機能・相同性 | membrane / Uncharacterized protein 機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |  Rosellinia necatrix megabirnavirus 1/W779 (ウイルス) Rosellinia necatrix megabirnavirus 1/W779 (ウイルス) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.3 Å | |||||||||
 データ登録者 データ登録者 | Wang H / Okamoto K / Miyazaki N / Suzuki N | |||||||||
| 資金援助 |  スウェーデン, スウェーデン,  日本, 2件 日本, 2件
| |||||||||
 引用 引用 |  ジャーナル: PLoS Pathog / 年: 2023 ジャーナル: PLoS Pathog / 年: 2023タイトル: Capsid structure of a fungal dsRNA megabirnavirus reveals its previously unidentified surface architecture. 著者: Han Wang / Lakha Salaipeth / Naoyuki Miyazaki / Nobuhiro Suzuki / Kenta Okamoto /   要旨: Rosellinia necatrix megabirnavirus 1-W779 (RnMBV1) is a non-enveloped icosahedral double-stranded (ds)RNA virus that infects the ascomycete fungus Rosellinia necatrix, a causative agent that induces ...Rosellinia necatrix megabirnavirus 1-W779 (RnMBV1) is a non-enveloped icosahedral double-stranded (ds)RNA virus that infects the ascomycete fungus Rosellinia necatrix, a causative agent that induces a lethal plant disease white root rot. Herein, we have first resolved the atomic structure of the RnMBV1 capsid at 3.2 Å resolution using cryo-electron microscopy (cryo-EM) single-particle analysis. Compared with other non-enveloped icosahedral dsRNA viruses, the RnMBV1 capsid protein structure exhibits an extra-long C-terminal arm and a surface protrusion domain. In addition, the previously unrecognized crown proteins are identified in a symmetry-expanded cryo-EM model and are present over the 3-fold axes. These exclusive structural features of the RnMBV1 capsid could have been acquired for playing essential roles in transmission and/or particle assembly of the megabirnaviruses. Our findings, therefore, will reinforce the understanding of how the structural and molecular machineries of the megabirnaviruses influence the virulence of the disease-related ascomycete fungus. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_15859.map.gz emd_15859.map.gz | 689.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-15859-v30.xml emd-15859-v30.xml emd-15859.xml emd-15859.xml | 14.4 KB 14.4 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_15859.png emd_15859.png | 236.3 KB | ||
| Filedesc metadata |  emd-15859.cif.gz emd-15859.cif.gz | 5.9 KB | ||
| その他 |  emd_15859_half_map_1.map.gz emd_15859_half_map_1.map.gz emd_15859_half_map_2.map.gz emd_15859_half_map_2.map.gz | 598.9 MB 599.1 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-15859 http://ftp.pdbj.org/pub/emdb/structures/EMD-15859 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-15859 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-15859 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_15859_validation.pdf.gz emd_15859_validation.pdf.gz | 1.2 MB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_15859_full_validation.pdf.gz emd_15859_full_validation.pdf.gz | 1.2 MB | 表示 | |
| XML形式データ |  emd_15859_validation.xml.gz emd_15859_validation.xml.gz | 20.2 KB | 表示 | |
| CIF形式データ |  emd_15859_validation.cif.gz emd_15859_validation.cif.gz | 24.1 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-15859 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-15859 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-15859 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-15859 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_15859.map.gz / 形式: CCP4 / 大きさ: 744.3 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_15859.map.gz / 形式: CCP4 / 大きさ: 744.3 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.12 Å | ||||||||||||||||||||||||||||||||||||
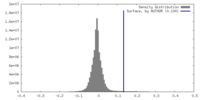
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #1
| ファイル | emd_15859_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
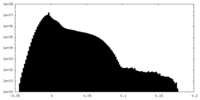
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_15859_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
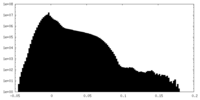
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Rosellinia necatrix megabirnavirus 1/W779
| 全体 | 名称:  Rosellinia necatrix megabirnavirus 1/W779 (ウイルス) Rosellinia necatrix megabirnavirus 1/W779 (ウイルス) |
|---|---|
| 要素 |
|
-超分子 #1: Rosellinia necatrix megabirnavirus 1/W779
| 超分子 | 名称: Rosellinia necatrix megabirnavirus 1/W779 / タイプ: virus / ID: 1 / 親要素: 0 / 含まれる分子: all / NCBI-ID: 658904 / 生物種: Rosellinia necatrix megabirnavirus 1/W779 / ウイルスタイプ: VIRION / ウイルス・単離状態: STRAIN / ウイルス・エンベロープ: No / ウイルス・中空状態: No |
|---|
-分子 #1: RnMBV1 Crown protein
| 分子 | 名称: RnMBV1 Crown protein / タイプ: protein_or_peptide / ID: 1 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Rosellinia necatrix megabirnavirus 1/W779 (ウイルス) Rosellinia necatrix megabirnavirus 1/W779 (ウイルス)株: isolate -/Japan/W779/2001 |
| 分子量 | 理論値: 151.8835 KDa |
| 配列 | 文字列: MGITYRDAQI FSACVEALSA RNNRITLTSF PLTAGQGQAP TATPAWYPVD LFVADATAVY GRRQLFAWTV DKVRPTRNVA FVTDRVAMD FSAALLSLMA ELEAVAPDVY AAIHGGATPG ADLGDRITQL ENRRVGCLAY VMATVVRAPI THNVRSFSAM L ASDPQAHA ...文字列: MGITYRDAQI FSACVEALSA RNNRITLTSF PLTAGQGQAP TATPAWYPVD LFVADATAVY GRRQLFAWTV DKVRPTRNVA FVTDRVAMD FSAALLSLMA ELEAVAPDVY AAIHGGATPG ADLGDRITQL ENRRVGCLAY VMATVVRAPI THNVRSFSAM L ASDPQAHA ALLAYLTPNS AGQLDGAPIY FRRSDVDLRN NHLALHAEVV PGLPNMVPLT KAMVEVALAN VEWWSDPLGY DS LTSFGGL ELLSLCDALA VCELSVAYGL KESGYCYLRF AGGCPLAEVI LARLGYNPPL GVAVGWALYN GIKLDWYSKV ISV GHNMRL HVCDTAGEAN ACLIDVLTGE YDGMPVGGVD TVSCWVEQLD LLAAAAGVGR NLSNLHCGVQ TPPRTINTTR RRLL ASLVR TLIADPTLTD EELLHGAVRG TLNGLPRDRA LWRCLQVVNT TVREFLAQDL DAMVRDRREC TTYASRAAFA ERCAM SGNA SGLVGRQYSD MPAALEGEAR ACGLSAIDAI EIVRVVASGE PIRVLLDQHG RPATRPNGRL TADELRRCRP LVVGQG GQV GFLPFVPFIV GGVGATVAAA SGLALATFAT VTGAGAVALG GLGLGAGVAA LSITVGQLSY QVTRRALTTI LPGGREF GL DDLTRVLGGM VGRYISFVDT WFAHGRGDVF QETDAVPAGT VVFVLPNIEY EVLELRERAL GRWSTLLVTT PNGVIAMR A NGALPLRVVD AREAGQTFEW STAARRRFTR AQANAINMMV TASKRVPGLK GSIDAAPSQG TGGSGTDLAG ILQRLSALE QTSVPRAEFD ALQGRVAACE AKITELEADR VPRIDFTELR DRVHHIDGIG LSCLAHLARD LGITVPHNVR TFRQMRANVG EVIWARFVD AVAESFSPMG GRPIFVRTDP AQPRNNHVSL VDEPTTTGFN GTVTPAMRRL TVADLTGDLV DTEWFSWTPY D ASGPLGGT IEGIEAYLTD FTSKLKAELE ATPTRTELGV AVGTRAPPLS DRLAAVERVI GMQEGNQVWR SNELRELWVA ID SIVTGRG QREFTTATIK WPAAFPSAVA TAGRSFGQPG LAGYGELCTL ARQLNALVAG VRNGVVSGMT RNGAGVLQLS TIS SATGNL TSDQQAVLRA CFFPATPRVG EYQIVYPVGG TMGLTRVDPS TNSSIGQYTR ESLVAARNAM PRFAVHTTTP DTVG VAWDN QSAAGLPMGA APVLTVSVNQ LSGVPVTEAD KQRWDAKQDK FKIVNTDDRV AALSWVDSVD GFAAPGSDML LDYQA PAGT GSLPFGSKYA MAVAIGGSLG SQLSEAQVSA ARVVLGNGVW RDAVIDVLRK LHNVMYGGKY GRIDDIAAMR SYLNDG TGL LPGSEPIVDV GGAEGNACAR ATILLRGFSS TMVGVDLKIQ MLVELYGAEP ATAALLYRGW TMQ UniProtKB: Uncharacterized protein |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: FEI FALCON III (4k x 4k) 平均電子線量: 48.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: SPOT SCAN / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.75 µm / 最小 デフォーカス(公称値): 1.0 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期モデル | モデルのタイプ: NONE |
|---|---|
| 最終 再構成 | 解像度のタイプ: BY AUTHOR / 解像度: 3.3 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 使用した粒子像数: 244609 |
| 初期 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
| 最終 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
 ムービー
ムービー コントローラー
コントローラー








 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)