+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Low resolution 3D reconstruction of ATG2A from cryo-EM | ||||||||||||
 マップデータ マップデータ | |||||||||||||
 試料 試料 |
| ||||||||||||
 キーワード キーワード | Lipid transfer protein / autophagy / LIPID TRANSPORT | ||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報phagophore / lipid transfer activity / organelle membrane contact site / glycophagy / positive regulation of autophagosome assembly / phagophore assembly site membrane / autophagy of mitochondrion / pexophagy / piecemeal microautophagy of the nucleus / phosphatidylinositol-3-phosphate binding ...phagophore / lipid transfer activity / organelle membrane contact site / glycophagy / positive regulation of autophagosome assembly / phagophore assembly site membrane / autophagy of mitochondrion / pexophagy / piecemeal microautophagy of the nucleus / phosphatidylinositol-3-phosphate binding / phagophore assembly site / reticulophagy / autophagosome assembly / protein-membrane adaptor activity / lipid droplet / positive regulation of autophagy / endoplasmic reticulum membrane 類似検索 - 分子機能 | ||||||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | ||||||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 14.7 Å | ||||||||||||
 データ登録者 データ登録者 | Cherepanov P / Chiduza GN / Pye VE / van Vliet AR / Tooze SA | ||||||||||||
| 資金援助 |  英国, European Union, 3件 英国, European Union, 3件
| ||||||||||||
 引用 引用 |  ジャーナル: Mol Cell / 年: 2022 ジャーナル: Mol Cell / 年: 2022タイトル: ATG9A and ATG2A form a heteromeric complex essential for autophagosome formation. 著者: Alexander R van Vliet / George N Chiduza / Sarah L Maslen / Valerie E Pye / Dhira Joshi / Stefano De Tito / Harold B J Jefferies / Evangelos Christodoulou / Chloë Roustan / Emma Punch / ...著者: Alexander R van Vliet / George N Chiduza / Sarah L Maslen / Valerie E Pye / Dhira Joshi / Stefano De Tito / Harold B J Jefferies / Evangelos Christodoulou / Chloë Roustan / Emma Punch / Javier H Hervás / Nicola O'Reilly / J Mark Skehel / Peter Cherepanov / Sharon A Tooze /   要旨: ATG9A and ATG2A are essential core members of the autophagy machinery. ATG9A is a lipid scramblase that allows equilibration of lipids across a membrane bilayer, whereas ATG2A facilitates lipid flow ...ATG9A and ATG2A are essential core members of the autophagy machinery. ATG9A is a lipid scramblase that allows equilibration of lipids across a membrane bilayer, whereas ATG2A facilitates lipid flow between tethered membranes. Although both have been functionally linked during the formation of autophagosomes, the molecular details and consequences of their interaction remain unclear. By combining data from peptide arrays, crosslinking, and hydrogen-deuterium exchange mass spectrometry together with cryoelectron microscopy, we propose a molecular model of the ATG9A-2A complex. Using this integrative structure modeling approach, we identify several interfaces mediating ATG9A-2A interaction that would allow a direct transfer of lipids from ATG2A into the lipid-binding perpendicular branch of ATG9A. Mutational analyses combined with functional activity assays demonstrate their importance for autophagy, thereby shedding light on this protein complex at the heart of autophagy. | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_15605.map.gz emd_15605.map.gz | 1.2 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-15605-v30.xml emd-15605-v30.xml emd-15605.xml emd-15605.xml | 16.9 KB 16.9 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_15605_fsc.xml emd_15605_fsc.xml | 5.1 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_15605.png emd_15605.png | 75.1 KB | ||
| マスクデータ |  emd_15605_msk_1.map emd_15605_msk_1.map | 10.5 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-15605.cif.gz emd-15605.cif.gz | 6.2 KB | ||
| その他 |  emd_15605_half_map_1.map.gz emd_15605_half_map_1.map.gz emd_15605_half_map_2.map.gz emd_15605_half_map_2.map.gz | 7.9 MB 7.9 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-15605 http://ftp.pdbj.org/pub/emdb/structures/EMD-15605 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-15605 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-15605 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_15605_validation.pdf.gz emd_15605_validation.pdf.gz | 187.5 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_15605_full_validation.pdf.gz emd_15605_full_validation.pdf.gz | 187.1 KB | 表示 | |
| XML形式データ |  emd_15605_validation.xml.gz emd_15605_validation.xml.gz | 503 B | 表示 | |
| CIF形式データ |  emd_15605_validation.cif.gz emd_15605_validation.cif.gz | 373 B | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-15605 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-15605 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-15605 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-15605 | HTTPS FTP |
-関連構造データ
| 関連構造データ | C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_15605.map.gz / 形式: CCP4 / 大きさ: 10.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_15605.map.gz / 形式: CCP4 / 大きさ: 10.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 2.52 Å | ||||||||||||||||||||||||||||||||||||
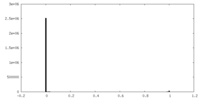
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_15605_msk_1.map emd_15605_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
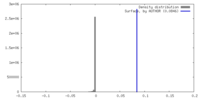
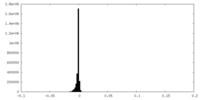
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_15605_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
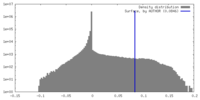
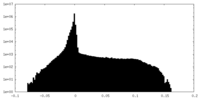
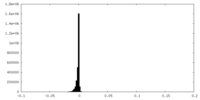
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_15605_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
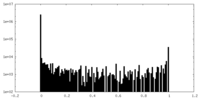
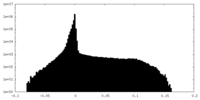
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Purified ATG2A
| 全体 | 名称: Purified ATG2A |
|---|---|
| 要素 |
|
-超分子 #1: Purified ATG2A
| 超分子 | 名称: Purified ATG2A / タイプ: organelle_or_cellular_component / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
-分子 #1: Autophagy-related protein 2 homolog A
| 分子 | 名称: Autophagy-related protein 2 homolog A / タイプ: protein_or_peptide / ID: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MDYKDDDDKD YKDDDDKDYK DDDDKGSGAG AGAGAILNSR VSGLRSRMSR WLWPWSNCVK ERVCRYLLHH YLGHFFQEHL SLDQLSLDLY KGSVALRDIH LEIWSVNEVL ESMESPLELV EGFVGSIEVA VPWAALLTDH CTVRVSGLQL TLQPRRGPAP GAADSQSWAS ...文字列: MDYKDDDDKD YKDDDDKDYK DDDDKGSGAG AGAGAILNSR VSGLRSRMSR WLWPWSNCVK ERVCRYLLHH YLGHFFQEHL SLDQLSLDLY KGSVALRDIH LEIWSVNEVL ESMESPLELV EGFVGSIEVA VPWAALLTDH CTVRVSGLQL TLQPRRGPAP GAADSQSWAS CMTTSLQLAQ ECLRDGLPEP SEPPQPLEGL EMFAQTIETV LRRIKVTFLD TVVRVEHSPG DGERGVAVEV RVQRLEYCDE AVRDPSQAPP VDVHQPPAFL HKLLQLAGVR LHYEELPAQE EPPEPPLQIG SCSGYMELMV KLKQNEAFPG PKLEVAGQLG SLHLLLTPRQ LQQLQELLSA VSLTDHEGLA DKLNKSRPLG AEDLWLIEQD LNQQLQAGAV AEPLSPDPLT NPLLNLDNTD LFFSMAGLTS SVASALSELS LSDVDLASSV RSDMASRRLS AQAHPAGKMA PNPLLDTMRP DSLLKMTLGG VTLTLLQTSA PSSGPPDLAT HFFTEFDATK DGPFGSRDFH HLRPRFQRAC PCSHVRLTGT AVQLSWELRT GSRGRRTTSM EVHFGQLEVL ECLWPRGTSE PEYTEILTFP GTLGSQASAR PCAHLRHTQI LRRVPKSRPR RSVACHCHSE LALDLANFQA DVELGALDRL AALLRLATVP AEPPAGLLTE PLPAMEQQTV FRLSAPRATL RLRFPIADLR PEPDPWAGQA VRAEQLRLEL SEPQFRSELS SGPGPPVPTH LELTCSDLHG IYEDGGKPPV PCLRVSKALD PKSTGRKYFL PQVVVTVNPQ SSSTQWEVAP EKGEELELSV ESPCELREPE PSPFSSKRTM YETEEMVIPG DPEEMRTFQS RTLALSRCSL EVILPSVHIF LPSKEVYESI YNRINNDLLM WEPADLLPTP DPAAQPSGFP GPSGFWHDSF KMCKSAFKLA NCFDLTPDSD SDDEDAHFFS VGASGGPQAA APEAPSLHLQ STFSTLVTVL KGRITALCET KDEGGKRLEA VHGELVLDME HGTLFSVSQY CGQPGLGYFC LEAEKATLYH RAAVDDYPLP SHLDLPSFAP PAQLAPTIYP SEEGVTERGA SGRKGQGRGP HMLSTAVRIH LDPHKNVKEF LVTLRLHKAT LRHYMALPEQ SWHSQLLEFL DVLDDPVLGY LPPTVITILH THLFSCSVDY RPLYLPVRVL ITAETFTLSS NIIMDTSTFL LRFILDDSAL YLSDKCEVET LDLRRDYVCV LDVDLLELVI KTWKGSTEGK LSQPLFELRC SNNVVHVHSC ADSCALLVNL LQYVMSTGDL HPPPRPPSPT EIAGQKLSES PASLPSCPPV ETALINQRDL ADALLDTERS LRELAQPSGG HLPQASPISV YLFPGERSGA PPPSPPVGGP AGSLGSCSEE KEDEREEEGD GDTLDSDEFC ILDAPGLGIP PRDGEPVVTQ LHPGPIVVRD GYFSRPIGST DLLRAPAHFP VPSTRVVLRE VSLVWHLYGG RDFGPHPGHR ARTGLSGPRS SPSRCSGPNR PQNSWRTQGG SGRQHHVLME IQLSKVSFQH EVYPAEPATG PAAPSQELEE RPLSRQVFIV QELEVRDRLA SSQINKFLYL HTSERMPRRA HSNMLTIKAL HVAPTTNLGG PECCLRVSLM PLRLNVDQDA LFFLKDFFTS LVAGINPVVP GETSAEARPE TRAQPSSPLE GQAEGVETTG SQEAPGGGHS PSPPDQQPIY FREFRFTSEV PIWLDYHGKH VTMDQVGTFA GLLIGLAQLN CSELKLKRLC CRHGLLGVDK VLGYALNEWL QDIRKNQLPG LLGGVGPMHS VVQLFQGFRD LLWLPIEQYR KDGRLMRGLQ RGAASFGSST ASAALELSNR LVQAIQATAE TVYDILSPAA PVSRSLQDKR SARRLRRGQQ PADLREGVAK AYDTVREGIL DTAQTICDVA SRGHEQKGLT GAVGGVIRQL PPTVVKPLIL ATEATSSLLG GMRNQIVPDA HKDHALKWRS DSAQDNSAVD GTAGPGSTGS R UniProtKB: Autophagy-related protein 2 homolog A |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.56 mg/mL |
|---|---|
| 緩衝液 | pH: 8 / 詳細: 50 mM HEPES, 200 mM NaCl, 1 mM TCEP |
| グリッド | モデル: C-flat-1.2/1.3 / 材質: GOLD / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 295.15 K / 装置: FEI VITROBOT MARK IV |
| 詳細 | Grids prepared straight after size exclusion elution. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TALOS ARCTICA |
|---|---|
| 撮影 | フィルム・検出器のモデル: FEI FALCON III (4k x 4k) 検出モード: SUPER-RESOLUTION / 平均電子線量: 8.0 e/Å2 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 3.5 µm / 最小 デフォーカス(公称値): 1.0 µm |
| 試料ステージ | ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Talos Arctica / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー




 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)