+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-1536 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | ESCRT-III subunits CHMP2A and CHMP3 assemble in vitro into helical tubular structures | |||||||||
 マップデータ マップデータ | ESCRT-III subunits CHMP2A and CHMP3 assemble in vitro into helical tubular structures | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | helical reconstruction | |||||||||
| 生物種 |  | |||||||||
| 手法 | らせん対称体再構成法 / クライオ電子顕微鏡法 / ネガティブ染色法 / 解像度: 32.0 Å | |||||||||
 データ登録者 データ登録者 | Lata S / Schoehn G / Jain A / Pires R / Piehler J / Goettlinger H / Weissenhorn W | |||||||||
 引用 引用 |  ジャーナル: Science / 年: 2008 ジャーナル: Science / 年: 2008タイトル: Helical structures of ESCRT-III are disassembled by VPS4. 著者: Suman Lata / Guy Schoehn / Ankur Jain / Ricardo Pires / Jacob Piehler / Heinrich G Gottlinger / Winfried Weissenhorn /  要旨: During intracellular membrane trafficking and remodeling, protein complexes known as the ESCRTs (endosomal sorting complexes required for transport) interact with membranes and are required for ...During intracellular membrane trafficking and remodeling, protein complexes known as the ESCRTs (endosomal sorting complexes required for transport) interact with membranes and are required for budding processes directed away from the cytosol, including the budding of intralumenal vesicles to form multivesicular bodies; for the budding of some enveloped viruses; and for daughter cell scission in cytokinesis. We found that the ESCRT-III proteins CHMP2A and CHMP3 (charged multivesicular body proteins 2A and 3) could assemble in vitro into helical tubular structures that expose their membrane interaction sites on the outside of the tubule, whereas the AAA-type adenosine triphosphatase VPS4 could bind on the inside of the tubule and disassemble the tubes upon adenosine triphosphate hydrolysis. CHMP2A and CHMP3 copolymerized in solution, and their membrane targeting was cooperatively enhanced on planar lipid bilayers. Such helical CHMP structures could thus assemble within the neck of an inwardly budding vesicle, catalyzing late steps in budding under the control of VPS4. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_1536.map.gz emd_1536.map.gz | 8.5 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-1536-v30.xml emd-1536-v30.xml emd-1536.xml emd-1536.xml | 10.1 KB 10.1 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  EMD-1536.png EMD-1536.png | 263.3 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-1536 http://ftp.pdbj.org/pub/emdb/structures/EMD-1536 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-1536 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-1536 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_1536_validation.pdf.gz emd_1536_validation.pdf.gz | 232.1 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_1536_full_validation.pdf.gz emd_1536_full_validation.pdf.gz | 231.2 KB | 表示 | |
| XML形式データ |  emd_1536_validation.xml.gz emd_1536_validation.xml.gz | 5.5 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1536 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1536 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1536 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1536 | HTTPS FTP |
-関連構造データ
| 類似構造データ |
|---|
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_1536.map.gz / 形式: CCP4 / 大きさ: 51.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_1536.map.gz / 形式: CCP4 / 大きさ: 51.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | ESCRT-III subunits CHMP2A and CHMP3 assemble in vitro into helical tubular structures | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 3.5 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
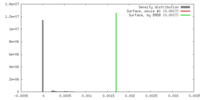
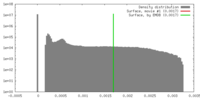
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : CHMP proteins
| 全体 | 名称: CHMP proteins |
|---|---|
| 要素 |
|
-超分子 #1000: CHMP proteins
| 超分子 | 名称: CHMP proteins / タイプ: sample / ID: 1000 / 集合状態: oligomer of dimers / Number unique components: 1 |
|---|
-分子 #1: CHMP
| 分子 | 名称: CHMP / タイプ: protein_or_peptide / ID: 1 / Name.synonym: CHMP 詳細: CHMP2AdeltaC (9-161)-CHMP3deltaC(9 to 183) . MBP attached to CHMP2AdeltaC 集合状態: oligomer of dimers / 組換発現: Yes |
|---|---|
| 由来(天然) | 生物種:  |
| 組換発現 | 生物種:  |
-実験情報
-構造解析
| 手法 | ネガティブ染色法, クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | らせん対称体再構成法 |
| 試料の集合状態 | filament |
- 試料調製
試料調製
| 濃度 | 1 mg/mL |
|---|---|
| 緩衝液 | pH: 7.6 / 詳細: 20 mM HEPES, pH 7.6, 150 mM NaCl |
| 染色 | タイプ: NEGATIVE 詳細: Vitrified samples were prepared according to the method of Dubochet et al., |
| グリッド | 詳細: Quantifoil R2/2 |
| 凍結 | 凍結剤: ETHANE / チャンバー内温度: 100 K / 装置: OTHER / 詳細: Vitrification instrument: Zeiss / 手法: Blot for 2 seconds before plunging |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI/PHILIPS CM200T |
|---|---|
| 温度 | 平均: 100 K |
| アライメント法 | Legacy - 非点収差: orrected at 100,000 times magnification |
| 撮影 | カテゴリ: FILM / フィルム・検出器のモデル: KODAK SO-163 FILM / デジタル化 - スキャナー: ZEISS SCAI / デジタル化 - サンプリング間隔: 7 µm / 実像数: 10 / 平均電子線量: 10 e/Å2 / 詳細: Images binned after scanning / ビット/ピクセル: 8 |
| 電子線 | 加速電圧: 200 kV / 電子線源: LAB6 |
| 電子光学系 | 倍率(補正後): 39500 / 照射モード: OTHER / 撮影モード: BRIGHT FIELD / Cs: 2 mm / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 1.2 µm / 倍率(公称値): 38000 |
| 試料ステージ | 試料ホルダー: Eucentric / 試料ホルダーモデル: GATAN LIQUID NITROGEN |
- 画像解析
画像解析
| 詳細 | Particles were selected by hand using X3d |
|---|---|
| 最終 再構成 | 想定した対称性 - らせんパラメータ - Δz: 32 Å 想定した対称性 - らせんパラメータ - ΔΦ: 21.71 ° アルゴリズム: OTHER / 解像度のタイプ: BY AUTHOR / 解像度: 32.0 Å / 解像度の算出法: OTHER / ソフトウェア - 名称: IHRSR / 詳細: 2500 out of 4400 particles have been used |
| CTF補正 | 詳細: each particle |
 ムービー
ムービー コントローラー
コントローラー



 UCSF Chimera
UCSF Chimera



 Y (Sec.)
Y (Sec.) X (Row.)
X (Row.) Z (Col.)
Z (Col.)