+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Type V-K CAST TnsB bound to LTR-SR | |||||||||||||||
 マップデータ マップデータ | ||||||||||||||||
 試料 試料 |
| |||||||||||||||
| 生物種 |  Scytonema hofmannii (バクテリア) Scytonema hofmannii (バクテリア) | |||||||||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 11.2 Å | |||||||||||||||
 データ登録者 データ登録者 | Tenjo-Castano F / Sofos N / Lopez-Mendez B / Stutzke LS / Fuglsang A / Stella S / Montoya G | |||||||||||||||
| 資金援助 |  デンマーク, 4件 デンマーク, 4件
| |||||||||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2022 ジャーナル: Nat Commun / 年: 2022タイトル: Structure of the TnsB transposase-DNA complex of type V-K CRISPR-associated transposon. 著者: Francisco Tenjo-Castaño / Nicholas Sofos / Blanca López-Méndez / Luisa S Stutzke / Anders Fuglsang / Stefano Stella / Guillermo Montoya /  要旨: CRISPR-associated transposons (CASTs) are mobile genetic elements that co-opted CRISPR-Cas systems for RNA-guided transposition. Here we present the 2.4 Å cryo-EM structure of the Scytonema ...CRISPR-associated transposons (CASTs) are mobile genetic elements that co-opted CRISPR-Cas systems for RNA-guided transposition. Here we present the 2.4 Å cryo-EM structure of the Scytonema hofmannii (sh) TnsB transposase from Type V-K CAST, bound to the strand transfer DNA. The strand transfer complex displays an intertwined pseudo-symmetrical architecture. Two protomers involved in strand transfer display a catalytically competent active site composed by DDE residues, while other two, which play a key structural role, show active sites where the catalytic residues are not properly positioned for phosphodiester hydrolysis. Transposon end recognition is accomplished by the NTD1/2 helical domains. A singular in trans association of NTD1 domains of the catalytically competent subunits with the inactive DDE domains reinforces the assembly. Collectively, the structural features suggest that catalysis is coupled to protein-DNA assembly to secure proper DNA integration. DNA binding residue mutants reveal that lack of specificity decreases activity, but it could increase transposition in some cases. Our structure sheds light on the strand transfer reaction of DDE transposases and offers new insights into CAST transposition. | |||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_15344.map.gz emd_15344.map.gz | 118.5 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-15344-v30.xml emd-15344-v30.xml emd-15344.xml emd-15344.xml | 19.5 KB 19.5 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_15344.png emd_15344.png | 25.2 KB | ||
| その他 |  emd_15344_half_map_1.map.gz emd_15344_half_map_1.map.gz emd_15344_half_map_2.map.gz emd_15344_half_map_2.map.gz | 226.8 MB 226.8 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-15344 http://ftp.pdbj.org/pub/emdb/structures/EMD-15344 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-15344 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-15344 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_15344_validation.pdf.gz emd_15344_validation.pdf.gz | 708.8 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_15344_full_validation.pdf.gz emd_15344_full_validation.pdf.gz | 708.3 KB | 表示 | |
| XML形式データ |  emd_15344_validation.xml.gz emd_15344_validation.xml.gz | 16 KB | 表示 | |
| CIF形式データ |  emd_15344_validation.cif.gz emd_15344_validation.cif.gz | 18.9 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-15344 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-15344 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-15344 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-15344 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_15344.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_15344.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 0.98 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #1
| ファイル | emd_15344_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
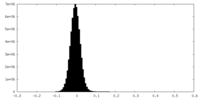
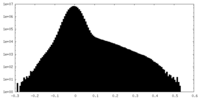
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_15344_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
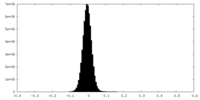
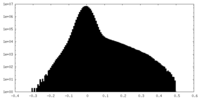
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Type V-K CAST TnsB bound to LTR-SR
| 全体 | 名称: Type V-K CAST TnsB bound to LTR-SR |
|---|---|
| 要素 |
|
-超分子 #1: Type V-K CAST TnsB bound to LTR-SR
| 超分子 | 名称: Type V-K CAST TnsB bound to LTR-SR / タイプ: complex / キメラ: Yes / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Scytonema hofmannii (バクテリア) Scytonema hofmannii (バクテリア) |
| 組換発現 | 生物種:  |
| 分子量 | 実験値: 359 KDa |
-分子 #1: TnsB
| 分子 | 名称: TnsB / タイプ: protein_or_peptide / ID: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Scytonema hofmannii (バクテリア) Scytonema hofmannii (バクテリア) |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MNSQQNPDLA VHPLAIPMEG LLGESATTLE KNVIATQLSE EAQVKLEVIQ SLLEPCDRTT YGQKLREAAE KLNVSLRTVQ RLVKNWEQDG LVGLTQTSRA DKGKHRIGEF WEN FITKTY KEGNKGSKRM TPKQVALRVE AKARELKDSK PPNYKTVLRV LAPILEKQQK ...文字列: MNSQQNPDLA VHPLAIPMEG LLGESATTLE KNVIATQLSE EAQVKLEVIQ SLLEPCDRTT YGQKLREAAE KLNVSLRTVQ RLVKNWEQDG LVGLTQTSRA DKGKHRIGEF WEN FITKTY KEGNKGSKRM TPKQVALRVE AKARELKDSK PPNYKTVLRV LAPILEKQQK AKSIRSPGWR GTTLSVKTRE GKDLSVDYSN HVWQCDHTRV DVLLVDQHGE ILSRPWLT T VIDTYSRCIM GINLGFDAPS SGVVALALRH AILPKRYGSE YKLHCEWGTY GKPEHFYTDG GKDFRSNHLS QIGAQLGFVC HLRDRPSEGG VVERPFKTLN DQLFSTLPGY TGSN VQERP EDAEKDARLT LRELEQLLVR YIVDRYNQSI DARMGDQTRF ERWEAGLPTV PVPIPERDLD ICLMKQSRRT VQRGGCLQFQ NLMYRGEYLA GYAGETVNLR FDPRDITTI LVYRQENNQE VFLTRAHAQG LETEQLALDE AEAASRRLRT AGKTISNQSL LQEVVDRDAL VATKKSRKER QKLEQTVLRS AAVDESNRES LPSQIVEPDE VESTETVHSQ YEDIEVWDYE QLREEYGFGS EFELENLYFQ |
-分子 #2: RightElement
| 分子 | 名称: RightElement / タイプ: dna / ID: 2 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種:  Scytonema hofmannii (バクテリア) Scytonema hofmannii (バクテリア) |
| 配列 | 文字列: TGCTACGTCT CTACGTGTAC AGTGACTAAT TATATGTCGT TGTGACAAAT TATTGTCATC AGTAAAATCC TTATACAGTA TAGATTATAG CGCTTTGGCA GTTTTAGCAT AACCTCTTTG CAGTGACAAA ATAGATGTCG TTGTCCGTGA TTGTGACAAA TTAGCTGTCG ...文字列: TGCTACGTCT CTACGTGTAC AGTGACTAAT TATATGTCGT TGTGACAAAT TATTGTCATC AGTAAAATCC TTATACAGTA TAGATTATAG CGCTTTGGCA GTTTTAGCAT AACCTCTTTG CAGTGACAAA ATAGATGTCG TTGTCCGTGA TTGTGACAAA TTAGCTGTCG CTTTGCAAGA TAGGAAAAAG CTTTTGTGTA TTTTCATAAT GACAAATTGA CTGTCGCAGG AGGTAA |
-分子 #3: LeftElement
| 分子 | 名称: LeftElement / タイプ: dna / ID: 3 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種:  Scytonema hofmannii (バクテリア) Scytonema hofmannii (バクテリア) |
| 配列 | 文字列: ATTATATTGA TGACATTTAA TTTGTCATCA ATTAATTAAG CAACGCTGAT GGGTCACGAC GACAATTAAA TAGTCACAAT GACATTAATC TGTCACCGAC GACAGATAAT TTGTCACTGT ACACTACGCC TTTTGTGG |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 構成要素:
| |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | TFS GLACIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: FEI FALCON III (4k x 4k) 検出モード: COUNTING / 撮影したグリッド数: 1 / 実像数: 738 / 平均露光時間: 40.0 sec. / 平均電子線量: 40.0 e/Å2 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 70.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 0.9 µm / 倍率(公称値): 150000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: AB INITIO MODEL |
|---|
 ムービー
ムービー コントローラー
コントローラー






 Z
Z Y
Y X
X