+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Single Particle cryo-EM of the lipid binding protein P116 (MPN213) refilled with FBS from Mycoplasma pneumoniae at 3.5 Angstrom resolution. | ||||||||||||
 マップデータ マップデータ | |||||||||||||
 試料 試料 |
| ||||||||||||
| 生物種 |  Mycoplasma pneumoniae M129 (バクテリア) Mycoplasma pneumoniae M129 (バクテリア) | ||||||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.5 Å | ||||||||||||
 データ登録者 データ登録者 | Sprankel L / Vizarraga D | ||||||||||||
| 資金援助 |  ドイツ, 3件 ドイツ, 3件
| ||||||||||||
 引用 引用 |  ジャーナル: Nat Struct Mol Biol / 年: 2023 ジャーナル: Nat Struct Mol Biol / 年: 2023タイトル: Essential protein P116 extracts cholesterol and other indispensable lipids for Mycoplasmas. 著者: Lasse Sprankel / David Vizarraga / Jesús Martín / Sina Manger / Jakob Meier-Credo / Marina Marcos / Josep Julve / Noemi Rotllan / Margot P Scheffer / Joan Carles Escolà-Gil / Julian D ...著者: Lasse Sprankel / David Vizarraga / Jesús Martín / Sina Manger / Jakob Meier-Credo / Marina Marcos / Josep Julve / Noemi Rotllan / Margot P Scheffer / Joan Carles Escolà-Gil / Julian D Langer / Jaume Piñol / Ignacio Fita / Achilleas S Frangakis /   要旨: Mycoplasma pneumoniae, responsible for approximately 30% of community-acquired human pneumonia, needs to extract lipids from the host environment for survival and proliferation. Here, we report a ...Mycoplasma pneumoniae, responsible for approximately 30% of community-acquired human pneumonia, needs to extract lipids from the host environment for survival and proliferation. Here, we report a comprehensive structural and functional analysis of the previously uncharacterized protein P116 (MPN_213). Single-particle cryo-electron microscopy of P116 reveals a homodimer presenting a previously unseen fold, forming a huge hydrophobic cavity, which is fully accessible to solvent. Lipidomics analysis shows that P116 specifically extracts lipids such as phosphatidylcholine, sphingomyelin and cholesterol. Structures of different conformational states reveal the mechanism by which lipids are extracted. This finding immediately suggests a way to control Mycoplasma infection by interfering with lipid uptake. | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_15276.map.gz emd_15276.map.gz | 484 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-15276-v30.xml emd-15276-v30.xml emd-15276.xml emd-15276.xml | 18.6 KB 18.6 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
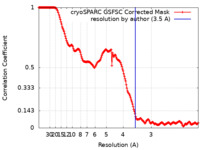
| FSC (解像度算出) |  emd_15276_fsc.xml emd_15276_fsc.xml | 16.9 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_15276.png emd_15276.png | 36.4 KB | ||
| マスクデータ |  emd_15276_msk_1.map emd_15276_msk_1.map | 512 MB |  マスクマップ マスクマップ | |
| その他 |  emd_15276_half_map_1.map.gz emd_15276_half_map_1.map.gz emd_15276_half_map_2.map.gz emd_15276_half_map_2.map.gz | 475.5 MB 475.5 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-15276 http://ftp.pdbj.org/pub/emdb/structures/EMD-15276 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-15276 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-15276 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_15276_validation.pdf.gz emd_15276_validation.pdf.gz | 649.2 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_15276_full_validation.pdf.gz emd_15276_full_validation.pdf.gz | 648.8 KB | 表示 | |
| XML形式データ |  emd_15276_validation.xml.gz emd_15276_validation.xml.gz | 26.3 KB | 表示 | |
| CIF形式データ |  emd_15276_validation.cif.gz emd_15276_validation.cif.gz | 34.9 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-15276 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-15276 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-15276 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-15276 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_15276.map.gz / 形式: CCP4 / 大きさ: 512 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_15276.map.gz / 形式: CCP4 / 大きさ: 512 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.05 Å | ||||||||||||||||||||||||||||||||||||
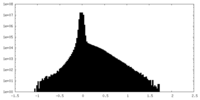
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_15276_msk_1.map emd_15276_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
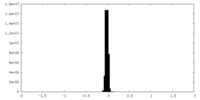
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_15276_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
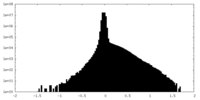
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_15276_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
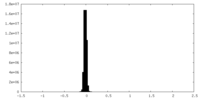
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : P116 homodimer refilled
| 全体 | 名称: P116 homodimer refilled |
|---|---|
| 要素 |
|
-超分子 #1: P116 homodimer refilled
| 超分子 | 名称: P116 homodimer refilled / タイプ: complex / ID: 1 / キメラ: Yes / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Mycoplasma pneumoniae M129 (バクテリア) Mycoplasma pneumoniae M129 (バクテリア) |
-分子 #1: P116 refilled with FBS
| 分子 | 名称: P116 refilled with FBS / タイプ: protein_or_peptide / ID: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Mycoplasma pneumoniae M129 (バクテリア) Mycoplasma pneumoniae M129 (バクテリア) |
| 組換発現 | 生物種:  |
| 配列 | 文字列: NKTHQVEHES EQSDFQDIRF GLNSVKLPKA Q PAAATRIT VENGTDKLVN YKSSPQQLFL AKNALKDKLQ GEFDKFLSDA KAFPALTADL QE WVDQQLF NPNQSFFDLS APRSNFTLSS DKKASLDFIF RFTNFTESVQ LLKLPEGVSV VVD SKQSFD YYVNASAQKL ...文字列: NKTHQVEHES EQSDFQDIRF GLNSVKLPKA Q PAAATRIT VENGTDKLVN YKSSPQQLFL AKNALKDKLQ GEFDKFLSDA KAFPALTADL QE WVDQQLF NPNQSFFDLS APRSNFTLSS DKKASLDFIF RFTNFTESVQ LLKLPEGVSV VVD SKQSFD YYVNASAQKL LVLPLSLPDY TLGLNYMFDH ITLNGKVVNK FSFNPFKTNL NLAF SNVYN GVDVFEAQKN LVGKGKYLNT HVKAEDVKKD VNANIKNQFD IAKIIAELMG KALKE FGNQ QEGQPLSFLK VMDKVKEDFE KLFNLVRPGL GKFVKDLIQS SSQAENKITV YKLIFD NKK TILNLLKELS IPELNSSLGL VDVLFDGITD SDGLYERLQS FKDLIVPAVK TNEKTAA LS PLIEELLTQK DTYVFDLIQK HKGILTNLLK NFLADFQKST PFMADQVAIF TELFDNEG A FDLFGEADFV DKIAELFLTK RTVKNGEKIE TKDSLLVTSL KSLLGEKVAA LGDLLDSYI FKNELLNRSV EVAKAEAKDT KGATDYKKEQ AKALKKLFKH IGENTLSKTN LDKITLKEVK NTENVELEE TETTLKVKKL DVEYKVELGN FEIKNGLIKA MLEFLPDTKD LETTLDKLLF K GESYKAMK DKYIKEGFPG YGWAKGVVPG AFESIENTFK SAIDKTKSIR DLFGDMLFGN DL SSVKETD SFITLGGSFD IKYGGENLNV LPAYYSLINS EIGYQIIGVD TTIDATKVKV ELK NKEYKG KSPAINGQVK LSQSFFNVWT NMFDSITKQI FQKKYEFKDN IQVFARNEDN TSRL ELDIS DPEQRVIPFA FVDGFGIQLK AVDKNITKEA GNTEPKSPVI QLYEALNKEK DQKQQ SKQS PKQLDTKTQL GYLLKLGDNW SKDDYKSLID DTIINNNYLE ASFNSKITVD RLGIPI DLW LFKIWPKFNL EIPMQGSLQL YSSSVIFPYG IYDTSVQDAA KIVKRLNFTD MGFKLND PK PNFWFVGFKH HHHH |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.4 mg/mL | ||||||
|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.4 / 構成要素:
| ||||||
| グリッド | モデル: C-flat-1.2/1.3 / 材質: COPPER / メッシュ: 300 / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 45 sec. / 前処理 - 雰囲気: AIR | ||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277.15 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 特殊光学系 | エネルギーフィルター - 名称: GIF Quantum SE / エネルギーフィルター - スリット幅: 20 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / デジタル化 - サイズ - 横: 3710 pixel / デジタル化 - サイズ - 縦: 3838 pixel / 撮影したグリッド数: 1 / 実像数: 4019 / 平均電子線量: 50.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 70.0 µm / 最大 デフォーカス(補正後): 3.5 µm / 最小 デフォーカス(補正後): 1.0 µm / 倍率(補正後): 130000 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 3.5 µm / 最小 デフォーカス(公称値): 1.0 µm / 倍率(公称値): 130000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: REAL |
|---|
 ムービー
ムービー コントローラー
コントローラー









 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)