+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-1527 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | The structure of phosphorylase kinase holoenzyme at 9.9 A resolution and location of the catalytic subunit and the substrate glycogen phosphorylase | |||||||||
 マップデータ マップデータ | 3D map of PhK | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Phosphorylase kinase | |||||||||
| 生物種 |  | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 9.9 Å | |||||||||
 データ登録者 データ登録者 | Venien-Bryan C / Jonic S / Skamnaki V / Brown N / Bishler N / Oikonomakos NG / Boisset N / Johnson LN | |||||||||
 引用 引用 |  ジャーナル: Structure / 年: 2009 ジャーナル: Structure / 年: 2009タイトル: The structure of phosphorylase kinase holoenzyme at 9.9 angstroms resolution and location of the catalytic subunit and the substrate glycogen phosphorylase. 著者: Catherine Vénien-Bryan / Slavica Jonic / Vasiliki Skamnaki / Nick Brown / Nicolas Bischler / Nikos G Oikonomakos / Nicolas Boisset / Louise N Johnson /  要旨: Phosphorylase kinase (PhK) coordinates hormonal and neuronal signals to initiate the breakdown of glycogen. The enzyme catalyzes the phosphorylation of inactive glycogen phosphorylase b (GPb), ...Phosphorylase kinase (PhK) coordinates hormonal and neuronal signals to initiate the breakdown of glycogen. The enzyme catalyzes the phosphorylation of inactive glycogen phosphorylase b (GPb), resulting in the formation of active glycogen phosphorylase a. We present a 9.9 angstroms resolution structure of PhK heterotetramer (alphabetagammadelta)4 determined by cryo-electron microscopy single-particle reconstruction. The enzyme has a butterfly-like shape comprising two lobes with 222 symmetry. This three-dimensional structure has allowed us to dock the catalytic gamma subunit to the PhK holoenzyme at a location that is toward the ends of the lobes. We have also determined the structure of PhK decorated with GPb at 18 angstroms resolution, which shows the location of the substrate near the kinase subunit. The PhK preparation contained a number of smaller particles whose structure at 9.8 angstroms resolution was consistent with a proteolysed activated form of PhK that had lost the alpha subunits and possibly the gamma subunits. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_1527.map.gz emd_1527.map.gz | 20.6 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-1527-v30.xml emd-1527-v30.xml emd-1527.xml emd-1527.xml | 10.5 KB 10.5 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  1527.gif 1527.gif | 76.9 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-1527 http://ftp.pdbj.org/pub/emdb/structures/EMD-1527 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-1527 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-1527 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_1527_validation.pdf.gz emd_1527_validation.pdf.gz | 209.6 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_1527_full_validation.pdf.gz emd_1527_full_validation.pdf.gz | 208.7 KB | 表示 | |
| XML形式データ |  emd_1527_validation.xml.gz emd_1527_validation.xml.gz | 5.6 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1527 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1527 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1527 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1527 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_1527.map.gz / 形式: CCP4 / 大きさ: 21.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_1527.map.gz / 形式: CCP4 / 大きさ: 21.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | 3D map of PhK | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 2.07 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
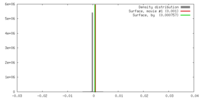
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Phosphorylase kinase holoenzyme purified from rabbit muscle
| 全体 | 名称: Phosphorylase kinase holoenzyme purified from rabbit muscle |
|---|---|
| 要素 |
|
-超分子 #1000: Phosphorylase kinase holoenzyme purified from rabbit muscle
| 超分子 | 名称: Phosphorylase kinase holoenzyme purified from rabbit muscle タイプ: sample / ID: 1000 集合状態: hexadecamer assembly of four different subunits arranged as an (abgd)4 tetramer Number unique components: 4 |
|---|---|
| 分子量 | 実験値: 1.3 MDa / 理論値: 1.3 MDa |
-分子 #1: Phosphorylase kinase
| 分子 | 名称: Phosphorylase kinase / タイプ: protein_or_peptide / ID: 1 / Name.synonym: PhK / コピー数: 4 / 集合状態: hexadecamer / 組換発現: No |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 実験値: 1.3 MDa / 理論値: 1.3 MDa |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 1.0 mg/mL |
|---|---|
| 緩衝液 | pH: 8.2 詳細: 100mM NaCl, 0.3 mM CaCl2, 5mM MgCl2, 50 mM Hepes, pH 8.2 |
| グリッド | 詳細: 400-mesh copper grid coated with a thin holey-carbon film |
| 凍結 | 凍結剤: ETHANE / 装置: HOMEMADE PLUNGER 詳細: Vitrification instrument: Manual. 5 microL were applied on a 200 mesh copper grid, coated with a thin holey carbon film. After blotting the excess of solution with Whatman paper, the grid was ...詳細: Vitrification instrument: Manual. 5 microL were applied on a 200 mesh copper grid, coated with a thin holey carbon film. After blotting the excess of solution with Whatman paper, the grid was rapidly plunged into liquid ethane 手法: Single-sided blotting |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | JEOL 2010UHR |
|---|---|
| 温度 | 最低: 91.15 K / 最高: 93.15 K / 平均: 93.15 K |
| 詳細 | low dose illumination |
| 日付 | 2005年4月13日 |
| 撮影 | カテゴリ: FILM / フィルム・検出器のモデル: KODAK SO-163 FILM デジタル化 - スキャナー: NIKON SUPER COOLSCAN 9000 デジタル化 - サンプリング間隔: 2.07 µm / 実像数: 98 / 平均電子線量: 10 e/Å2 / ビット/ピクセル: 8 |
| Tilt angle min | 0 |
| Tilt angle max | 0 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 0.5 mm / 最大 デフォーカス(公称値): 4.8 µm / 最小 デフォーカス(公称値): 2.8 µm / 倍率(公称値): 50000 |
| 試料ステージ | 試料ホルダー: Gatan / 試料ホルダーモデル: GATAN LIQUID NITROGEN |
- 画像解析
画像解析
| 詳細 | The particles were selected using an automatic selection program |
|---|---|
| CTF補正 | 詳細: Wiener filtration of volumes from focal series |
| 最終 再構成 | 想定した対称性 - 点群: D2 (2回x2回 2面回転対称) アルゴリズム: OTHER / 解像度のタイプ: BY AUTHOR / 解像度: 9.9 Å / 解像度の算出法: FSC 0.5 CUT-OFF / ソフトウェア - 名称: SPIDER 詳細: Final map was calculated using five groups of defocus 使用した粒子像数: 18123 |
-原子モデル構築 1
| 初期モデル | PDB ID: |
|---|---|
| ソフトウェア | 名称: Situs |
| 詳細 | Protocol: rigid body |
| 精密化 | 空間: RECIPROCAL / プロトコル: RIGID BODY FIT 当てはまり具合の基準: cross correlation coefficient |
 ムービー
ムービー コントローラー
コントローラー









 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)