+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-1512 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | CryoEM structure of Blackcurrant Reversion Virus | |||||||||
 マップデータ マップデータ | This is a reconstruction of blackcurrant reversion virus at 17 angstrom resolution. | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Cryo / electron / microscopy / structure / blackcurrant / reversion / virus | |||||||||
| 生物種 |  Blackcurrant reversion virus (ウイルス) Blackcurrant reversion virus (ウイルス) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / ネガティブ染色法 / 解像度: 17.0 Å | |||||||||
 データ登録者 データ登録者 | Seitsonen JJT / Susi P / Lemmetty A / Butcher SJ | |||||||||
 引用 引用 |  ジャーナル: Virology / 年: 2008 ジャーナル: Virology / 年: 2008タイトル: Structure of the mite-transmitted Blackcurrant reversion nepovirus using electron cryo-microscopy. 著者: Jani J T Seitsonen / Petri Susi / Anne Lemmetty / Sarah J Butcher /  要旨: Blackcurrant reversion nepovirus (BRV; genus Nepovirus) has a single-stranded, bipartite RNA genome surrounded by 60 copies of a single capsid protein (CP). BRV is the most important mite-transmitted ...Blackcurrant reversion nepovirus (BRV; genus Nepovirus) has a single-stranded, bipartite RNA genome surrounded by 60 copies of a single capsid protein (CP). BRV is the most important mite-transmitted viral pathogen of the Ribes species. It is the causal agent of blackcurrant reversion disease. We determined the structure of BRV to 1.7 nm resolution using electron cryo- microscopy (cryoEM) and image reconstruction. The reconstruction reveals a pseudo T=3 viral capsid similar to that of tobacco ringspot virus (TRSV). We modelled the BRV capsid protein to that of TRSV and fitted it into the cryoEM reconstruction. The fit indicated that the extended C-terminus of BRV-CP is located on the capsid surface and the N-terminus on the interior. We generated peptide antibodies to two putatively exposed C-terminal sequences and these reacted with the virus. Hence homology modelling may be useful for defining epitopes for antibody generation for diagnostic testing of BRV in commercial crops. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_1512.map.gz emd_1512.map.gz | 4 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-1512-v30.xml emd-1512-v30.xml emd-1512.xml emd-1512.xml | 9.9 KB 9.9 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  1512.gif 1512.gif | 75.4 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-1512 http://ftp.pdbj.org/pub/emdb/structures/EMD-1512 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-1512 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-1512 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_1512_validation.pdf.gz emd_1512_validation.pdf.gz | 203.3 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_1512_full_validation.pdf.gz emd_1512_full_validation.pdf.gz | 202.5 KB | 表示 | |
| XML形式データ |  emd_1512_validation.xml.gz emd_1512_validation.xml.gz | 6.7 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1512 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1512 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1512 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1512 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_1512.map.gz / 形式: CCP4 / 大きさ: 30.3 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_1512.map.gz / 形式: CCP4 / 大きさ: 30.3 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | This is a reconstruction of blackcurrant reversion virus at 17 angstrom resolution. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 2.26203 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
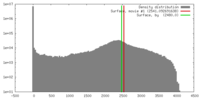
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Purified blackcurrant reversion virus in 0.05 M Na-citrate buffer...
| 全体 | 名称: Purified blackcurrant reversion virus in 0.05 M Na-citrate buffer (pH 7.0) |
|---|---|
| 要素 |
|
-超分子 #1000: Purified blackcurrant reversion virus in 0.05 M Na-citrate buffer...
| 超分子 | 名称: Purified blackcurrant reversion virus in 0.05 M Na-citrate buffer (pH 7.0) タイプ: sample / ID: 1000 詳細: The sample was stored at -80 degrees celcius and thawed before loading onto grids 集合状態: Complete particle, (60-mer) with the RNA genome inside Number unique components: 1 |
|---|---|
| 分子量 | 実験値: 3.30 MDa / 理論値: 3.45 MDa 手法: SDS-PAGE (55 kDa per capsid protein, 60 capsid proteins per capsid) (Lemmetty et al. 1997, The American Phytopathology Society) |
-超分子 #1: Blackcurrant reversion virus
| 超分子 | 名称: Blackcurrant reversion virus / タイプ: virus / ID: 1 / Name.synonym: BRV / NCBI-ID: 65743 / 生物種: Blackcurrant reversion virus / ウイルスタイプ: VIRION / ウイルス・単離状態: SPECIES / ウイルス・エンベロープ: No / ウイルス・中空状態: No / Syn species name: BRV |
|---|---|
| 宿主 | 生物種:  Chenopodium quinoa (植物) / 別称: PLANTAE(HIGHER PLANTS) Chenopodium quinoa (植物) / 別称: PLANTAE(HIGHER PLANTS) |
| 分子量 | 実験値: 3.30 MDa / 理論値: 3.45 MDa |
| ウイルス殻 | Shell ID: 1 / 名称: Coat protein / 直径: 290 Å / T番号(三角分割数): 1 |
-実験情報
-構造解析
| 手法 | ネガティブ染色法, クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 1 mg/mL |
|---|---|
| 緩衝液 | pH: 7 / 詳細: 0.05 M Na-citrate buffer |
| 染色 | タイプ: NEGATIVE 詳細: Vitrified. Grids were blotted for roughly one second before being plunged into liquid ethane. |
| グリッド | 詳細: C-Flat 224 grids |
| 凍結 | 凍結剤: ETHANE / 装置: HOMEMADE PLUNGER / 詳細: Vitrification instrument: Guillotine / 手法: Blot for 1 second before plunging |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TECNAI F20 |
|---|---|
| アライメント法 | Legacy - 非点収差: astigmatism was corrected at 62000x magnification |
| 詳細 | Part of the data was recorded on Kodak SO163 film at nominal magnification of 50000. |
| 撮影 | カテゴリ: CCD フィルム・検出器のモデル: GATAN ULTRASCAN 4000 (4k x 4k) デジタル化 - サンプリング間隔: 15 µm / 実像数: 65 / 平均電子線量: 17 e/Å2 詳細: 65 images total, 10 images were scanned from film using Zeiss Photoscan TD scanner with a stepsize of 7 micrometers. |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 倍率(補正後): 66400 / 照射モード: SPOT SCAN / 撮影モード: BRIGHT FIELD / Cs: 2.0 mm / 最大 デフォーカス(公称値): 4.6 µm / 最小 デフォーカス(公称値): 1.6 µm / 倍率(公称値): 68000 |
| 試料ステージ | 試料ホルダー: Gatan 626 / 試料ホルダーモデル: GATAN LIQUID NITROGEN |
| 実験機器 |  モデル: Tecnai F20 / 画像提供: FEI Company |
- 画像解析
画像解析
| CTF補正 | 詳細: each micrograph |
|---|---|
| 最終 再構成 | 想定した対称性 - 点群: I (正20面体型対称) / アルゴリズム: OTHER / 解像度のタイプ: BY AUTHOR / 解像度: 17.0 Å / 解像度の算出法: FSC 0.5 CUT-OFF / ソフトウェア - 名称: PFT, POR, EM3DR, P3DR 詳細: Particles from CCD data and film data were combined for the final reconstruction. 使用した粒子像数: 831 |
 ムービー
ムービー コントローラー
コントローラー










 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)