+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | HOPS tethering complex from yeast, composite map | ||||||||||||
 マップデータ マップデータ | Tethering complex HOPS, composite map | ||||||||||||
 試料 試料 |
| ||||||||||||
 キーワード キーワード | HOPS / tethering complex / lysosome / membrane fusion / Rab GTPase / cryo-EM / CYTOSOLIC PROTEIN | ||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報histone catabolic process / organelle fusion / CORVET complex / HOPS complex / endosomal vesicle fusion / vesicle tethering / regulation of vacuole fusion, non-autophagic / vacuole-mitochondrion membrane contact site / vacuolar protein processing / Golgi to vacuole transport ...histone catabolic process / organelle fusion / CORVET complex / HOPS complex / endosomal vesicle fusion / vesicle tethering / regulation of vacuole fusion, non-autophagic / vacuole-mitochondrion membrane contact site / vacuolar protein processing / Golgi to vacuole transport / regulation of SNARE complex assembly / vesicle fusion with vacuole / vacuole fusion, non-autophagic / cytoplasm to vacuole targeting by the Cvt pathway / Golgi to endosome transport / vesicle docking / vacuole organization / protein targeting to vacuole / late endosome to vacuole transport / endosome organization / piecemeal microautophagy of the nucleus / fungal-type vacuole / fungal-type vacuole membrane / vesicle docking involved in exocytosis / endosomal transport / vesicle-mediated transport / positive regulation of TORC1 signaling / guanyl-nucleotide exchange factor activity / cellular response to starvation / macroautophagy / intracellular protein transport / RING-type E3 ubiquitin transferase / autophagy / small GTPase binding / endocytosis / ubiquitin protein ligase activity / late endosome / protein transport / actin binding / early endosome membrane / protein-macromolecule adaptor activity / endosome / zinc ion binding / ATP binding / cytosol / cytoplasm 類似検索 - 分子機能 | ||||||||||||
| 生物種 |  | ||||||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 4.4 Å | ||||||||||||
 データ登録者 データ登録者 | Shvarev D / Schoppe J / Koenig C / Perz A / Fuellbrunn N / Kiontke S / Langemeyer L / Januliene D / Schnelle K / Kuemmel D ...Shvarev D / Schoppe J / Koenig C / Perz A / Fuellbrunn N / Kiontke S / Langemeyer L / Januliene D / Schnelle K / Kuemmel D / Froehlich F / Moeller A / Ungermann C | ||||||||||||
| 資金援助 |  ドイツ, 3件 ドイツ, 3件
| ||||||||||||
 引用 引用 |  ジャーナル: Elife / 年: 2022 ジャーナル: Elife / 年: 2022タイトル: Structure of the HOPS tethering complex, a lysosomal membrane fusion machinery. 著者: Dmitry Shvarev / Jannis Schoppe / Caroline König / Angela Perz / Nadia Füllbrunn / Stephan Kiontke / Lars Langemeyer / Dovile Januliene / Kilian Schnelle / Daniel Kümmel / Florian ...著者: Dmitry Shvarev / Jannis Schoppe / Caroline König / Angela Perz / Nadia Füllbrunn / Stephan Kiontke / Lars Langemeyer / Dovile Januliene / Kilian Schnelle / Daniel Kümmel / Florian Fröhlich / Arne Moeller / Christian Ungermann /  要旨: Lysosomes are essential for cellular recycling, nutrient signaling, autophagy, and pathogenic bacteria and viruses invasion. Lysosomal fusion is fundamental to cell survival and requires HOPS, a ...Lysosomes are essential for cellular recycling, nutrient signaling, autophagy, and pathogenic bacteria and viruses invasion. Lysosomal fusion is fundamental to cell survival and requires HOPS, a conserved heterohexameric tethering complex. On the membranes to be fused, HOPS binds small membrane-associated GTPases and assembles SNAREs for fusion, but how the complex fulfills its function remained speculative. Here, we used cryo-electron microscopy to reveal the structure of HOPS. Unlike previously reported, significant flexibility of HOPS is confined to its extremities, where GTPase binding occurs. The SNARE-binding module is firmly attached to the core, therefore, ideally positioned between the membranes to catalyze fusion. Our data suggest a model for how HOPS fulfills its dual functionality of tethering and fusion and indicate why it is an essential part of the membrane fusion machinery. | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_14964.map.gz emd_14964.map.gz | 1.1 GB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-14964-v30.xml emd-14964-v30.xml emd-14964.xml emd-14964.xml | 30.6 KB 30.6 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_14964.png emd_14964.png | 75.6 KB | ||
| Filedesc metadata |  emd-14964.cif.gz emd-14964.cif.gz | 10.1 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-14964 http://ftp.pdbj.org/pub/emdb/structures/EMD-14964 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-14964 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-14964 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_14964_validation.pdf.gz emd_14964_validation.pdf.gz | 518.6 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_14964_full_validation.pdf.gz emd_14964_full_validation.pdf.gz | 518.2 KB | 表示 | |
| XML形式データ |  emd_14964_validation.xml.gz emd_14964_validation.xml.gz | 9 KB | 表示 | |
| CIF形式データ |  emd_14964_validation.cif.gz emd_14964_validation.cif.gz | 10.6 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-14964 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-14964 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-14964 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-14964 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  7zu0MC  7ztyC C: 同じ文献を引用 ( M: このマップから作成された原子モデル |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_14964.map.gz / 形式: CCP4 / 大きさ: 1.1 GB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_14964.map.gz / 形式: CCP4 / 大きさ: 1.1 GB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Tethering complex HOPS, composite map | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.924 Å | ||||||||||||||||||||||||||||||||||||
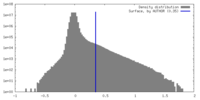
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Tethering complex HOPS
| 全体 | 名称: Tethering complex HOPS |
|---|---|
| 要素 |
|
-超分子 #1: Tethering complex HOPS
| 超分子 | 名称: Tethering complex HOPS / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  |
-分子 #1: E3 ubiquitin-protein ligase PEP5
| 分子 | 名称: E3 ubiquitin-protein ligase PEP5 / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO / EC番号: RING-type E3 ubiquitin transferase |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 117.617219 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MSLSSWRQFQ LFENIPIRDP NFGGDSLLYS DPTLCAATIV DPQTLIIAVN SNIIKVVKLN QSQVIHEFQS FPHDFQITFL KVINGEFLV ALAESIGKPS LIRVYKLEKL PNREQLYHSQ VELKNGNNTY PISVVSISND LSCIVVGFIN GKIILIRGDI S RDRGSQQR ...文字列: MSLSSWRQFQ LFENIPIRDP NFGGDSLLYS DPTLCAATIV DPQTLIIAVN SNIIKVVKLN QSQVIHEFQS FPHDFQITFL KVINGEFLV ALAESIGKPS LIRVYKLEKL PNREQLYHSQ VELKNGNNTY PISVVSISND LSCIVVGFIN GKIILIRGDI S RDRGSQQR IIYEDPSKEP ITALFLNNDA TACFAATTSR ILLFNTTGRN RGRPSLVLNS KNGLDLNCGS FNPATNEFIC CL SNFIEFF SSSGKKHQFA FDLSLRKRIF CVDKDHILIV TEETGVPTTS ISVNELSPTI INRIFIIDAK NKIISLNFVV SSA IIDIFS TSQSGKNITY LLTSEGVMHR ITPKSLENQI NIIIQKELYP FALQLAKQHS LSPLDVQEIH KKYGDYLFKK GLRK EATDQ YIQCLDVVET SEIISKFGVK EVPDPESMRN LADYLWSLIK NSISQRDHVT LLLIVLIKLK DVEGIDTFIQ HFDRK GIWN EGVVMDDMDD VTFFYSDNDF FDLDLILELM KESDFKRLSY RLAKKYSKDS LIIVDILLNL LHNPVKAIKY IKSLPI DET LRCLVTYSKK LLEESPNETN ALLIEVFTGK FKPSTFEVDL DRRDTTGDFS ENIRTVFYSY KTFFNYMNSN GTSDAMS ES SEASHEHEEP TYHPPKPSIV FSSFVTKPFE FVVFLEACLA CYQQYEGFDE DRQVILTTLY DLYLNLAQND VPERIDDW R SRATGVLRES NKLVYSAASN NTSKRVDNSI MLLISHMDQS SASAKDKTKI DIASFANDNP EMDLLSTFRA MTLNEEPST CLKFLEKYGT EEPKLLQVAL SYFVSNKLIF KEMGGNEVLK EKVLRPIIEG ERMPLLDIIK ALSRTNVAHF GLIQDIIIDH VKTEDTEIK RNEKLIESYD KELKEKNKKL KNTINSDQPL HVPLKNQTCF MCRLTLDIPV VFFKCGHIYH QHCLNEEEDT L ESERKLFK CPKCLVDLET SNKLFEAQHE VVEKNDLLNF ALNSEEGSRD RFKVITEFLG RGAISYSDIT I UniProtKB: E3 ubiquitin-protein ligase PEP5 |
-分子 #2: Vacuolar protein sorting-associated protein 16
| 分子 | 名称: Vacuolar protein sorting-associated protein 16 / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 92.857 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MKNPSFDWER LKDVFYRSRA IGELKWPTQY EEFKCALSLT VIAVEIQDFI QVYNYFGQLL GKINLQRIHE DIIKFEFDKD EKLILVTKS SIKIVKGWSP LTIESVPLQD PTIDTIWDYH NGIMLLAKSR DIYKLNGNEW ELLYENKDKK YNLLTKNHWS C NDDSIILL ...文字列: MKNPSFDWER LKDVFYRSRA IGELKWPTQY EEFKCALSLT VIAVEIQDFI QVYNYFGQLL GKINLQRIHE DIIKFEFDKD EKLILVTKS SIKIVKGWSP LTIESVPLQD PTIDTIWDYH NGIMLLAKSR DIYKLNGNEW ELLYENKDKK YNLLTKNHWS C NDDSIILL DVDHVYQVST SNGALLKLIT DSSWHKVTIS SRGFICLYNM KDNKLQIFRD PARILMEHNL DSTPDDICWC GN DTVACSF EDEIKLYGPD GLYVTFWYPF TVTNLRAEVD GLKVITTEKI YFLSRVQPQT SNIFRIGSTE PGAMLVDSFS LLE DHAPKA IEILKNFVLE KGVLDCIAAA IDEFEPKLQK MLLNAASYGK ASLQYKSFDA SIFVNACNTI KLLNCFRSFG IFLT VEEYR CISLKGVIDR LLKYHRYYEC IQICKLANER FLLGYVFTEW AKDKIKGSPD MEDDELLDKI KSRLSVIDMT DTLQM VAVA KVAYLEGRFQ LSRNLALLEK NEEARIEQLY NLDDDSIALK ECIKVQNYSL TISLLIALSK KLTNSQLTKL LIIDMF NNP LYLYYMRMDK AYLYDFYRQT DRFIDLAHVL LQQGKEQQSL HSFLPQIKDL YSQVQNSEVV NNTIEQLQRQ EKLWIYQ ES LGKRFAISFT NMTLDQTLSK LIETGQDKQV KEIVKKFKIS EKKLYHLKCK TLVEAKKFDE LLQFAQSRKS PIGYMPFY T YLKSRGHMDK ASPYVNMIPG LSYQEKKKLY VECRGFRDAI QLAGKEKDIP GLKEIYNIIP PNEPELKALA NETMSRI UniProtKB: Vacuolar protein sorting-associated protein 16 |
-分子 #3: Vacuolar membrane protein PEP3
| 分子 | 名称: Vacuolar membrane protein PEP3 / タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 107.531047 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MIKTRIEEVQ LQFLTGNTEL THLKVSNDQL IVTTQRTIYR INLQDPAIVN HFDCPLSKEL ETIMNVHVSP MGSVILIRTN FGRYMLLKD GEFTQLNKIK NLDLSSLHWI NETTFLMGIK KTPKLYRVEL TGKDITTKLW YENKKLSGGI DGIAYWEGSL L LTIKDNIL ...文字列: MIKTRIEEVQ LQFLTGNTEL THLKVSNDQL IVTTQRTIYR INLQDPAIVN HFDCPLSKEL ETIMNVHVSP MGSVILIRTN FGRYMLLKD GEFTQLNKIK NLDLSSLHWI NETTFLMGIK KTPKLYRVEL TGKDITTKLW YENKKLSGGI DGIAYWEGSL L LTIKDNIL YWRDVTNMKF PLVLPDESEQ FERLKHHAIK KFDSYNGLFA WVTSNGIVFG DLKEKQMEKD PASNNFGKFL SS SKVLLNF ELPDYQNDKD HLIKDIVLTA FHILLLRKNT VTMVSQLNND VVFHETIPRH QLTGSNTDSN EKFLGLVRDS VKE TFWCFS NINVFEIIIE NEPNSVWNLL VRDNKFDKAL SLKGLTVREI ESVKLSKAMY LFHTAKDFHS AAQTLGSMKD LSHF GEIAL NFLQIKDYND LNVILIKQLD NVPWKSTQVV LSSWIIWNFM KQLNDIELKI NTTKPASTDE DNLLNWNLNL KEKSN ELTK FLESHLEKLD NETVYQIMSK QNRQNELLIF ASLINDMKFL LSFWIDQGNW YESLKILLTI NNHDLVYKYS LILLLN SPE ATVSTWMKIK DLDPNKLIPT ILKFFTNWQN NSKLITNISE YPENYSLTYL KWCVREVPKM CNPIVYNSIL YMMITDP RN DMILENDIIK FMKSNENKYD LNFQLRLSLK FKKTKTSIFL LTRLNLFEDA IDLALKNNLI DDCKVIVNDE ILIEDYKL R KRLWLKIAKH LLLSMKDIDI KQLIRTILND SNEILTIKDL LPFFNEYTTI ANLKEELIKF LENHNMKMNE ISEDIINSK NLKVEINTEI SKFNEIYRIL EPGKSCDECG KFLQIKKFIV FPCGHCFHWN CIIRVILNSN DYNLRQKTEN FLKAKSKHNL NDLENIIVE KCGLCSDINI NKIDQPISID ETELAKWNE UniProtKB: Vacuolar membrane protein PEP3 |
-分子 #4: Vacuolar protein sorting-associated protein 33
| 分子 | 名称: Vacuolar protein sorting-associated protein 33 / タイプ: protein_or_peptide / ID: 4 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 79.354977 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MNRFWNTKKF SLTNADGLCA TLNEISQNDE VLVVQPSVLP VLNSLLTFQD LTQSTPVRKI TLLDDQLSDD LPSALGSVPQ MDLIFLIDV RTSLRLPPQL LDAAQKHNLS SLHIIYCRWK PSFQNTLEDT EQWQKDGFDL NSKKTHFPNV IESQLKELSN E YTLYPWDL ...文字列: MNRFWNTKKF SLTNADGLCA TLNEISQNDE VLVVQPSVLP VLNSLLTFQD LTQSTPVRKI TLLDDQLSDD LPSALGSVPQ MDLIFLIDV RTSLRLPPQL LDAAQKHNLS SLHIIYCRWK PSFQNTLEDT EQWQKDGFDL NSKKTHFPNV IESQLKELSN E YTLYPWDL LPFPQIDENV LLTHSLYNME NVNMYYPNLR SLQSATESIL VDDMVNSLQS LIFETNSIIT NVVSIGNLSK RC SHLLKKR IDEHQTENDL FIKGTLYGER TNCGLEMDLI ILERNTDPIT PLLTQLTYAG ILDDLYEFNS GIKIKEKDMN FNY KEDKIW NDLKFLNFGS IGPQLNKLAK ELQTQYDTRH KAESVHEIKE FVDSLGSLQQ RQAFLKNHTT LSSDVLKVVE TEEY GSFNK ILELELEILM GNTLNNDIED IILELQYQYE VDQKKILRLI CLLSLCKNSL REKDYEYLRT FMIDSWGIEK CFQLE SLAE LGFFTSKTGK TDLHITTSKS TRLQKEYRYI SQWFNTVPIE DEHAADKITN ENDDFSEATF AYSGVVPLTM RLVQML YDR SILFHNYSSQ QPFILSREPR VSQTEDLIEQ LYGDSHAIEE SIWVPGTITK KINASIKSNN RRSIDGSNGT FHAAEDI AL VVFLGGVTMG EIAIMKHLQK ILGKKGINKR FIIIADGLIN GTRIMNSIS UniProtKB: Vacuolar protein sorting-associated protein 33 |
-分子 #5: Vacuolar morphogenesis protein 6
| 分子 | 名称: Vacuolar morphogenesis protein 6 / タイプ: protein_or_peptide / ID: 5 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 123.049414 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MLRAQKLHSL KSSDITAILP TEQSQKLVLA KKNGDVEVYS RDGNTLKLFQ VYPDLLQNAK NDPLPPVIEN FYFANELSTI FAQCKETLI LLSTTNLHEY DRIIDRRGIN HCWLFERSHK NKEEKNTYLI YSTINTAKMR VLIWEGRTYK NMMEASLSYR K ETIRSIYP ...文字列: MLRAQKLHSL KSSDITAILP TEQSQKLVLA KKNGDVEVYS RDGNTLKLFQ VYPDLLQNAK NDPLPPVIEN FYFANELSTI FAQCKETLI LLSTTNLHEY DRIIDRRGIN HCWLFERSHK NKEEKNTYLI YSTINTAKMR VLIWEGRTYK NMMEASLSYR K ETIRSIYP GETGITLATD LGIYHWPYNK PSLIRIEKTV KNKFPKDMIS ALTELKEQAE KVIEKKPKKN SHFDAQSFSS MD RMSRKSS MSSLWYRTIR NERGNKIRYT FELDGNDATP MIIDGATKKI FKVELMHNNE EPFLIATDHA TFSESNSEFD HMQ YLSSNL LMLYNSSTIK FVDYENGFTF LQQKIPEGIK WVKNLSGTYF LVWTSNDEVQ LFSYHVDDGS EDDDQESICG DIND PDFYQ LWRKVLFYKF FIDSPHSKEL CVSDNPEESL DICAMKLRDL TVMWCLRIFD KFQNYMVQLE RSRNSRMIRS KCEEM IIKS IFDLFIKFWA PPQLVILKVF PSAISSLVLE ITGQEHHCLL KEAEEVKETY DIPPHLLNRW CLPYLTDTRR HLQNLL SKE NDDESRITWC YRDREIKQSF DFFLISNHDD VDLNTMLTLI DTVLFKCYLY YNPPMVGPFI RVENHCDSHV IVTELKI RH MFKDLIDFYY KRGNHEEALK FLTDLVDELE NDNTDQKQRQ KIDHGVKILV IYYLKKLSNP QLDVIFTYTD WLLNRHND S IKEILSSIFF YDSQACSSRD HLKVYGYIKK FDKLLAIQYL EFAISTFRLE GNKLHTVLIK LYLENLDIPS TRIKLKSLL ETTSVYEPRT ILKLLNDAIE SGSDQLPTNQ LNFVKYLKIF PLSKLENHKE AVHILLDEID DYKAATSYCN DVYQSDSTKG EELLLYLYS KLVSIYDSNR NSKLILNFLQ DHGSKLNSAE IYKNLPQDIS LYDIGRVVSQ LLKKHTSKMD ETRLEKALLQ V ELVATTYK LNERMSSYGV LSDSHKCPIC KKVISNFGTD SISWFTREGR NIITHYNCGK VLQERFNAKN EKSSRIKQKT LG EVINELN NK UniProtKB: Vacuolar morphogenesis protein 6 |
-分子 #6: Vacuolar protein sorting-associated protein 41
| 分子 | 名称: Vacuolar protein sorting-associated protein 41 / タイプ: protein_or_peptide / ID: 6 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 116.530555 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MTTDNHQNDS VLDQQSGERT IDESNSISDE NNVDNKREDV NVTSPTKSVS CISQAENGVA SRTDESTITG SATDAETGDD DDDDDDDDD EDEDDEDEPP LLKYTRISQL PKNFFQRDSI SSCLFGDTFF AFGTHSGILH LTTCAFEPIK TIKCHRSSIL C INTDGKYF ...文字列: MTTDNHQNDS VLDQQSGERT IDESNSISDE NNVDNKREDV NVTSPTKSVS CISQAENGVA SRTDESTITG SATDAETGDD DDDDDDDDD EDEDDEDEPP LLKYTRISQL PKNFFQRDSI SSCLFGDTFF AFGTHSGILH LTTCAFEPIK TIKCHRSSIL C INTDGKYF ATGSIDGTVI IGSMDDPQNI TQYDFKRPIN SVALHSNFQA SRMFVSGGMA GDVVLSQRNW LGNRIDIVLN KK KKKKTRK DDLSSDMKGP IMGIYTMGDL ILWMDDDGIT FCDVPTRSQL LNIPFPSRIF NVQDVRPDLF RPHVHFLESD RVV IGWGSN IWLFKVSFTK DSNSIKSGDS NSQSNNMSHF NPTTNIGSLL SSAASSFRGT PDKKVELECH FTVSMLITGL ASFK DDQLL CLGFDIDIEE EATIDEDMKE GKNFSKRPEN LLAKGNAPEL KIVDLFNGDE IYNDEVIMKN YEKLSINDYH LGKHI DKTT PEYYLISSND AIRVQELSLK DHFDWFMERK QYYKAWKIGK YVIGSEERFS IGLKFLNSLV TKKDWGTLVD HLNIIF EET LNSLDSNSYD VTQNVLKEWA DIIEILITSG NIVEIAPLIP KKPALRKSVY DDVLHYFLAN DMINKFHEYI TKWDLKL FS VEDFEEELET RIEAASEPTA SSKEEGSNIT YRTELVHLYL KENKYTKAIP HLLKAKDLRA LTIIKIQNLL PQYLDQIV D IILLPYKGEI SHISKLSIFE IQTIFNKPID LLFENRHTIS VARIYEIFEH DCPKSFKKIL FCYLIKFLDT DDSFMISPY ENQLIELYSE YDRQSLLPFL QKHNNYNVES AIEVCSSKLG LYNELIYLWG KIGETKKALS LIIDELKNPQ LAIDFVKNWG DSELWEFMI NYSLDKPNFT KAILTCSDET SEIYLKVIRG MSDDLQIDNL QDIIKHIVQE NSLSLEVRDN ILVIINDETK K FANEFLKI RSQGKLFQVD ESDIEINDDL NGVLDYKDDD DKDYKDDDDK DYKDDDDK UniProtKB: Vacuolar protein sorting-associated protein 41 |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.4 |
|---|---|
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | TFS GLACIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: FEI FALCON IV (4k x 4k) 平均電子線量: 50.0 e/Å2 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD 最大 デフォーカス(公称値): 2.8000000000000003 µm 最小 デフォーカス(公称値): 0.8 µm |
- 画像解析
画像解析
| 初期モデル | モデルのタイプ: NONE |
|---|---|
| 最終 再構成 | 解像度のタイプ: BY AUTHOR / 解像度: 4.4 Å / 解像度の算出法: OTHER 詳細: Resolution of the consensus map of the lower part of the complex is used here 使用した粒子像数: 244661 |
| 初期 角度割当 | タイプ: MAXIMUM LIKELIHOOD / ソフトウェア - 名称: cryoSPARC (ver. 3.3.1) |
| 最終 角度割当 | タイプ: MAXIMUM LIKELIHOOD / ソフトウェア - 名称: cryoSPARC (ver. 3.3.1) |
 ムービー
ムービー コントローラー
コントローラー














 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)