+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-1485 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | The archaeal DNA Ligase-PCNA-DNA complex | |||||||||
 マップデータ マップデータ | This is a map of DNA ligase-PCNA-DNA complex | |||||||||
 試料 試料 |
| |||||||||
| 生物種 |   Pyrococcus furiosus (古細菌) / synthetic construct (人工物) Pyrococcus furiosus (古細菌) / synthetic construct (人工物) | |||||||||
| 手法 | 単粒子再構成法 / ネガティブ染色法 / 解像度: 17.0 Å | |||||||||
 データ登録者 データ登録者 | Mayanagi K / Kiyonari S / Ishino Y / Shirai T / Morikawa K | |||||||||
 引用 引用 |  ジャーナル: Proc Natl Acad Sci U S A / 年: 2009 ジャーナル: Proc Natl Acad Sci U S A / 年: 2009タイトル: Mechanism of replication machinery assembly as revealed by the DNA ligase-PCNA-DNA complex architecture. 著者: Kouta Mayanagi / Shinichi Kiyonari / Mihoko Saito / Tsuyoshi Shirai / Yoshizumi Ishino / Kosuke Morikawa /  要旨: The 3D structure of the ternary complex, consisting of DNA ligase, the proliferating cell nuclear antigen (PCNA) clamp, and DNA, was investigated by single-particle analysis. This report presents the ...The 3D structure of the ternary complex, consisting of DNA ligase, the proliferating cell nuclear antigen (PCNA) clamp, and DNA, was investigated by single-particle analysis. This report presents the structural view, where the crescent-shaped DNA ligase with 3 distinct domains surrounds the central DNA duplex, encircled by the closed PCNA ring, thus forming a double-layer structure with dual contacts between the 2 proteins. The relative orientations of the DNA ligase domains, which remarkably differ from those of the known crystal structures, suggest that a large domain rearrangement occurs upon ternary complex formation. A second contact was found between the PCNA ring and the middle adenylation domain of the DNA ligase. Notably, the map revealed a substantial DNA tilt from the PCNA ring axis. This structure allows us to propose a switching mechanism for the replication factors operating on the PCNA ring. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_1485.map.gz emd_1485.map.gz | 209.8 KB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-1485-v30.xml emd-1485-v30.xml emd-1485.xml emd-1485.xml | 9.8 KB 9.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  1485.png 1485.png | 286 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-1485 http://ftp.pdbj.org/pub/emdb/structures/EMD-1485 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-1485 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-1485 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_1485_validation.pdf.gz emd_1485_validation.pdf.gz | 200.5 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_1485_full_validation.pdf.gz emd_1485_full_validation.pdf.gz | 199.6 KB | 表示 | |
| XML形式データ |  emd_1485_validation.xml.gz emd_1485_validation.xml.gz | 4.6 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1485 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1485 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1485 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1485 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_1485.map.gz / 形式: CCP4 / 大きさ: 670.9 KB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_1485.map.gz / 形式: CCP4 / 大きさ: 670.9 KB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | This is a map of DNA ligase-PCNA-DNA complex | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 3.1 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
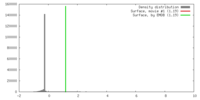
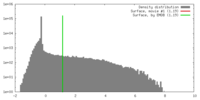
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Pyrococcus furiosus DNA Ligase bound to PCNA and DNA
| 全体 | 名称: Pyrococcus furiosus DNA Ligase bound to PCNA and DNA |
|---|---|
| 要素 |
|
-超分子 #1000: Pyrococcus furiosus DNA Ligase bound to PCNA and DNA
| 超分子 | 名称: Pyrococcus furiosus DNA Ligase bound to PCNA and DNA タイプ: sample / ID: 1000 / 詳細: The sample was monodisperse / 集合状態: Heteropentamer / Number unique components: 3 |
|---|---|
| 分子量 | 理論値: 160 KDa 手法: One DNA Ligase binds to three PCNA and one nicked DNA |
-分子 #1: Pyrococcus furiosus DNA Ligase
| 分子 | 名称: Pyrococcus furiosus DNA Ligase / タイプ: protein_or_peptide / ID: 1 / Name.synonym: DNA Ligase / 組換発現: No |
|---|---|
| 由来(天然) | 生物種:   Pyrococcus furiosus (古細菌) Pyrococcus furiosus (古細菌) |
-分子 #2: Proliferating cell nuclear antigen
| 分子 | 名称: Proliferating cell nuclear antigen / タイプ: protein_or_peptide / ID: 2 / Name.synonym: PCNA / コピー数: 3 / 集合状態: Trimeric / 組換発現: Yes |
|---|---|
| 由来(天然) | 生物種:   Pyrococcus furiosus (古細菌) Pyrococcus furiosus (古細菌) |
| 分子量 | 理論値: 28 KDa |
-分子 #3: PCNA-nicked DNA
| 分子 | 名称: PCNA-nicked DNA / タイプ: dna / ID: 3 / Name.synonym: DNA / 分類: DNA / Structure: DOUBLE HELIX / Synthetic?: Yes |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
-実験情報
-構造解析
| 手法 | ネガティブ染色法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.02 mg/mL |
|---|---|
| 緩衝液 | pH: 6.5 / 詳細: 20 mM MES,50 mM NaCl,10 mM MgCl2,0.1 mM ATP |
| 染色 | タイプ: NEGATIVE 詳細: The sample solution was applied to a copper grid supporting a continuous thin-carbon film, left for 1 min, then stained with 3 drops of 2% uranyl acetate, and air dried. |
| 凍結 | 凍結剤: NONE / 装置: OTHER |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | JEOL 1010 |
|---|---|
| アライメント法 | Legacy - 非点収差: Objective lens astigmatism was corrected at 400,000 times magnification |
| 撮影 | カテゴリ: CCD / フィルム・検出器のモデル: GENERIC GATAN / 実像数: 400 |
| Tilt angle min | 0 |
| Tilt angle max | 0 |
| 電子線 | 加速電圧: 100 kV / 電子線源: TUNGSTEN HAIRPIN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 5.6 mm / 倍率(公称値): 400000 |
| 試料ステージ | 試料ホルダー: Eucentric / 試料ホルダーモデル: OTHER |
- 画像解析
画像解析
| 最終 再構成 | 想定した対称性 - 点群: C1 (非対称) / アルゴリズム: OTHER / 解像度のタイプ: BY AUTHOR / 解像度: 17.0 Å / 解像度の算出法: FSC 0.5 CUT-OFF / ソフトウェア - 名称: EMAN,IMAGIC / 使用した粒子像数: 19544 |
|---|
-原子モデル構築 1
| 初期モデル | PDB ID: Chain - Chain ID: A |
|---|---|
| 詳細 | PDBEntryID_givenInChain. |
| 精密化 | 空間: REAL |
-原子モデル構築 2
| 初期モデル | PDB ID: Chain - Chain ID: A |
|---|---|
| 詳細 | PDBEntryID_givenInChain. |
| 精密化 | 空間: REAL |
 ムービー
ムービー コントローラー
コントローラー



 UCSF Chimera
UCSF Chimera








 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)