+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | SARS-CoV-2 S 2P trimer in complex with monovalent DARPin R2 (State 1) - Consensus Map | |||||||||
 マップデータ マップデータ | DeepEMhancer sharpened map | |||||||||
 試料 試料 |
| |||||||||
| 生物種 |   | |||||||||
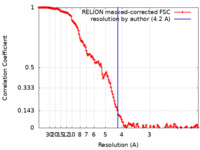
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 4.2 Å | |||||||||
 データ登録者 データ登録者 | Hurdiss DL / Drulyte I | |||||||||
| 資金援助 |  スイス, 1件 スイス, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Biotechnol / 年: 2022 ジャーナル: Nat Biotechnol / 年: 2022タイトル: The trispecific DARPin ensovibep inhibits diverse SARS-CoV-2 variants. 著者: Sylvia Rothenberger / Daniel L Hurdiss / Marcel Walser / Francesca Malvezzi / Jennifer Mayor / Sarah Ryter / Hector Moreno / Nicole Liechti / Andreas Bosshart / Chloé Iss / Valérie Calabro ...著者: Sylvia Rothenberger / Daniel L Hurdiss / Marcel Walser / Francesca Malvezzi / Jennifer Mayor / Sarah Ryter / Hector Moreno / Nicole Liechti / Andreas Bosshart / Chloé Iss / Valérie Calabro / Andreas Cornelius / Tanja Hospodarsch / Alexandra Neculcea / Thamar Looser / Anja Schlegel / Simon Fontaine / Denis Villemagne / Maria Paladino / Dieter Schiegg / Susanne Mangold / Christian Reichen / Filip Radom / Yvonne Kaufmann / Doris Schaible / Iris Schlegel / Christof Zitt / Gabriel Sigrist / Marcel Straumann / Julia Wolter / Marco Comby / Feyza Sacarcelik / Ieva Drulyte / Heyrhyoung Lyoo / Chunyan Wang / Wentao Li / Wenjuan Du / H Kaspar Binz / Rachel Herrup / Sabrina Lusvarghi / Sabari Nath Neerukonda / Russell Vassell / Wei Wang / Julia M Adler / Kathrin Eschke / Mariana Nascimento / Azza Abdelgawad / Achim D Gruber / Judith Bushe / Olivia Kershaw / Charles G Knutson / Kamal K Balavenkatraman / Krishnan Ramanathan / Emanuel Wyler / Luiz Gustavo Teixeira Alves / Seth Lewis / Randall Watson / Micha A Haeuptle / Alexander Zürcher / Keith M Dawson / Daniel Steiner / Carol D Weiss / Patrick Amstutz / Frank J M van Kuppeveld / Michael T Stumpp / Berend-Jan Bosch / Olivier Engler / Jakob Trimpert /     要旨: The emergence of severe acute respiratory syndrome coronavirus 2 (SARS-CoV-2) variants with potential resistance to existing drugs emphasizes the need for new therapeutic modalities with broad ...The emergence of severe acute respiratory syndrome coronavirus 2 (SARS-CoV-2) variants with potential resistance to existing drugs emphasizes the need for new therapeutic modalities with broad variant activity. Here we show that ensovibep, a trispecific DARPin (designed ankyrin repeat protein) clinical candidate, can engage the three units of the spike protein trimer of SARS-CoV-2 and inhibit ACE2 binding with high potency, as revealed by cryo-electron microscopy analysis. The cooperative binding together with the complementarity of the three DARPin modules enable ensovibep to inhibit frequent SARS-CoV-2 variants, including Omicron sublineages BA.1 and BA.2. In Roborovski dwarf hamsters infected with SARS-CoV-2, ensovibep reduced fatality similarly to a standard-of-care monoclonal antibody (mAb) cocktail. When used as a single agent in viral passaging experiments in vitro, ensovibep reduced the emergence of escape mutations in a similar fashion to the same mAb cocktail. These results support further clinical evaluation of ensovibep as a broad variant alternative to existing targeted therapies for Coronavirus Disease 2019 (COVID-19). | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_14810.map.gz emd_14810.map.gz | 210.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-14810-v30.xml emd-14810-v30.xml emd-14810.xml emd-14810.xml | 24.8 KB 24.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_14810_fsc.xml emd_14810_fsc.xml | 14.2 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_14810.png emd_14810.png | 90.5 KB | ||
| その他 |  emd_14810_additional_1.map.gz emd_14810_additional_1.map.gz emd_14810_half_map_1.map.gz emd_14810_half_map_1.map.gz emd_14810_half_map_2.map.gz emd_14810_half_map_2.map.gz | 194.7 MB 195.5 MB 195.3 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-14810 http://ftp.pdbj.org/pub/emdb/structures/EMD-14810 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-14810 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-14810 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_14810.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_14810.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | DeepEMhancer sharpened map | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.045 Å | ||||||||||||||||||||||||||||||||||||
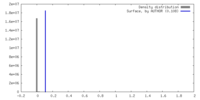
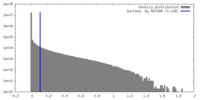
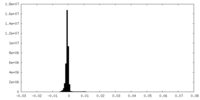
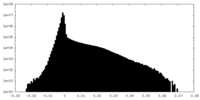
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-追加マップ: Unsharpened map
| ファイル | emd_14810_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Unsharpened map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half map 1
| ファイル | emd_14810_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map 1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half map 2
| ファイル | emd_14810_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map 2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : SARS-CoV-2 S 2P trimer in complex with monovalent DARPin R2
| 全体 | 名称: SARS-CoV-2 S 2P trimer in complex with monovalent DARPin R2 |
|---|---|
| 要素 |
|
-超分子 #1: SARS-CoV-2 S 2P trimer in complex with monovalent DARPin R2
| 超分子 | 名称: SARS-CoV-2 S 2P trimer in complex with monovalent DARPin R2 タイプ: complex / ID: 1 / キメラ: Yes / 親要素: 0 / 含まれる分子: all |
|---|---|
| 分子量 | 理論値: 464 KDa |
-超分子 #2: SARS-CoV-2 spike glycoprotein
| 超分子 | 名称: SARS-CoV-2 spike glycoprotein / タイプ: complex / ID: 2 / 親要素: 1 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  株: Severe acute respiratory syndrome coronavirus 2 (Wuhan-Hu-1) |
-超分子 #3: monovalent DARPin R2
| 超分子 | 名称: monovalent DARPin R2 / タイプ: complex / ID: 3 / 親要素: 1 / 含まれる分子: #2 |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
-分子 #1: SARS-CoV-2 spike glycoprotein
| 分子 | 名称: SARS-CoV-2 spike glycoprotein / タイプ: protein_or_peptide / ID: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MFVFLVLLPL VSSQCVNLTT RTQLPPAYT NSFTRGVYYP D KVFRSSVL HSTQDLFLPF FS NVTWFHA IHVSGTNGTK RFD NPVLPF NDGVYFASTE KSNI IRGWI FGTTLDSKTQ SLLIV NNAT NVVIKVCEFQ FCNDPF LGV YYHKNNKSWM ESEFRVY SS ...文字列: MFVFLVLLPL VSSQCVNLTT RTQLPPAYT NSFTRGVYYP D KVFRSSVL HSTQDLFLPF FS NVTWFHA IHVSGTNGTK RFD NPVLPF NDGVYFASTE KSNI IRGWI FGTTLDSKTQ SLLIV NNAT NVVIKVCEFQ FCNDPF LGV YYHKNNKSWM ESEFRVY SS ANNCTFEYVS QPFLMDLE G KQGNFKNLRE FVFKNIDGY FKIYSKHTPI NLVRDLPQGF SALEPLVDL PIGINITRFQ T LLALHRSY LTPGDSSSGW TA GAAAYYV GYLQPRTFLL KYN ENGTIT DAVDCALDPL SETK CTLKS FTVEKGIYQT SNFRV QPTE SIVRFPNITN LCPFGE VFN ATRFASVYAW NRKRISN CV ADYSVLYNSA SFSTFKCY G VSPTKLNDLC FTNVYADSF VIRGDEVRQI APGQTGKIAD YNYKLPDDF TGCVIAWNSN N LDSKVGGN YNYLYRLFRK SN LKPFERD ISTEIYQAGS TPC NGVEGF NCYFPLQSYG FQPT NGVGY QPYRVVVLSF ELLHA PATV CGPKKSTNLV KNKCVN FNF NGLTGTGVLT ESNKKFL PF QQFGRDIADT TDAVRDPQ T LEILDITPCS FGGVSVITP GTNTSNQVAV LYQDVNCTEV PVAIHADQL TPTWRVYSTG S NVFQTRAG CLIGAEHVNN SY ECDIPIG AGICASYQTQ TNS PAAARS VASQSIIAYT MSLG AENSV AYSNNSIAIP TNFTI SVTT EILPVSMTKT SVDCTM YIC GDSTECSNLL LQYGSFC TQ LNRALTGIAV EQDKNTQE V FAQVKQIYKT PPIKDFGGF NFSQILPDPS KPSKRSFIED LLFNKVTLA DAGFIKQYGD C LGDIAARD LICAQKFNGL TV LPPLLTD EMIAQYTSAL LAG TITSGW TFGAGAALQI PFAM QMAYR FNGIGVTQNV LYENQ KLIA NQFNSAIGKI QDSLSS TAS ALGKLQDVVN QNAQALN TL VKQLSSNFGA ISSVLNDI L SRLDPPEAEV QIDRLITGR LQSLQTYVTQ QLIRAAEIRA SANLAATKM SECVLGQSKR V DFCGKGYH LMSFPQSAPH GV VFLHVTY VPAQEKNFTT APA ICHDGK AHFPREGVFV SNGT HWFVT QRNFYEPQII TTDNT FVSG NCDVVIGIVN NTVYDP LQP ELDSFKEELD KYFKNHT SP DVDLGDISGI NASVVNIQ K EIDRLNEVAK NLNESLIDL LIKGSGYIPE APRDGQAYVR KDGEWVLLS TFLIKLVPRG S LEWSHPQF EK |
-分子 #2: monovalent DARPin R2
| 分子 | 名称: monovalent DARPin R2 / タイプ: protein_or_peptide / ID: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MRGSHHHHHH GSDLGKKLLQ AARAGQLDEV RELLKAGADV NAKDREGKTP LHVAAQEGHL EIVEVLLKAG ADVNAKDVWG RTPLHLAAWI GHLEIVEVLL KAGADVNAKD VSGATPLHAA ALHGHLEIVE VLLNAGADVN AQDKSGKTPA DLAARAGHQD IAEVLQKAA |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 2.5 mg/mL |
|---|---|
| 緩衝液 | pH: 8 |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 95 % / チャンバー内温度: 277 K |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 詳細 | Data collected with a 30 degree stage tilt. |
| 撮影 | フィルム・検出器のモデル: FEI FALCON IV (4k x 4k) デジタル化 - サイズ - 横: 4096 pixel / デジタル化 - サイズ - 縦: 4096 pixel / 撮影したグリッド数: 1 / 実像数: 3024 / 平均電子線量: 40.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 50.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 1.25 µm / 倍率(公称値): 75000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー







 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)