+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
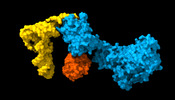
| タイトル | Cryo-EM structure of aIF1A:aIF5B:Met-tRNAiMet complex from a Pyrococcus abyssi 30S initiation complex | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Initiation complex / translation initiation / small ribosomal subunit / aIF5b / TRANSLATION | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報UDP phosphatase activity / GDP phosphatase activity / proteoglycan biosynthetic process / intron homing / intein-mediated protein splicing / translation initiation factor activity / endonuclease activity / GTPase activity / calcium ion binding / GTP binding ...UDP phosphatase activity / GDP phosphatase activity / proteoglycan biosynthetic process / intron homing / intein-mediated protein splicing / translation initiation factor activity / endonuclease activity / GTPase activity / calcium ion binding / GTP binding / RNA binding / cytoplasm 類似検索 - 分子機能 | |||||||||
| 生物種 |    Pyrococcus abyssi GE5 (古細菌) Pyrococcus abyssi GE5 (古細菌) | |||||||||
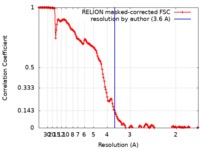
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.6 Å | |||||||||
 データ登録者 データ登録者 | Coureux PD / Bourgeois G / Mechulam Y / Schmitt E / Kazan R | |||||||||
| 資金援助 |  フランス, 1件 フランス, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nucleic Acids Res / 年: 2022 ジャーナル: Nucleic Acids Res / 年: 2022タイトル: Role of aIF5B in archaeal translation initiation. 著者: Ramy Kazan / Gabrielle Bourgeois / Christine Lazennec-Schurdevin / Eric Larquet / Yves Mechulam / Pierre-Damien Coureux / Emmanuelle Schmitt /  要旨: In eukaryotes and in archaea late steps of translation initiation involve the two initiation factors e/aIF5B and e/aIF1A. In eukaryotes, the role of eIF5B in ribosomal subunit joining is established ...In eukaryotes and in archaea late steps of translation initiation involve the two initiation factors e/aIF5B and e/aIF1A. In eukaryotes, the role of eIF5B in ribosomal subunit joining is established and structural data showing eIF5B bound to the full ribosome were obtained. To achieve its function, eIF5B collaborates with eIF1A. However, structural data illustrating how these two factors interact on the small ribosomal subunit have long been awaited. The role of the archaeal counterparts, aIF5B and aIF1A, remains to be extensively addressed. Here, we study the late steps of Pyrococcus abyssi translation initiation. Using in vitro reconstituted initiation complexes and light scattering, we show that aIF5B bound to GTP accelerates subunit joining without the need for GTP hydrolysis. We report the crystallographic structures of aIF5B bound to GDP and GTP and analyze domain movements associated to these two nucleotide states. Finally, we present the cryo-EM structure of an initiation complex containing 30S bound to mRNA, Met-tRNAiMet, aIF5B and aIF1A at 2.7 Å resolution. Structural data shows how archaeal 5B and 1A factors cooperate to induce a conformation of the initiator tRNA favorable to subunit joining. Archaeal and eukaryotic features of late steps of translation initiation are discussed. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_14763.map.gz emd_14763.map.gz | 288.6 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-14763-v30.xml emd-14763-v30.xml emd-14763.xml emd-14763.xml | 25.2 KB 25.2 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_14763_fsc.xml emd_14763_fsc.xml | 15.3 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_14763.png emd_14763.png | 55.6 KB | ||
| Filedesc metadata |  emd-14763.cif.gz emd-14763.cif.gz | 7.3 KB | ||
| その他 |  emd_14763_additional_1.map.gz emd_14763_additional_1.map.gz emd_14763_half_map_1.map.gz emd_14763_half_map_1.map.gz emd_14763_half_map_2.map.gz emd_14763_half_map_2.map.gz | 201.7 MB 202.5 MB 202.4 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-14763 http://ftp.pdbj.org/pub/emdb/structures/EMD-14763 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-14763 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-14763 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_14763_validation.pdf.gz emd_14763_validation.pdf.gz | 1002.3 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_14763_full_validation.pdf.gz emd_14763_full_validation.pdf.gz | 1001.9 KB | 表示 | |
| XML形式データ |  emd_14763_validation.xml.gz emd_14763_validation.xml.gz | 22.8 KB | 表示 | |
| CIF形式データ |  emd_14763_validation.cif.gz emd_14763_validation.cif.gz | 30.2 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-14763 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-14763 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-14763 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-14763 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  7zkiMC  7yypC  7yznC  7zagC  7zahC  7zaiC  7zhgC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_14763.map.gz / 形式: CCP4 / 大きさ: 307.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_14763.map.gz / 形式: CCP4 / 大きさ: 307.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.86 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-追加マップ: Not sharpened map
| ファイル | emd_14763_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Not sharpened map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_14763_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_14763_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : tRNA-aIF5B-aIF1A moiety of a Pyrococcus abyssi translation initia...
| 全体 | 名称: tRNA-aIF5B-aIF1A moiety of a Pyrococcus abyssi translation initiation complex with 30S ribosomal subunit,tRNA, mRNA and initiation factors 1A and 5B. |
|---|---|
| 要素 |
|
-超分子 #1: tRNA-aIF5B-aIF1A moiety of a Pyrococcus abyssi translation initia...
| 超分子 | 名称: tRNA-aIF5B-aIF1A moiety of a Pyrococcus abyssi translation initiation complex with 30S ribosomal subunit,tRNA, mRNA and initiation factors 1A and 5B. タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#3 |
|---|
-超分子 #2: tRNA-Met
| 超分子 | 名称: tRNA-Met / タイプ: complex / ID: 2 / 親要素: 1 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  |
-超分子 #3: Translation initiation factor 1A and Probable translation initiat...
| 超分子 | 名称: Translation initiation factor 1A and Probable translation initiation factor IF-2 タイプ: complex / ID: 3 / 親要素: 1 / 含まれる分子: #2-#3 |
|---|---|
| 由来(天然) | 生物種:   Pyrococcus abyssi GE5 (古細菌) Pyrococcus abyssi GE5 (古細菌) |
-分子 #1: Met-tRNAiMet
| 分子 | 名称: Met-tRNAiMet / タイプ: rna / ID: 1 / コピー数: 1 |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 24.833904 KDa |
| 配列 | 文字列: AGCGGGG(4SU)GG AGCAGCCUGG (H2U)AGCUCGUCG GG(OMC)UCAUAAC CCGAAGAUCG UCGG(5MU)(PSU)CAAA UCCGGCCCC CGCUACCA GENBANK: GENBANK: CP026027.1 |
-分子 #2: Translation initiation factor 1A
| 分子 | 名称: Translation initiation factor 1A / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Pyrococcus abyssi GE5 (古細菌) / 株: GE5 / Orsay Pyrococcus abyssi GE5 (古細菌) / 株: GE5 / Orsay |
| 分子量 | 理論値: 15.336709 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MGSSSHHHHH HSSGLVPRGS HMPKKERKVE GDEVIRVPLP EGNQLFGVVE QALGAGWMDV RCEDGKIRRC RIPGKLRRRV WIRVGDLVI VQPWPVQSDK RGDIVYRYTQ TQVDWLLRKG KITQEFLTGG SLLVE UniProtKB: Translation initiation factor 1A |
-分子 #3: Translation initiation factor 5B
| 分子 | 名称: Translation initiation factor 5B / タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Pyrococcus abyssi GE5 (古細菌) / 株: GE5 / Orsay Pyrococcus abyssi GE5 (古細菌) / 株: GE5 / Orsay |
| 分子量 | 理論値: 69.122945 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MGSSHHHHHH SSGLVPRGSH MMTKRIRQPI IAVLGHVDHG KTTLLDRIRK TNVAAKEAGG ITQHIGATEV PIEVVKKIAG PLIKLWKAE IKLPGLLFID TPGHEAFTSL RARGGSLADL AVLVVDINEG FQPQTIESIE ILRKYRTPFV VAANKIDRIK G WVIEEDEP ...文字列: MGSSHHHHHH SSGLVPRGSH MMTKRIRQPI IAVLGHVDHG KTTLLDRIRK TNVAAKEAGG ITQHIGATEV PIEVVKKIAG PLIKLWKAE IKLPGLLFID TPGHEAFTSL RARGGSLADL AVLVVDINEG FQPQTIESIE ILRKYRTPFV VAANKIDRIK G WVIEEDEP FLMNIKKQDQ RAVQELETKL WELIGKFYEF GFQANRFDRV QNFTRELAIV PISAKYGIGI AELLVLIAGL SQ RYLEEKL KIEVEGPARG TILEVREEPG LGHTIDVIIY DGTLHKDDTI VVGGKDKAIV TKIRALLKPK PLDEIRDPRF RFD YVDEVT AAAGVKIAAP GLEEALAGSP VIAAPTPEDV EKAKQEILEQ IERVVISTDK VGVIVKADTL GSLEALSKEL QEKE IPIRK ADVGNVSKTD VMEALSVKEE EPKYGVILGF NVKVNEDAEE VAKAKDVKIF VGNVIYKLIE DYEEWVKEEE EKKKR ELLS KVTFPGVIRL YPDERYVFRR SNPAIVGIEV IEGRIKPGVT LIKQNGQKVG VIRSIKSRDE FLQEAKKGQA VAIAIE GAI VGRHIHPGET LYVDLSRDDA ITLLKHLRDT LEDTDIKALK MIAKVKAKED PFWRAI UniProtKB: Apyrase, Probable translation initiation factor IF-2, Probable translation initiation factor IF-2 |
-分子 #4: METHIONINE
| 分子 | 名称: METHIONINE / タイプ: ligand / ID: 4 / コピー数: 1 / 式: MET |
|---|---|
| 分子量 | 理論値: 149.211 Da |
| Chemical component information |  ChemComp-MET: |
-分子 #5: PHOSPHOAMINOPHOSPHONIC ACID-GUANYLATE ESTER
| 分子 | 名称: PHOSPHOAMINOPHOSPHONIC ACID-GUANYLATE ESTER / タイプ: ligand / ID: 5 / コピー数: 1 / 式: GNP |
|---|---|
| 分子量 | 理論値: 522.196 Da |
| Chemical component information |  ChemComp-GNP: |
-分子 #6: MAGNESIUM ION
| 分子 | 名称: MAGNESIUM ION / タイプ: ligand / ID: 6 / コピー数: 1 / 式: MG |
|---|---|
| 分子量 | 理論値: 24.305 Da |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 6.7 |
|---|---|
| グリッド | モデル: Quantifoil R2/1 / 材質: COPPER / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: CONTINUOUS / 支持フィルム - Film thickness: 2 |
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | TFS KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 平均電子線量: 39.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 0.8 µm |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー














 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)