+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-1471 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | The Structural Basis of Membrane Invagination by F-BAR Domains | |||||||||
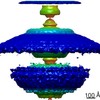
 マップデータ マップデータ | 3D reconstruction of a membrane tubule coated by a helical lattice of dimeric F-BAR modules from the human protein CIP4. | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | F-BAR / FCH / PCH / CIP4 / FBP17 / Toca / Membrane Tubule. Human Protein. | |||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
| 手法 | らせん対称体再構成法 / クライオ電子顕微鏡法 / 解像度: 17.0 Å | |||||||||
 データ登録者 データ登録者 | Frost A / Perera R / Roux A / Spasov K / Egelman E / De Camilli P / Unger V | |||||||||
 引用 引用 |  ジャーナル: Cell / 年: 2008 ジャーナル: Cell / 年: 2008タイトル: Structural basis of membrane invagination by F-BAR domains. 著者: Adam Frost / Rushika Perera / Aurélien Roux / Krasimir Spasov / Olivier Destaing / Edward H Egelman / Pietro De Camilli / Vinzenz M Unger /  要旨: BAR superfamily domains shape membranes through poorly understood mechanisms. We solved structures of F-BAR modules bound to flat and curved bilayers using electron (cryo)microscopy. We show that ...BAR superfamily domains shape membranes through poorly understood mechanisms. We solved structures of F-BAR modules bound to flat and curved bilayers using electron (cryo)microscopy. We show that membrane tubules form when F-BARs polymerize into helical coats that are held together by lateral and tip-to-tip interactions. On gel-state membranes or after mutation of residues along the lateral interaction surface, F-BARs adsorb onto bilayers via surfaces other than their concave face. We conclude that membrane binding is separable from membrane bending, and that imposition of the module's concave surface forces fluid-phase bilayers to bend locally. Furthermore, exposure of the domain's lateral interaction surface through a change in orientation serves as the crucial trigger for assembly of the helical coat and propagation of bilayer bending. The geometric constraints and sequential assembly of the helical lattice explain how F-BAR and classical BAR domains segregate into distinct microdomains, and provide insight into the spatial regulation of membrane invagination. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_1471.map.gz emd_1471.map.gz | 62.5 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-1471-v30.xml emd-1471-v30.xml emd-1471.xml emd-1471.xml | 8.3 KB 8.3 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  1471.gif 1471.gif | 79.7 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-1471 http://ftp.pdbj.org/pub/emdb/structures/EMD-1471 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-1471 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-1471 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_1471_validation.pdf.gz emd_1471_validation.pdf.gz | 286.4 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_1471_full_validation.pdf.gz emd_1471_full_validation.pdf.gz | 285.5 KB | 表示 | |
| XML形式データ |  emd_1471_validation.xml.gz emd_1471_validation.xml.gz | 7.3 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1471 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1471 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1471 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1471 | HTTPS FTP |
-関連構造データ
| 類似構造データ |
|---|
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_1471.map.gz / 形式: CCP4 / 大きさ: 238.4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_1471.map.gz / 形式: CCP4 / 大きさ: 238.4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | 3D reconstruction of a membrane tubule coated by a helical lattice of dimeric F-BAR modules from the human protein CIP4. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 2.28 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : membrane tubule coated with a helical lattice of dimer F-BAR modu...
| 全体 | 名称: membrane tubule coated with a helical lattice of dimer F-BAR modules from the human protein CIP4 |
|---|---|
| 要素 |
|
-超分子 #1000: membrane tubule coated with a helical lattice of dimer F-BAR modu...
| 超分子 | 名称: membrane tubule coated with a helical lattice of dimer F-BAR modules from the human protein CIP4 タイプ: sample / ID: 1000 詳細: membrane composed of phospholipids and cholesterol. recombinant protein Number unique components: 2 |
|---|
-分子 #1: CIP4
| 分子 | 名称: CIP4 / タイプ: protein_or_peptide / ID: 1 / Name.synonym: CIP4 / 組換発現: Yes |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) / 別称: Human / 細胞中の位置: plasmsa membrane and cytoplasm Homo sapiens (ヒト) / 別称: Human / 細胞中の位置: plasmsa membrane and cytoplasm |
| 組換発現 | 生物種:  |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | らせん対称体再構成法 |
| 試料の集合状態 | filament |
- 試料調製
試料調製
| 凍結 | 凍結剤: ETHANE / 装置: OTHER |
|---|
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TECNAI F20 |
|---|---|
| 日付 | 2006年1月3日 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD |
| 試料ステージ | 試料ホルダー: eucentric, single tilt / 試料ホルダーモデル: GATAN LIQUID NITROGEN |
| 実験機器 |  モデル: Tecnai F20 / 画像提供: FEI Company |
- 画像解析
画像解析
| 最終 再構成 | 想定した対称性 - らせんパラメータ - 軸対称性: C2 (2回回転対称) アルゴリズム: OTHER / 解像度のタイプ: BY AUTHOR / 解像度: 17.0 Å / 解像度の算出法: OTHER / ソフトウェア - 名称: SPIDER, MRC, IHRSR |
|---|---|
| CTF補正 | 詳細: ACE, image |
 ムービー
ムービー コントローラー
コントローラー





 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)























