+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | SLFN11 E209A monomer | |||||||||
 マップデータ マップデータ | sharpened map | |||||||||
 試料 試料 |
| |||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
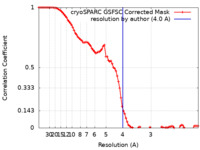
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 4.0 Å | |||||||||
 データ登録者 データ登録者 | Metzner FJ / Kugler M / Wenzl SJ / Lammens K | |||||||||
| 資金援助 |  ドイツ, European Union, 2件 ドイツ, European Union, 2件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2022 ジャーナル: Nat Commun / 年: 2022タイトル: Mechanistic understanding of human SLFN11. 著者: Felix J Metzner / Simon J Wenzl / Michael Kugler / Stefan Krebs / Karl-Peter Hopfner / Katja Lammens /  要旨: Schlafen 11 (SLFN11) is an interferon-inducible antiviral restriction factor with tRNA endoribonuclease and DNA binding functions. It is recruited to stalled replication forks in response to ...Schlafen 11 (SLFN11) is an interferon-inducible antiviral restriction factor with tRNA endoribonuclease and DNA binding functions. It is recruited to stalled replication forks in response to replication stress and inhibits replication of certain viruses such as the human immunodeficiency virus 1 (HIV-1) by modulating the tRNA pool. SLFN11 has been identified as a predictive biomarker in cancer, as its expression correlates with a beneficial response to DNA damage inducing anticancer drugs. However, the mechanism and interdependence of these two functions are largely unknown. Here, we present cryo-electron microscopy (cryo-EM) structures of human SLFN11 in its dimeric apoenzyme state, bound to tRNA and in complex with single-strand DNA. Full-length SLFN11 neither hydrolyses nor binds ATP and the helicase domain appears in an autoinhibited state. Together with biochemical and structure guided mutagenesis studies, our data give detailed insights into the mechanism of endoribonuclease activity as well as suggestions on how SLFN11 may block stressed replication forks. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
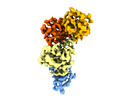
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_14693.map.gz emd_14693.map.gz | 59.7 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-14693-v30.xml emd-14693-v30.xml emd-14693.xml emd-14693.xml | 14.1 KB 14.1 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_14693_fsc.xml emd_14693_fsc.xml | 8.5 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_14693.png emd_14693.png | 55.2 KB | ||
| マスクデータ |  emd_14693_msk_1.map emd_14693_msk_1.map | 64 MB |  マスクマップ マスクマップ | |
| その他 |  emd_14693_half_map_1.map.gz emd_14693_half_map_1.map.gz emd_14693_half_map_2.map.gz emd_14693_half_map_2.map.gz | 59.3 MB 59.3 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-14693 http://ftp.pdbj.org/pub/emdb/structures/EMD-14693 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-14693 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-14693 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_14693_validation.pdf.gz emd_14693_validation.pdf.gz | 644.5 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_14693_full_validation.pdf.gz emd_14693_full_validation.pdf.gz | 644.1 KB | 表示 | |
| XML形式データ |  emd_14693_validation.xml.gz emd_14693_validation.xml.gz | 16.4 KB | 表示 | |
| CIF形式データ |  emd_14693_validation.cif.gz emd_14693_validation.cif.gz | 21.2 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-14693 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-14693 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-14693 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-14693 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_14693.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_14693.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | sharpened map | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.046 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_14693_msk_1.map emd_14693_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: half map A
| ファイル | emd_14693_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half map A | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: half map B
| ファイル | emd_14693_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half map B | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : SLFN11 E209A monomer
| 全体 | 名称: SLFN11 E209A monomer |
|---|---|
| 要素 |
|
-超分子 #1: SLFN11 E209A monomer
| 超分子 | 名称: SLFN11 E209A monomer / タイプ: organelle_or_cellular_component / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 組換発現 | 生物種:  Trichoplusia ni (イラクサキンウワバ) Trichoplusia ni (イラクサキンウワバ) |
-分子 #1: SLFN11 E209A
| 分子 | 名称: SLFN11 E209A / タイプ: protein_or_peptide / ID: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 組換発現 | 生物種:  Trichoplusia ni (イラクサキンウワバ) Trichoplusia ni (イラクサキンウワバ) |
| 配列 | 文字列: MADYKDDDDK GTDYKDDDDK LEVLFQGPME ANQCPLVVEP SYPDLVINVG EVTLGEENRK KLQKIQRDQE KERVMRAACA LLNSGGGVIR MAKKVEHPVE MGLDLEQSLR ELIQSSDLQA FFETKQQGRC FYIFVKSWSS GPFPEDRSVK PRLCSLSSSL YRRSETSVRS ...文字列: MADYKDDDDK GTDYKDDDDK LEVLFQGPME ANQCPLVVEP SYPDLVINVG EVTLGEENRK KLQKIQRDQE KERVMRAACA LLNSGGGVIR MAKKVEHPVE MGLDLEQSLR ELIQSSDLQA FFETKQQGRC FYIFVKSWSS GPFPEDRSVK PRLCSLSSSL YRRSETSVRS MDSREAFCFL KTKRKPKILE EGPFHKIHKG VYQELPNSDP ADPNSDPADL IFQKDYLEYG EILPFPASQL VEFKQFSTKH FQEYVKRTIP EYVPAFANTG GGYLFIGVDD KSREVLGCAK ENVDPDSLRR KIEQAIYKLP CVHFCQPQRP ITFTLKIVNV LKRGELYGYA CMIRVNPFCC AVFSEAPNSW IVEDKYVCSL TTEKWVGMMT DTDPDLLQLS EDFECQLSLS SGPPLSRPVY SKKGLEHKKE LQQLLFSVPP GYLRYTPESL WRDLISEHRG LEELINKQMQ PFFRGILIFS RSWAVDLNLQ EKPGVICDAL LIAQNSTPIL YTILREQDAE GQDYCTRTAF TLKQKLVNMG GYTGKVCVRA KVLCLSPESS AEALEAAVSP MDYPASYSLA GTQHMEALLQ SLVIVLLGFR SLLSDQLGCE VLNLLTAQQY EIFSRSLRKN RELFVHGLPG SGKTIMAMKI MEKIRNVFHC EAHRILYVCE NQPLRNFISD RNICRAETRK TFLRENFEHI QHIVIDEAQN FRTEDGDWYG KAKSITRRAK GGPGILWIFL DYFQTSHLDC SGLPPLSDQY PREELTRIVR NADPIAKYLQ KEMQVIRSNP SFNIPTGCLE VFPEAEWSQG VQGTLRIKKY LTVEQIMTCV ADTCRRFFDR GYSPKDVAVL VSTAKEVEHY KYELLKAMRK KRVVQLSDAC DMLGDHIVLD SVRRFSGLER SIVFGIHPRT ADPAILPNVL ICLASRAKQH LYIFPWGGH |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 9 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 平均電子線量: 43.58 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.9 µm / 最小 デフォーカス(公称値): 1.1 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー










 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)