+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | CAND1-CUL1-RBX1 | |||||||||
 マップデータ マップデータ | CAND1-CUL1-RBX1 RELION postprocess | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | cullin-RING E3 ubiquitin ligase / Assembly factor / CAND1 / LIGASE | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報SCF complex assembly / Parkin-FBXW7-Cul1 ubiquitin ligase complex / negative regulation of catalytic activity / cullin-RING-type E3 NEDD8 transferase / NEDD8 transferase activity / cullin-RING ubiquitin ligase complex / cellular response to chemical stress / Cul7-RING ubiquitin ligase complex / ubiquitin-dependent protein catabolic process via the C-end degron rule pathway / Loss of Function of FBXW7 in Cancer and NOTCH1 Signaling ...SCF complex assembly / Parkin-FBXW7-Cul1 ubiquitin ligase complex / negative regulation of catalytic activity / cullin-RING-type E3 NEDD8 transferase / NEDD8 transferase activity / cullin-RING ubiquitin ligase complex / cellular response to chemical stress / Cul7-RING ubiquitin ligase complex / ubiquitin-dependent protein catabolic process via the C-end degron rule pathway / Loss of Function of FBXW7 in Cancer and NOTCH1 Signaling / positive regulation of protein autoubiquitination / RNA polymerase II transcription initiation surveillance / protein neddylation / NEDD8 ligase activity / VCB complex / negative regulation of response to oxidative stress / Cul5-RING ubiquitin ligase complex / SCF ubiquitin ligase complex / Cul2-RING ubiquitin ligase complex / ubiquitin-ubiquitin ligase activity / negative regulation of type I interferon production / Cul4A-RING E3 ubiquitin ligase complex / SCF-dependent proteasomal ubiquitin-dependent protein catabolic process / Cul4-RING E3 ubiquitin ligase complex / Cul3-RING ubiquitin ligase complex / Cul4B-RING E3 ubiquitin ligase complex / ubiquitin ligase complex scaffold activity / negative regulation of mitophagy / Prolactin receptor signaling / cullin family protein binding / positive regulation of RNA polymerase II transcription preinitiation complex assembly / protein monoubiquitination / ubiquitin ligase complex / protein K48-linked ubiquitination / Nuclear events stimulated by ALK signaling in cancer / Regulation of BACH1 activity / MAP3K8 (TPL2)-dependent MAPK1/3 activation / transcription-coupled nucleotide-excision repair / regulation of cellular response to insulin stimulus / positive regulation of TORC1 signaling / post-translational protein modification / intrinsic apoptotic signaling pathway / negative regulation of insulin receptor signaling pathway / NIK-->noncanonical NF-kB signaling / SCF-beta-TrCP mediated degradation of Emi1 / TBP-class protein binding / T cell activation / Degradation of DVL / Dectin-1 mediated noncanonical NF-kB signaling / animal organ morphogenesis / Iron uptake and transport / Activation of NF-kappaB in B cells / Recognition of DNA damage by PCNA-containing replication complex / Degradation of GLI1 by the proteasome / GSK3B and BTRC:CUL1-mediated-degradation of NFE2L2 / Negative regulation of NOTCH4 signaling / Vif-mediated degradation of APOBEC3G / Hedgehog 'on' state / Degradation of GLI2 by the proteasome / GLI3 is processed to GLI3R by the proteasome / DNA Damage Recognition in GG-NER / FBXL7 down-regulates AURKA during mitotic entry and in early mitosis / cellular response to amino acid stimulus / Degradation of beta-catenin by the destruction complex / Evasion by RSV of host interferon responses / Oxygen-dependent proline hydroxylation of Hypoxia-inducible Factor Alpha / NOTCH1 Intracellular Domain Regulates Transcription / Dual Incision in GG-NER / Transcription-Coupled Nucleotide Excision Repair (TC-NER) / Formation of TC-NER Pre-Incision Complex / Constitutive Signaling by NOTCH1 PEST Domain Mutants / Constitutive Signaling by NOTCH1 HD+PEST Domain Mutants / G1/S transition of mitotic cell cycle / CLEC7A (Dectin-1) signaling / SCF(Skp2)-mediated degradation of p27/p21 / negative regulation of canonical Wnt signaling pathway / Formation of Incision Complex in GG-NER / Regulation of expression of SLITs and ROBOs / FCERI mediated NF-kB activation / RING-type E3 ubiquitin transferase / Interleukin-1 signaling / Orc1 removal from chromatin / Dual incision in TC-NER / Gap-filling DNA repair synthesis and ligation in TC-NER / Regulation of RAS by GAPs / Cyclin D associated events in G1 / Regulation of RUNX2 expression and activity / KEAP1-NFE2L2 pathway / : / protein polyubiquitination / Regulation of PLK1 Activity at G2/M Transition / positive regulation of protein catabolic process / Downstream TCR signaling / Antigen processing: Ubiquitination & Proteasome degradation / cellular response to UV / ubiquitin protein ligase activity / positive regulation of proteasomal ubiquitin-dependent protein catabolic process / Neddylation / MAPK cascade / cellular response to oxidative stress 類似検索 - 分子機能 | |||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
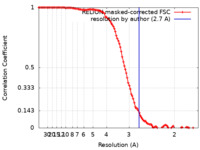
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.7 Å | |||||||||
 データ登録者 データ登録者 | Baek K / Schulman BA | |||||||||
| 資金援助 |  ドイツ, European Union, 2件 ドイツ, European Union, 2件
| |||||||||
 引用 引用 |  ジャーナル: Cell / 年: 2023 ジャーナル: Cell / 年: 2023タイトル: Systemwide disassembly and assembly of SCF ubiquitin ligase complexes. 著者: Kheewoong Baek / Daniel C Scott / Lukas T Henneberg / Moeko T King / Matthias Mann / Brenda A Schulman /   要旨: Cells respond to environmental cues by remodeling their inventories of multiprotein complexes. Cellular repertoires of SCF (SKP1-CUL1-F box protein) ubiquitin ligase complexes, which mediate much ...Cells respond to environmental cues by remodeling their inventories of multiprotein complexes. Cellular repertoires of SCF (SKP1-CUL1-F box protein) ubiquitin ligase complexes, which mediate much protein degradation, require CAND1 to distribute the limiting CUL1 subunit across the family of ∼70 different F box proteins. Yet, how a single factor coordinately assembles numerous distinct multiprotein complexes remains unknown. We obtained cryo-EM structures of CAND1-bound SCF complexes in multiple states and correlated mutational effects on structures, biochemistry, and cellular assays. The data suggest that CAND1 clasps idling catalytic domains of an inactive SCF, rolls around, and allosterically rocks and destabilizes the SCF. New SCF production proceeds in reverse, through SKP1-F box allosterically destabilizing CAND1. The CAND1-SCF conformational ensemble recycles CUL1 from inactive complexes, fueling mixing and matching of SCF parts for E3 activation in response to substrate availability. Our data reveal biogenesis of a predominant family of E3 ligases, and the molecular basis for systemwide multiprotein complex assembly. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
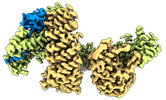
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_14561.map.gz emd_14561.map.gz | 11.2 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-14561-v30.xml emd-14561-v30.xml emd-14561.xml emd-14561.xml | 25.7 KB 25.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_14561_fsc.xml emd_14561_fsc.xml | 11.4 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_14561.png emd_14561.png | 118 KB | ||
| マスクデータ |  emd_14561_msk_1.map emd_14561_msk_1.map | 125 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-14561.cif.gz emd-14561.cif.gz | 7.7 KB | ||
| その他 |  emd_14561_additional_1.map.gz emd_14561_additional_1.map.gz emd_14561_additional_2.map.gz emd_14561_additional_2.map.gz emd_14561_half_map_1.map.gz emd_14561_half_map_1.map.gz emd_14561_half_map_2.map.gz emd_14561_half_map_2.map.gz | 98.5 MB 111.6 MB 98.6 MB 98.6 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-14561 http://ftp.pdbj.org/pub/emdb/structures/EMD-14561 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-14561 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-14561 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_14561_validation.pdf.gz emd_14561_validation.pdf.gz | 702.6 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_14561_full_validation.pdf.gz emd_14561_full_validation.pdf.gz | 702.2 KB | 表示 | |
| XML形式データ |  emd_14561_validation.xml.gz emd_14561_validation.xml.gz | 18.7 KB | 表示 | |
| CIF形式データ |  emd_14561_validation.cif.gz emd_14561_validation.cif.gz | 24.7 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-14561 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-14561 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-14561 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-14561 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  7z8rMC  7z8tC  7z8vC  7zbwC  7zbzC  8cdjC  8cdkC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_14561.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_14561.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | CAND1-CUL1-RBX1 RELION postprocess | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.8512 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_14561_msk_1.map emd_14561_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: CAND1-CUL1-RBX1 3D Refinement
| ファイル | emd_14561_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | CAND1-CUL1-RBX1 3D Refinement | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: CAND1-CUL1-RBX1 deepEMhancer
| ファイル | emd_14561_additional_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | CAND1-CUL1-RBX1 deepEMhancer | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: CAND1-CUL1-RBX1 halfmap1
| ファイル | emd_14561_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | CAND1-CUL1-RBX1 halfmap1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: CAND1-CUL1-RBX1 halfmap2
| ファイル | emd_14561_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | CAND1-CUL1-RBX1 halfmap2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : CAND1-CUL1-RBX1
| 全体 | 名称: CAND1-CUL1-RBX1 |
|---|---|
| 要素 |
|
-超分子 #1: CAND1-CUL1-RBX1
| 超分子 | 名称: CAND1-CUL1-RBX1 / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#3 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
-分子 #1: Cullin-1
| 分子 | 名称: Cullin-1 / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 89.800367 KDa |
| 組換発現 | 生物種:  Trichoplusia ni (イラクサキンウワバ) Trichoplusia ni (イラクサキンウワバ) |
| 配列 | 文字列: MSSTRSQNPH GLKQIGLDQI WDDLRAGIQQ VYTRQSMAKS RYMELYTHVY NYCTSVHQSN QARGAGVPPS KSKKGQTPGG AQFVGLELY KRLKEFLKNY LTNLLKDGED LMDESVLKFY TQQWEDYRFS SKVLNGICAY LNRHWVRREC DEGRKGIYEI Y SLALVTWR ...文字列: MSSTRSQNPH GLKQIGLDQI WDDLRAGIQQ VYTRQSMAKS RYMELYTHVY NYCTSVHQSN QARGAGVPPS KSKKGQTPGG AQFVGLELY KRLKEFLKNY LTNLLKDGED LMDESVLKFY TQQWEDYRFS SKVLNGICAY LNRHWVRREC DEGRKGIYEI Y SLALVTWR DCLFRPLNKQ VTNAVLKLIE KERNGETINT RLISGVVQSY VELGLNEDDA FAKGPTLTVY KESFESQFLA DT ERFYTRE STEFLQQNPV TEYMKKAEAR LLEEQRRVQV YLHESTQDEL ARKCEQVLIE KHLEIFHTEF QNLLDADKNE DLG RMYNLV SRIQDGLGEL KKLLETHIHN QGLAAIEKCG EAALNDPKMY VQTVLDVHKK YNALVMSAFN NDAGFVAALD KACG RFINN NAVTKMAQSS SKSPELLARY CDSLLKKSSK NPEEAELEDT LNQVMVVFKY IEDKDVFQKF YAKMLAKRLV HQNSA SDDA EASMISKLKQ ACGFEYTSKL QRMFQDIGVS KDLNEQFKKH LTNSEPLDLD FSIQVLSSGS WPFQQSCTFA LPSELE RSY QRFTAFYASR HSGRKLTWLY QLSKGELVTN CFKNRYTLQA STFQMAILLQ YNTEDAYTVQ QLTDSTQIKM DILAQVL QI LLKSKLLVLE DENANVDEVE LKPDTLIKLY LGYKNKKLRV NINVPMKTEQ KQEQETTHKN IEEDRKLLIQ AAIVRIMK M RKVLKHQQLL GEVLTQLSSR FKPRVPVIKK CIDILIEKEY LERVDGEKDT YSYLA UniProtKB: Cullin-1 |
-分子 #2: Cullin-associated NEDD8-dissociated protein 1
| 分子 | 名称: Cullin-associated NEDD8-dissociated protein 1 / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 137.386281 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: GSPEFPGRMA SASYHISNLL EKMTSSDKDF RFMATNDLMT ELQKDSIKLD DDSERKVVKM ILKLLEDKNG EVQNLAVKCL GPLVSKVKE YQVETIVDTL CTNMLSDKEQ LRDISSIGLK TVIGELPPAS SGSALAANVC KKITGRLTSA IAKQEDVSVQ L EALDIMAD ...文字列: GSPEFPGRMA SASYHISNLL EKMTSSDKDF RFMATNDLMT ELQKDSIKLD DDSERKVVKM ILKLLEDKNG EVQNLAVKCL GPLVSKVKE YQVETIVDTL CTNMLSDKEQ LRDISSIGLK TVIGELPPAS SGSALAANVC KKITGRLTSA IAKQEDVSVQ L EALDIMAD MLSRQGGLLV NFHPSILTCL LPQLTSPRLA VRKRTIIALG HLVMSCGNIV FVDLIEHLLS ELSKNDSMST TR TYIQCIA AISRQAGHRI GEYLEKIIPL VVKFCNVDDD ELREYCIQAF ESFVRRCPKE VYPHVSTIIN ICLKYLTYDP NYN YDDEDE DENAMDADGG DDDDQGSDDE YSDDDDMSWK VRRAAAKCLD AVVSTRHEML PEFYKTVSPA LISRFKEREE NVKA DVFHA YLSLLKQTRP VQSWLCDPDA MEQGETPLTM LQSQVPNIVK ALHKQMKEKS VKTRQCCFNM LTELVNVLPG ALTQH IPVL VPGIIFSLND KSSSSNLKID ALSCLYVILC NHSPQVFHPH VQALVPPVVA CVGDPFYKIT SEALLVTQQL VKVIRP LDQ PSSFDATPYI KDLFTCTIKR LKAADIDQEV KERAISCMGQ IICNLGDNLG SDLPNTLQIF LERLKNEITR LTTVKAL TL IAGSPLKIDL RPVLGEGVPI LASFLRKNQR ALKLGTLSAL DILIKNYSDS LTAAMIDAVL DELPPLISES DMHVSQMA I SFLTTLAKVY PSSLSKISGS ILNELIGLVR SPLLQGGALS AMLDFFQALV VTGTNNLGYM DLLRMLTGPV YSQSTALTH KQSYYSIAKC VAALTRACPK EGPAVVGQFI QDVKNSRSTD SIRLLALLSL GEVGHHIDLS GQLELKSVIL EAFSSPSEEV KSAASYALG SISVGNLPEY LPFVLQEITS QPKRQYLLLH SLKEIISSAS VVGLKPYVEN IWALLLKHCE CAEEGTRNVV V ECLGKLTL IDPETLLPRL KGYLISGSSY ARSSVVTAVK FTISDHPQPI DPLLKNCIGD FLKTLEDPDL NVRRVALVTF NS AAHNKPS LIRDLLDTVL PHLYNETKVR KELIREVEMG PFKHTVDDGL DIRKAAFECM YTLLDSCLDR LDIFEFLNHV EDG LKDHYD IKMLTFLMLV RLSTLCPSAV LQRLDRLVEP LRATCTTKVK ANSVKQEFEK QDELKRSAMR AVAALLTIPE AEKS PLMSE FQSQISSNPE LAAIFESIQK DSSSTNLESM DTS UniProtKB: Cullin-associated NEDD8-dissociated protein 1 |
-分子 #3: E3 ubiquitin-protein ligase RBX1, N-terminally processed
| 分子 | 名称: E3 ubiquitin-protein ligase RBX1, N-terminally processed タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 12.089677 KDa |
| 組換発現 | 生物種:  Trichoplusia ni (イラクサキンウワバ) Trichoplusia ni (イラクサキンウワバ) |
| 配列 | 文字列: GSMDVDTPSG TNSGAGKKRF EVKKWNAVAL WAWDIVVDNC AICRNHIMDL CIECQANQAS ATSEECTVAW GVCNHAFHFH CISRWLKTR QVCPLDNREW EFQKYGH UniProtKB: E3 ubiquitin-protein ligase RBX1 |
-分子 #4: ZINC ION
| 分子 | 名称: ZINC ION / タイプ: ligand / ID: 4 / コピー数: 3 / 式: ZN |
|---|---|
| 分子量 | 理論値: 65.409 Da |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 2 mg/mL |
|---|---|
| 緩衝液 | pH: 7.5 |
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 平均電子線量: 60.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.0 µm / 最小 デフォーカス(公称値): 0.5 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー
























































 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)