+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of DNA-bound human RAD17-RFC clamp loader and 9-1-1 checkpoint clamp | |||||||||
 マップデータ マップデータ | Map | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | DNA damage checkpoint / Rad17-RFC / 9-1-1 / DNA BINDING PROTEIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報meiotic DNA integrity checkpoint signaling / checkpoint clamp complex / meiotic recombination checkpoint signaling / Rad17 RFC-like complex / Elg1 RFC-like complex / DNA replication factor C complex / Ctf18 RFC-like complex / double-stranded DNA 3'-5' DNA exonuclease activity / exodeoxyribonuclease III / DNA clamp loader activity ...meiotic DNA integrity checkpoint signaling / checkpoint clamp complex / meiotic recombination checkpoint signaling / Rad17 RFC-like complex / Elg1 RFC-like complex / DNA replication factor C complex / Ctf18 RFC-like complex / double-stranded DNA 3'-5' DNA exonuclease activity / exodeoxyribonuclease III / DNA clamp loader activity / positive regulation of DNA-directed DNA polymerase activity / DNA replication checkpoint signaling / Polymerase switching / regulation of phosphorylation / embryo development ending in birth or egg hatching / chromatin-protein adaptor activity / protein localization to site of double-strand break / positive regulation of intrinsic apoptotic signaling pathway in response to DNA damage / mitotic DNA replication checkpoint signaling / Polymerase switching on the C-strand of the telomere / mitotic intra-S DNA damage checkpoint signaling / HDR through Single Strand Annealing (SSA) / DNA strand elongation involved in DNA replication / DNA synthesis involved in DNA repair / Impaired BRCA2 binding to RAD51 / negative regulation of DNA replication / Presynaptic phase of homologous DNA pairing and strand exchange / PCNA-Dependent Long Patch Base Excision Repair / ATP-dependent activity, acting on DNA / Activation of ATR in response to replication stress / response to UV / 3'-5' exonuclease activity / substantia nigra development / telomere maintenance / DNA damage checkpoint signaling / Translesion synthesis by REV1 / Translesion synthesis by POLK / Translesion synthesis by POLI / Gap-filling DNA repair synthesis and ligation in GG-NER / cellular response to ionizing radiation / nucleotide-excision repair / Termination of translesion DNA synthesis / Recognition of DNA damage by PCNA-containing replication complex / Translesion Synthesis by POLH / double-strand break repair via homologous recombination / G2/M DNA damage checkpoint / HDR through Homologous Recombination (HRR) / SH3 domain binding / Dual Incision in GG-NER / DNA-templated DNA replication / histone deacetylase binding / Dual incision in TC-NER / intrinsic apoptotic signaling pathway in response to DNA damage / Gap-filling DNA repair synthesis and ligation in TC-NER / site of double-strand break / chromosome / Processing of DNA double-strand break ends / Regulation of TP53 Activity through Phosphorylation / damaged DNA binding / DNA replication / DNA repair / intracellular membrane-bounded organelle / DNA damage response / chromatin binding / protein kinase binding / nucleolus / enzyme binding / ATP hydrolysis activity / DNA binding / nucleoplasm / ATP binding / nucleus / cytoplasm / cytosol 類似検索 - 分子機能 | |||||||||
| 生物種 |  Homo sapiens (ヒト) / synthetic construct (人工物) Homo sapiens (ヒト) / synthetic construct (人工物) | |||||||||
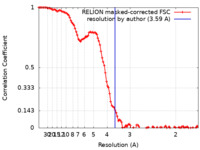
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.59 Å | |||||||||
 データ登録者 データ登録者 | Day M / Oliver AW / Pearl LH | |||||||||
| 資金援助 |  英国, 2件 英国, 2件
| |||||||||
 引用 引用 |  ジャーナル: Nucleic Acids Res / 年: 2022 ジャーナル: Nucleic Acids Res / 年: 2022タイトル: Structure of the human RAD17-RFC clamp loader and 9-1-1 checkpoint clamp bound to a dsDNA-ssDNA junction. 著者: Matthew Day / Antony W Oliver / Laurence H Pearl /  要旨: The RAD9-RAD1-HUS1 (9-1-1) clamp forms one half of the DNA damage checkpoint system that signals the presence of substantial regions of single-stranded DNA arising from replication fork collapse or ...The RAD9-RAD1-HUS1 (9-1-1) clamp forms one half of the DNA damage checkpoint system that signals the presence of substantial regions of single-stranded DNA arising from replication fork collapse or resection of DNA double strand breaks. Loaded at the 5'-recessed end of a dsDNA-ssDNA junction by the RAD17-RFC clamp loader complex, the phosphorylated C-terminal tail of the RAD9 subunit of 9-1-1 engages with the mediator scaffold TOPBP1 which in turn activates the ATR kinase, localised through the interaction of its constitutive partner ATRIP with RPA-coated ssDNA. Using cryogenic electron microscopy (cryoEM) we have determined the structure of a complex of the human RAD17-RFC clamp loader bound to human 9-1-1, engaged with a dsDNA-ssDNA junction. The structure answers the key questions of how RAD17 confers specificity for 9-1-1 over PCNA, and how the clamp loader specifically recognises the recessed 5' DNA end and fixes the orientation of 9-1-1 on the ssDNA. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_14527.map.gz emd_14527.map.gz | 87.6 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-14527-v30.xml emd-14527-v30.xml emd-14527.xml emd-14527.xml | 28.6 KB 28.6 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_14527_fsc.xml emd_14527_fsc.xml | 10.6 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_14527.png emd_14527.png | 113.7 KB | ||
| Filedesc metadata |  emd-14527.cif.gz emd-14527.cif.gz | 9 KB | ||
| その他 |  emd_14527_half_map_1.map.gz emd_14527_half_map_1.map.gz emd_14527_half_map_2.map.gz emd_14527_half_map_2.map.gz | 77.4 MB 77.4 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-14527 http://ftp.pdbj.org/pub/emdb/structures/EMD-14527 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-14527 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-14527 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_14527_validation.pdf.gz emd_14527_validation.pdf.gz | 912.3 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_14527_full_validation.pdf.gz emd_14527_full_validation.pdf.gz | 911.9 KB | 表示 | |
| XML形式データ |  emd_14527_validation.xml.gz emd_14527_validation.xml.gz | 17.7 KB | 表示 | |
| CIF形式データ |  emd_14527_validation.cif.gz emd_14527_validation.cif.gz | 23.3 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-14527 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-14527 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-14527 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-14527 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  7z6hMC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
| 電子顕微鏡画像生データ |  EMPIAR-11062 (タイトル: Structure of the human RAD17-RFC clamp loader and 9-1-1 checkpoint clamp bound to a dsDNA-ssDNA junction EMPIAR-11062 (タイトル: Structure of the human RAD17-RFC clamp loader and 9-1-1 checkpoint clamp bound to a dsDNA-ssDNA junctionData size: 4.4 TB Data #1: unaligned multiframe EER movies for human RAD17-RFC clamp loader and 9-1-1 checkpoint clamp bound to a dsDNA-ssDNA junction [micrographs - multiframe]) |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_14527.map.gz / 形式: CCP4 / 大きさ: 98.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_14527.map.gz / 形式: CCP4 / 大きさ: 98.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Map | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.86 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: Half-map 1
| ファイル | emd_14527_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half-map 1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half-map 2
| ファイル | emd_14527_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half-map 2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
+全体 : DNA-bound human RAD17-RFC clamp loader and 9-1-1 checkpoint clamp
+超分子 #1: DNA-bound human RAD17-RFC clamp loader and 9-1-1 checkpoint clamp
+分子 #1: Cell cycle checkpoint control protein RAD9A
+分子 #2: Cell cycle checkpoint protein RAD1,Cell cycle checkpoint protein RAD17
+分子 #3: Checkpoint protein HUS1
+分子 #4: Replication factor C subunit 4
+分子 #5: Replication factor C subunit 3
+分子 #6: Replication factor C subunit 2
+分子 #7: Replication factor C subunit 5
+分子 #8: Hairpin DNA
+分子 #9: PHOSPHOTHIOPHOSPHORIC ACID-ADENYLATE ESTER
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.21 mg/mL |
|---|---|
| 緩衝液 | pH: 7.5 |
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / 前処理 - タイプ: PLASMA CLEANING |
| 凍結 | 凍結剤: ETHANE-PROPANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: FEI FALCON IV (4k x 4k) 平均電子線量: 34.9 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: OTHER / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 1.5 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー












 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)