+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | pMMO structure from native membranes by cryoET and STA | |||||||||
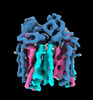
 マップデータ マップデータ | native pMMO density map by cryoET and STA | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | pMMO / array / native membranes / MEMBRANE PROTEIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報methane monooxygenase (particulate) / monooxygenase activity / metal ion binding / membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |  Methylococcus capsulatus str. Bath (バクテリア) Methylococcus capsulatus str. Bath (バクテリア) | |||||||||
| 手法 | サブトモグラム平均法 / クライオ電子顕微鏡法 / 解像度: 4.8 Å | |||||||||
 データ登録者 データ登録者 | Zhu Y / Ni T | |||||||||
| 資金援助 |  英国, 2件 英国, 2件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2022 ジャーナル: Nat Commun / 年: 2022タイトル: Structure and activity of particulate methane monooxygenase arrays in methanotrophs. 著者: Yanan Zhu / Christopher W Koo / C Keith Cassidy / Matthew C Spink / Tao Ni / Laura C Zanetti-Domingues / Benji Bateman / Marisa L Martin-Fernandez / Juan Shen / Yuewen Sheng / Yun Song / ...著者: Yanan Zhu / Christopher W Koo / C Keith Cassidy / Matthew C Spink / Tao Ni / Laura C Zanetti-Domingues / Benji Bateman / Marisa L Martin-Fernandez / Juan Shen / Yuewen Sheng / Yun Song / Zhengyi Yang / Amy C Rosenzweig / Peijun Zhang /    要旨: Methane-oxidizing bacteria play a central role in greenhouse gas mitigation and have potential applications in biomanufacturing. Their primary metabolic enzyme, particulate methane monooxygenase ...Methane-oxidizing bacteria play a central role in greenhouse gas mitigation and have potential applications in biomanufacturing. Their primary metabolic enzyme, particulate methane monooxygenase (pMMO), is housed in copper-induced intracytoplasmic membranes (ICMs), of which the function and biogenesis are not known. We show by serial cryo-focused ion beam (cryoFIB) milling/scanning electron microscope (SEM) volume imaging and lamellae-based cellular cryo-electron tomography (cryoET) that these ICMs are derived from the inner cell membrane. The pMMO trimer, resolved by cryoET and subtomogram averaging to 4.8 Å in the ICM, forms higher-order hexagonal arrays in intact cells. Array formation correlates with increased enzymatic activity, highlighting the importance of studying the enzyme in its native environment. These findings also demonstrate the power of cryoET to structurally characterize native membrane enzymes in the cellular context. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_14399.map.gz emd_14399.map.gz | 20.7 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-14399-v30.xml emd-14399-v30.xml emd-14399.xml emd-14399.xml | 12.4 KB 12.4 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_14399.png emd_14399.png | 96.8 KB | ||
| Filedesc metadata |  emd-14399.cif.gz emd-14399.cif.gz | 5.5 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-14399 http://ftp.pdbj.org/pub/emdb/structures/EMD-14399 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-14399 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-14399 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_14399_validation.pdf.gz emd_14399_validation.pdf.gz | 486.2 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_14399_full_validation.pdf.gz emd_14399_full_validation.pdf.gz | 485.8 KB | 表示 | |
| XML形式データ |  emd_14399_validation.xml.gz emd_14399_validation.xml.gz | 5.7 KB | 表示 | |
| CIF形式データ |  emd_14399_validation.cif.gz emd_14399_validation.cif.gz | 6.4 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-14399 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-14399 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-14399 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-14399 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  7yzyMC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_14399.map.gz / 形式: CCP4 / 大きさ: 22.2 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_14399.map.gz / 形式: CCP4 / 大きさ: 22.2 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | native pMMO density map by cryoET and STA | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.34 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : pMMO
| 全体 | 名称: pMMO |
|---|---|
| 要素 |
|
-超分子 #1: pMMO
| 超分子 | 名称: pMMO / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Methylococcus capsulatus str. Bath (バクテリア) Methylococcus capsulatus str. Bath (バクテリア) |
-分子 #1: Particulate methane monooxygenase beta subunit
| 分子 | 名称: Particulate methane monooxygenase beta subunit / タイプ: protein_or_peptide / ID: 1 / コピー数: 3 / 光学異性体: LEVO / EC番号: methane monooxygenase (particulate) |
|---|---|
| 由来(天然) | 生物種:  Methylococcus capsulatus str. Bath (バクテリア) Methylococcus capsulatus str. Bath (バクテリア)株: ATCC 33009 / NCIMB 11132 / Bath |
| 分子量 | 理論値: 28.445098 KDa |
| 配列 | 文字列: MSAAQSAVRS HAEAVQVSRT IDWMALFVVF FVIVGSYHIH AMLTMGDWDF WSDWKDRRLW VTVTPIVLVT FPAAVQSYLW ERYRLPWGA TVCVLGLLLG EWINRYFNFW GWTYFPINFV FPASLVPGAI ILDTVLMLSG SYLFTAIVGA MGWGLIFYPG N WPIIAPLH ...文字列: MSAAQSAVRS HAEAVQVSRT IDWMALFVVF FVIVGSYHIH AMLTMGDWDF WSDWKDRRLW VTVTPIVLVT FPAAVQSYLW ERYRLPWGA TVCVLGLLLG EWINRYFNFW GWTYFPINFV FPASLVPGAI ILDTVLMLSG SYLFTAIVGA MGWGLIFYPG N WPIIAPLH VPVEYNGMLM SIADIQGYNY VRTGTPEYIR MVEKGTLRTF GKDVAPVSAF FSAFMSILIY FMWHFIGRWF SN ERFLQST UniProtKB: Particulate methane monooxygenase beta subunit |
-分子 #2: Methane monooxygenase subunit C2
| 分子 | 名称: Methane monooxygenase subunit C2 / タイプ: protein_or_peptide / ID: 2 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Methylococcus capsulatus str. Bath (バクテリア) Methylococcus capsulatus str. Bath (バクテリア)株: ATCC 33009 / NCIMB 11132 / Bath |
| 分子量 | 理論値: 33.21418 KDa |
| 配列 | 文字列: MHETKQGGEK RFTGAICRCS HRYNSMEVKM AATTIGGAAA AEAPLLDKKW LTFALAIYTV FYLWVRWYEG VYGWSAGLDS FAPEFETYW MNFLYTEIVL EIVTASILWG YLWKTRDRNL AALTPREELR RNFTHLVWLV AYAWAIYWGA SYFTEQDGTW H QTIVRDTD ...文字列: MHETKQGGEK RFTGAICRCS HRYNSMEVKM AATTIGGAAA AEAPLLDKKW LTFALAIYTV FYLWVRWYEG VYGWSAGLDS FAPEFETYW MNFLYTEIVL EIVTASILWG YLWKTRDRNL AALTPREELR RNFTHLVWLV AYAWAIYWGA SYFTEQDGTW H QTIVRDTD FTPSHIIEFY LSYPIYIITG FAAFIYAKTR LPFFAKGISL PYLVLVVGPF MILPNVGLNE WGHTFWFMEE LF VAPLHYG FVIFGWLALA VMGTLTQTFY SFAQGGLGQS LCEAVDEGLI AK UniProtKB: Methane monooxygenase subunit C2 |
-分子 #3: Particulate methane monooxygenase alpha subunit
| 分子 | 名称: Particulate methane monooxygenase alpha subunit / タイプ: protein_or_peptide / ID: 3 / コピー数: 3 / 光学異性体: LEVO / EC番号: methane monooxygenase (particulate) |
|---|---|
| 由来(天然) | 生物種:  Methylococcus capsulatus str. Bath (バクテリア) Methylococcus capsulatus str. Bath (バクテリア)株: ATCC 33009 / NCIMB 11132 / Bath |
| 分子量 | 理論値: 46.129746 KDa |
| 配列 | 文字列: MKTIKDRIAK WSAIGLLSAV AATAFYAPSA SAHGEKSQAA FMRMRTIHWY DLSWSKEKVK INETVEIKGK FHVFEGWPET VDEPDVAFL NVGMPGPVFI RKESYIGGQL VPRSVRLEIG KTYDFRVVLK ARRPGDWHVH TMMNVQGGGP IIGPGKWITV E GSMSEFRN ...文字列: MKTIKDRIAK WSAIGLLSAV AATAFYAPSA SAHGEKSQAA FMRMRTIHWY DLSWSKEKVK INETVEIKGK FHVFEGWPET VDEPDVAFL NVGMPGPVFI RKESYIGGQL VPRSVRLEIG KTYDFRVVLK ARRPGDWHVH TMMNVQGGGP IIGPGKWITV E GSMSEFRN PVTTLTGQTV DLENYNEGNT YFWHAFWFAI GVAWIGYWSR RPIFIPRLLM VDAGRADELV SATDRKVAMG FL AATILIV VMAMSSANSK YPITIPLQAG TMRGMKPLEL PAPTVSVKVE DATYRVPGRA MRMKLTITNH GNSPIRLGEF YTA SVRFLD SDVYKDTTGY PEDLLAEDGL SVSDNSPLAP GETRTVDVTA SDAAWEVYRL SDIIYDPDSR FAGLLFFFDA TGNR QVVQI DAPLIPSFM UniProtKB: Particulate methane monooxygenase alpha subunit |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | サブトモグラム平均法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 6.8 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 撮影したグリッド数: 2 / 平均電子線量: 3.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 6.0 µm / 最小 デフォーカス(公称値): 2.5 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 最終 再構成 | 想定した対称性 - 点群: C3 (3回回転対称) / 解像度のタイプ: BY AUTHOR / 解像度: 4.8 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 使用したサブトモグラム数: 127417 |
|---|---|
| 抽出 | トモグラム数: 187 / 使用した粒子像数: 127417 |
| 最終 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
 ムービー
ムービー コントローラー
コントローラー




 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)




















