+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of the Cyanobium sp. PCC 7001 RuBisCO enzyme at 3.8 A resolution with C1 symmetry | |||||||||
 マップデータ マップデータ | Cryo-EM structure of the Cyanobium sp. PCC 7001 RuBisCO enzyme at 3.8 A resolution with C1 symmetry | |||||||||
 試料 試料 |
| |||||||||
| 生物種 |  Cyanobium sp. PCC 7001 (バクテリア) Cyanobium sp. PCC 7001 (バクテリア) | |||||||||
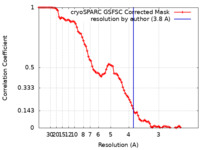
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.8 Å | |||||||||
 データ登録者 データ登録者 | Evans SL / Mann D / Bergeron JRC | |||||||||
| 資金援助 |  英国, 1件 英国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Structure / 年: 2023 ジャーナル: Structure / 年: 2023タイトル: Single-particle cryo-EM analysis of the shell architecture and internal organization of an intact α-carboxysome. 著者: Sasha L Evans / Monsour M J Al-Hazeem / Daniel Mann / Nicolas Smetacek / Andrew J Beavil / Yaqi Sun / Taiyu Chen / Gregory F Dykes / Lu-Ning Liu / Julien R C Bergeron /    要旨: Carboxysomes are proteinaceous bacterial microcompartments that sequester the key enzymes for carbon fixation in cyanobacteria and some proteobacteria. They consist of a virus-like icosahedral shell, ...Carboxysomes are proteinaceous bacterial microcompartments that sequester the key enzymes for carbon fixation in cyanobacteria and some proteobacteria. They consist of a virus-like icosahedral shell, encapsulating several enzymes, including ribulose 1,5-bisphosphate carboxylase/oxygenase (RuBisCO), responsible for the first step of the Calvin-Benson-Bassham cycle. Despite their significance in carbon fixation and great bioengineering potentials, the structural understanding of native carboxysomes is currently limited to low-resolution studies. Here, we report the characterization of a native α-carboxysome from a marine cyanobacterium by single-particle cryoelectron microscopy (cryo-EM). We have determined the structure of its RuBisCO enzyme, and obtained low-resolution maps of its icosahedral shell, and of its concentric interior organization. Using integrative modeling approaches, we have proposed a complete atomic model of an intact carboxysome, providing insight into its organization and assembly. This is critical for a better understanding of the carbon fixation mechanism and toward repurposing carboxysomes in synthetic biology for biotechnological applications. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_14376.map.gz emd_14376.map.gz | 59.6 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-14376-v30.xml emd-14376-v30.xml emd-14376.xml emd-14376.xml | 9.4 KB 9.4 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_14376_fsc.xml emd_14376_fsc.xml | 8.5 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_14376.png emd_14376.png | 59.1 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-14376 http://ftp.pdbj.org/pub/emdb/structures/EMD-14376 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-14376 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-14376 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_14376_validation.pdf.gz emd_14376_validation.pdf.gz | 547.3 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_14376_full_validation.pdf.gz emd_14376_full_validation.pdf.gz | 546.8 KB | 表示 | |
| XML形式データ |  emd_14376_validation.xml.gz emd_14376_validation.xml.gz | 11 KB | 表示 | |
| CIF形式データ |  emd_14376_validation.cif.gz emd_14376_validation.cif.gz | 14.3 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-14376 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-14376 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-14376 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-14376 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_14376.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_14376.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Cryo-EM structure of the Cyanobium sp. PCC 7001 RuBisCO enzyme at 3.8 A resolution with C1 symmetry | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.11 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : RuBisCO
| 全体 | 名称: RuBisCO |
|---|---|
| 要素 |
|
-超分子 #1: RuBisCO
| 超分子 | 名称: RuBisCO / タイプ: complex / ID: 1 / キメラ: Yes / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Cyanobium sp. PCC 7001 (バクテリア) Cyanobium sp. PCC 7001 (バクテリア) |
-分子 #1: CbbL
| 分子 | 名称: CbbL / タイプ: protein_or_peptide / ID: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Cyanobium sp. PCC 7001 (バクテリア) Cyanobium sp. PCC 7001 (バクテリア) |
| 配列 | 文字列: MSKKYDAGVK EYRDTYWTPD YVPLDTDLLA CFKCTGQEGV PKEEVAAAVA AESSTGTWST VWSELLVDLD FYKGRCYRIE DVPGDKEAFY AFIAYPLDLF EEGSVTNVLT SLVGNVFGFK ALRHLRLEDI RFPMAFIKTC PGPPNGICVE RDRMNKYGRP LLGCTIKPKL ...文字列: MSKKYDAGVK EYRDTYWTPD YVPLDTDLLA CFKCTGQEGV PKEEVAAAVA AESSTGTWST VWSELLVDLD FYKGRCYRIE DVPGDKEAFY AFIAYPLDLF EEGSVTNVLT SLVGNVFGFK ALRHLRLEDI RFPMAFIKTC PGPPNGICVE RDRMNKYGRP LLGCTIKPKL GLSGKNYGRV VYECLRGGLD FTKDDENINS QPFQRWQNRF EFVAEAVALA QQETGEKKGH YLNCTAATPE EMYERAEFAK ELGQPIIMHD YITGGFTANT GLSKWCRKNG MLLHIHRAMH AVIDRHPKHG IHFRVLAKCL RLSGGDQLHT GTVVGKLEGD RQTTLGFIDQ LRESFIPEDR SRGNFFDQDW GSMPGVFAVA SGGIHVWHMP ALVAIFGDDS VLQFGGGTHG HPWGSAAGAA ANRVALEACV KARNAGREIE KESRDILMEA AKHSPELAIA LETWKEIKFE FDTVDKLDVQ |
-分子 #2: CbbS
| 分子 | 名称: CbbS / タイプ: protein_or_peptide / ID: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Cyanobium sp. PCC 7001 (バクテリア) Cyanobium sp. PCC 7001 (バクテリア) |
| 配列 | 文字列: MPFKSTVGDY QTVATLETFG FLPPMTQDEI YDQIAYIIAQ GWSPLIEHVH PSRSMATYWS YWKLPFFGEK DLGVIVSELE ACHRAYPDHH VRLVGYDAYT QSQGACFVVF EGR |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 8 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 平均電子線量: 30.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 1.5 µm / 最小 デフォーカス(公称値): 0.5 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー












 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)