+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | ||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | CryoEM structure of mammalian AAP in complex with Meropenem | ||||||||||||||||||
 マップデータ マップデータ | |||||||||||||||||||
 試料 試料 |
| ||||||||||||||||||
 キーワード キーワード | inhibitor / complex / acylaminoacyl-peptidase / meropenem / carbapenem / acylpeptide hydrolase / HYDROLASE | ||||||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報acylaminoacyl-peptidase / omega peptidase activity / serine-type endopeptidase activity / proteolysis / cytoplasm 類似検索 - 分子機能 | ||||||||||||||||||
| 生物種 |  | ||||||||||||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.1 Å | ||||||||||||||||||
 データ登録者 データ登録者 | Kiss-Szeman AJ / Harmat V | ||||||||||||||||||
| 資金援助 |  ハンガリー, 5件 ハンガリー, 5件
| ||||||||||||||||||
 引用 引用 |  ジャーナル: Chem Sci / 年: 2022 ジャーナル: Chem Sci / 年: 2022タイトル: A carbapenem antibiotic inhibiting a mammalian serine protease: structure of the acylaminoacyl peptidase-meropenem complex. 著者: Anna J Kiss-Szemán / Luca Takács / Zoltán Orgován / Pál Stráner / Imre Jákli / Gitta Schlosser / Simonas Masiulis / Veronika Harmat / Dóra K Menyhárd / András Perczel /   要旨: The structure of porcine AAP (pAAP) in a covalently bound complex with meropenem was determined by cryo-EM to 2.1 Å resolution, showing the mammalian serine-protease inhibited by a carbapenem ...The structure of porcine AAP (pAAP) in a covalently bound complex with meropenem was determined by cryo-EM to 2.1 Å resolution, showing the mammalian serine-protease inhibited by a carbapenem antibiotic. AAP is a modulator of the ubiquitin-proteasome degradation system and the site of a drug-drug interaction between the widely used antipsychotic, valproate and carbapenems. The active form of pAAP - a toroidal tetramer - binds four meropenem molecules covalently linked to the catalytic Ser587 of the serine-protease triad, in an acyl-enzyme state. AAP is hindered from fully processing the antibiotic by the displacement and protonation of His707 of the catalytic triad. We show that AAP is made susceptible to the association by its unusually sheltered active pockets and flexible catalytic triads, while the carbapenems possess sufficiently small substituents on their β-lactam rings to fit into the shallow substrate-specificity pocket of the enzyme. | ||||||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_14149.map.gz emd_14149.map.gz | 168.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-14149-v30.xml emd-14149-v30.xml emd-14149.xml emd-14149.xml | 16.1 KB 16.1 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_14149.png emd_14149.png | 214 KB | ||
| Filedesc metadata |  emd-14149.cif.gz emd-14149.cif.gz | 6.5 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-14149 http://ftp.pdbj.org/pub/emdb/structures/EMD-14149 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-14149 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-14149 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_14149_validation.pdf.gz emd_14149_validation.pdf.gz | 582 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_14149_full_validation.pdf.gz emd_14149_full_validation.pdf.gz | 581.6 KB | 表示 | |
| XML形式データ |  emd_14149_validation.xml.gz emd_14149_validation.xml.gz | 7.1 KB | 表示 | |
| CIF形式データ |  emd_14149_validation.cif.gz emd_14149_validation.cif.gz | 8.2 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-14149 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-14149 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-14149 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-14149 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  7qunMC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_14149.map.gz / 形式: CCP4 / 大きさ: 178 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_14149.map.gz / 形式: CCP4 / 大きさ: 178 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.96 Å | ||||||||||||||||||||||||||||||||||||
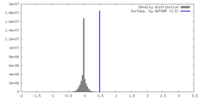
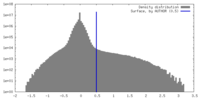
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Homotetramer of aclyaminoacyl-peptidase
| 全体 | 名称: Homotetramer of aclyaminoacyl-peptidase |
|---|---|
| 要素 |
|
-超分子 #1: Homotetramer of aclyaminoacyl-peptidase
| 超分子 | 名称: Homotetramer of aclyaminoacyl-peptidase / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  |
-分子 #1: Acylamino-acid-releasing enzyme
| 分子 | 名称: Acylamino-acid-releasing enzyme / タイプ: protein_or_peptide / ID: 1 / コピー数: 4 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 81.324391 KDa |
| 配列 | 文字列: MERQVLLSEP EEAAALYRGL SRQPALSAAC LGPEVTTQYG GRYRTVHTEW TQRDLERMEN IRFCRQYLVF HDGDSVVFAG PAGNSVETR GELLSRESPS GTMKAVLRKA GGTGTAEEKQ FLEVWEKNRK LKSFNLSALE KHGPVYEDDC FGCLSWSHSE T HLLYVADK ...文字列: MERQVLLSEP EEAAALYRGL SRQPALSAAC LGPEVTTQYG GRYRTVHTEW TQRDLERMEN IRFCRQYLVF HDGDSVVFAG PAGNSVETR GELLSRESPS GTMKAVLRKA GGTGTAEEKQ FLEVWEKNRK LKSFNLSALE KHGPVYEDDC FGCLSWSHSE T HLLYVADK KRPKAESFFQ TKALDVTGSD DEMARTKKPD QAIKGDQFLF YEDWGENMVS KSTPVLCVLD IESGNISVLE GV PESVSPG QAFWAPGDTG VVFVGWWHEP FRLGIRFCTN RRSALYYVDL TGGKCELLSD ESVAVTSPRL SPDQCRIVYL RFP SLVPHQ QCGQLCLYDW YTRVTSVVVD IVPRQLGEDF SGIYCSLLPL GCWSADSQRV VFDSPQRSRQ DLFAVDTQMG SVTS LTAGG SGGSWKLLTI DRDLMVVQFS TPSVPPSLKV GFLPPAGKEQ AVSWVSLEEA EPFPDISWSI RVLQPPPQQE HVQYA GLDF EAILLQPSNS PEKTQVPMVV MPHGGPHSSF VTAWMLFPAM LCKMGFAVLL VNYRGSTGFG QDSILSLPGN VGHQDV KDV QFAVEQVLQE EHFDAGRVAL MGGSHGGFLS CHLIGQYPET YSACVVRNPV INIASMMGST DIPDWCMVEA GFSYSSD CL PDLSVWAAML DKSPIKYAPQ VKTPLLLMLG QEDRRVPFKQ GMEYYRVLKA RNVPVRLLLY PKSTHALSEV EVESDSFM N AVLWLCTHLG S UniProtKB: Acylamino-acid-releasing enzyme |
-分子 #2: (2S,3R,4S)-4-{[(3S,5S)-5-(dimethylcarbamoyl)pyrrolidin-3-yl]sulfa...
| 分子 | 名称: (2S,3R,4S)-4-{[(3S,5S)-5-(dimethylcarbamoyl)pyrrolidin-3-yl]sulfanyl}-2-[(2S,3R)-3-hydroxy-1-oxobutan-2-yl]-3-methyl-3,4-dihydro-2H-pyrrole-5-carboxylic acid タイプ: ligand / ID: 2 / コピー数: 4 / 式: DWZ |
|---|---|
| 分子量 | 理論値: 385.478 Da |
| Chemical component information | 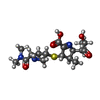 ChemComp-DWZ: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 1 mg/mL |
|---|---|
| 緩衝液 | pH: 7.5 / 構成要素 - 濃度: 10.0 mM / 構成要素 - 式: C4H11NO3 / 構成要素 - 名称: TRIS |
| グリッド | モデル: Quantifoil R2/1 / 材質: COPPER / メッシュ: 200 / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 25 sec. |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 95 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV |
| 詳細 | Covalent complex with carbapenem type antibiotic Meropenem |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: FEI FALCON IV (4k x 4k) 撮影したグリッド数: 4 / 実像数: 11625 / 平均露光時間: 5.93 sec. / 平均電子線量: 41.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 3.0 µm / 最小 デフォーカス(公称値): 1.0 µm / 倍率(公称値): 130000 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー









 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)