+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-1361 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of TOR and its complex with KOG1. | |||||||||
 マップデータ マップデータ | 3D reconstruction of the yeast TOR (target of rapamycin)protein complexed with KOG1 | |||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 | molecular_function / Regulatory associated protein of TOR / HEAT repeat / 1-phosphatidylinositol-3-kinase activity 機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |  | |||||||||
| 手法 | 単粒子再構成法 / ネガティブ染色法 / 解像度: 26.0 Å | |||||||||
 データ登録者 データ登録者 | Adami A / Garcia-Alvarez B / Arias-Palomo E / Barford D / Llorca O | |||||||||
 引用 引用 |  ジャーナル: Mol Cell / 年: 2007 ジャーナル: Mol Cell / 年: 2007タイトル: Structure of TOR and its complex with KOG1. 著者: Alessandra Adami / Begoña García-Alvarez / Ernesto Arias-Palomo / David Barford / Oscar Llorca /  要旨: The target of rapamycin (TOR) is a large (281 kDa) conserved Ser/Thr protein kinase that functions as a central controller of cell growth. TOR assembles into two distinct multiprotein complexes: ...The target of rapamycin (TOR) is a large (281 kDa) conserved Ser/Thr protein kinase that functions as a central controller of cell growth. TOR assembles into two distinct multiprotein complexes: TORC1 and TORC2. A defining feature of TORC1 is the interaction of TOR with KOG1 (Raptor in mammals) and its sensitivity to a rapamycin-FKBP12 complex. Here, we have reconstructed in three dimensions the 25 A resolution structures of endogenous budding yeast TOR1 and a TOR-KOG1 complex, using electron microscopy. TOR features distinctive N-terminal HEAT repeats that form a curved tubular-shaped domain that associates with the C-terminal WD40 repeat domain of KOG1. The N terminus of KOG1 is in proximity to the TOR kinase domain, likely functioning to bring substrates into the vicinity of the catalytic region. A model is proposed for the molecular architecture of the TOR-KOG1 complex explaining its sensitivity to rapamycin. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_1361.map.gz emd_1361.map.gz | 195.6 KB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-1361-v30.xml emd-1361-v30.xml emd-1361.xml emd-1361.xml | 10.4 KB 10.4 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  1361.gif 1361.gif | 54.9 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-1361 http://ftp.pdbj.org/pub/emdb/structures/EMD-1361 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-1361 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-1361 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_1361_validation.pdf.gz emd_1361_validation.pdf.gz | 196.1 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_1361_full_validation.pdf.gz emd_1361_full_validation.pdf.gz | 195.2 KB | 表示 | |
| XML形式データ |  emd_1361_validation.xml.gz emd_1361_validation.xml.gz | 5.2 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1361 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1361 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1361 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1361 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_1361.map.gz / 形式: CCP4 / 大きさ: 1.4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_1361.map.gz / 形式: CCP4 / 大きさ: 1.4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | 3D reconstruction of the yeast TOR (target of rapamycin)protein complexed with KOG1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 4.2 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : TOR-KOG1 complex
| 全体 | 名称: TOR-KOG1 complex |
|---|---|
| 要素 |
|
-超分子 #1000: TOR-KOG1 complex
| 超分子 | 名称: TOR-KOG1 complex / タイプ: sample / ID: 1000 集合状態: One monomer of TOR bound to one monomer of KOG1 Number unique components: 2 |
|---|---|
| 分子量 | 理論値: 457 KDa |
-分子 #1: TOR
| 分子 | 名称: TOR / タイプ: protein_or_peptide / ID: 1 / Name.synonym: target of rapamycin / コピー数: 1 / 集合状態: monomer / 組換発現: No |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 実験値: 281 KDa |
| 配列 | GO: 1-phosphatidylinositol-3-kinase activity / InterPro: HEAT repeat |
-分子 #2: KOG1
| 分子 | 名称: KOG1 / タイプ: protein_or_peptide / ID: 2 / Name.synonym: Kontroller of Growth 1 / コピー数: 1 / 集合状態: Monomer / 組換発現: Yes |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 実験値: 176 KDa |
| 組換発現 | 生物種:  |
| 配列 | GO: molecular_function / InterPro: Regulatory associated protein of TOR |
-実験情報
-構造解析
| 手法 | ネガティブ染色法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.1 詳細: 50mM HEPES-KOH pH7.1, 3mM DTT, 10% glycerol, 0.25% Tween 20 for TOR-KOG1, 150 mM KCl, 1 mM Mg acetate, 2 mM EGTA |
|---|---|
| 染色 | タイプ: NEGATIVE / 詳細: 1% uranyl acetate |
| グリッド | 詳細: 400 mesh Copper/Palladium grid |
| 凍結 | 凍結剤: NONE / 装置: OTHER |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | JEOL 1230 |
|---|---|
| アライメント法 | Legacy - 非点収差: correction with FFT and CCD camera |
| 詳細 | Microscope used - JEOL JEM-1230 |
| 撮影 | カテゴリ: FILM / フィルム・検出器のモデル: KODAK SO-163 FILM / デジタル化 - スキャナー: OTHER / デジタル化 - サンプリング間隔: 10.5 µm 詳細: images scanned with a MINOLTA Dimage Scan Multi Pro scanner at 2400 dpi and averaged to a final 4.2 angstroms per pixel at the specimen ビット/ピクセル: 16 |
| 電子線 | 加速電圧: 100 kV / 電子線源: TUNGSTEN HAIRPIN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.9 mm / 倍率(公称値): 50000 |
| 試料ステージ | 試料ホルダー: Eucentric / 試料ホルダーモデル: OTHER |
- 画像解析
画像解析
| CTF補正 | 詳細: CTF for each micrograph was estimated using CTFIND3 and the phases flipped with BSOFT |
|---|---|
| 最終 再構成 | 想定した対称性 - 点群: C1 (非対称) / アルゴリズム: OTHER / 解像度のタイプ: BY AUTHOR / 解像度: 26.0 Å / 解像度の算出法: FSC 0.5 CUT-OFF / ソフトウェア - 名称: EMAN / 使用した粒子像数: 5508 |
-原子モデル構築 1
| 初期モデル | PDB ID: |
|---|---|
| ソフトウェア | 名称: ADP_EM |
| 詳細 | Protocol: Rigid Body |
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT / 当てはまり具合の基準: R-factor |
 ムービー
ムービー コントローラー
コントローラー



 UCSF Chimera
UCSF Chimera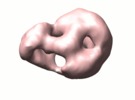









 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)






















