+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Helical reconstruction of TOROID (TORC1 Organized in Inhibited Domains) filaments. | |||||||||||||||
 マップデータ マップデータ | Helical reconstruction of TOROID (TORC1 Organized in Inhibited Domains) filaments. | |||||||||||||||
 試料 試料 |
| |||||||||||||||
| 生物種 |  | |||||||||||||||
| 手法 | らせん対称体再構成法 / クライオ電子顕微鏡法 / 解像度: 9.1 Å | |||||||||||||||
 データ登録者 データ登録者 | Felix J / Prouteau M / Bourgoint C / Bonnadei L / Desfosses A / Gabus C / Sadian Y / Savvides SN / Gutsche I / Loewith R | |||||||||||||||
| 資金援助 |  スイス, スイス,  フランス, 4件 フランス, 4件
| |||||||||||||||
 引用 引用 |  ジャーナル: Nat Struct Mol Biol / 年: 2023 ジャーナル: Nat Struct Mol Biol / 年: 2023タイトル: EGOC inhibits TOROID polymerization by structurally activating TORC1. 著者: Manoël Prouteau / Clélia Bourgoint / Jan Felix / Lenny Bonadei / Yashar Sadian / Caroline Gabus / Savvas N Savvides / Irina Gutsche / Ambroise Desfosses / Robbie Loewith /    要旨: Target of rapamycin complex 1 (TORC1) is a protein kinase controlling cell homeostasis and growth in response to nutrients and stresses. In Saccharomyces cerevisiae, glucose depletion triggers a ...Target of rapamycin complex 1 (TORC1) is a protein kinase controlling cell homeostasis and growth in response to nutrients and stresses. In Saccharomyces cerevisiae, glucose depletion triggers a redistribution of TORC1 from a dispersed localization over the vacuole surface into a large, inactive condensate called TOROID (TORC1 organized in inhibited domains). However, the mechanisms governing this transition have been unclear. Here, we show that acute depletion and repletion of EGO complex (EGOC) activity is sufficient to control TOROID distribution, independently of other nutrient-signaling pathways. The 3.9-Å-resolution structure of TORC1 from TOROID cryo-EM data together with interrogation of key interactions in vivo provide structural insights into TORC1-TORC1' and TORC1-EGOC interaction interfaces. These data support a model in which glucose-dependent activation of EGOC triggers binding to TORC1 at an interface required for TOROID assembly, preventing TORC1 polymerization and promoting release of active TORC1. | |||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_13595.map.gz emd_13595.map.gz | 22.5 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-13595-v30.xml emd-13595-v30.xml emd-13595.xml emd-13595.xml | 24.5 KB 24.5 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_13595_fsc.xml emd_13595_fsc.xml | 10.8 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_13595.png emd_13595.png | 3.8 MB | ||
| その他 |  emd_13595_half_map_1.map.gz emd_13595_half_map_1.map.gz emd_13595_half_map_2.map.gz emd_13595_half_map_2.map.gz | 79.2 MB 79.2 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-13595 http://ftp.pdbj.org/pub/emdb/structures/EMD-13595 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-13595 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-13595 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_13595_validation.pdf.gz emd_13595_validation.pdf.gz | 739.5 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_13595_full_validation.pdf.gz emd_13595_full_validation.pdf.gz | 739.1 KB | 表示 | |
| XML形式データ |  emd_13595_validation.xml.gz emd_13595_validation.xml.gz | 17.6 KB | 表示 | |
| CIF形式データ |  emd_13595_validation.cif.gz emd_13595_validation.cif.gz | 23.4 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-13595 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-13595 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-13595 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-13595 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_13595.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_13595.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Helical reconstruction of TOROID (TORC1 Organized in Inhibited Domains) filaments. | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 2.7 Å | ||||||||||||||||||||||||||||||||||||
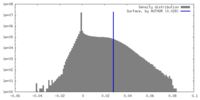
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: Helical reconstruction of TOROID (TORC1 Organized in Inhibited...
| ファイル | emd_13595_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Helical reconstruction of TOROID (TORC1 Organized in Inhibited Domains) filaments, half map 1. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
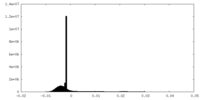
| 密度ヒストグラム |
-ハーフマップ: Helical reconstruction of TOROID (TORC1 Organized in Inhibited...
| ファイル | emd_13595_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Helical reconstruction of TOROID (TORC1 Organized in Inhibited Domains) filaments, half map 2. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
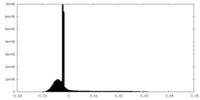
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : TORC1 Organized in Inhibited Domains (TOROID)
| 全体 | 名称: TORC1 Organized in Inhibited Domains (TOROID) |
|---|---|
| 要素 |
|
-超分子 #1: TORC1 Organized in Inhibited Domains (TOROID)
| 超分子 | 名称: TORC1 Organized in Inhibited Domains (TOROID) / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  |
-分子 #1: Target of rapamycin complex 1 subunit KOG1
| 分子 | 名称: Target of rapamycin complex 1 subunit KOG1 / タイプ: protein_or_peptide / ID: 1 詳細: S. cerevisiae TOROIDs (TORC1 Organized in Inhibited Domains) in the sample are composed of Kog1 (P38873), Lst8 (P41318) and Tor2 (P32600). 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 配列 | 文字列: MPEIYGPQPL KPLNTVMRHG FEEQYQSDQL LQSLANDFIF YFDDKRHKTN GNPIPEEDKQ RDVNRYYQP ITDWKIMKDR QKTVSAALLL CLNLGVDPPD VMKTHPCARV EAWVDPLNFQ D SKKAIEQI GKNLQAQYET LSLRTRYKQS LDPCVEDVKR FCNSLRRTSK ...文字列: MPEIYGPQPL KPLNTVMRHG FEEQYQSDQL LQSLANDFIF YFDDKRHKTN GNPIPEEDKQ RDVNRYYQP ITDWKIMKDR QKTVSAALLL CLNLGVDPPD VMKTHPCARV EAWVDPLNFQ D SKKAIEQI GKNLQAQYET LSLRTRYKQS LDPCVEDVKR FCNSLRRTSK EDRILFHYNG HG VPKPTKS GEIWVFNRGY TQYIPVSLYD LQTWLGAPCI FVYDCNSAEN ILINFQKFVQ KRI KDDEEG NHDVAAPSPT SAYQDCFQLA SCTSDELLLM SPELPADLFS CCLTCPIEIS IRIF LMQSP LKDSKYKIFF ENSTSNQPFG DSKNSFKSKI PNVNIPGMLS DRRTPLGELN WIFTA ITDT IAWTSLPRPL FKKLFRHDLM IAALFRNFLL AKRIMPWYNC HPVSDPELPD SITTHP MWK SWDLAMDEVL TKIVIDLKNA PPATALESQM ILQQQETLQN GGSSKSNAQD TKAGSIQ TQ SRFAVANLST MSLVNNPALQ SRKSISLQSS QQQLQQQQQQ QQQFTGFFEQ NLTAFELW L KYASNVRHPP EQLPIVLQVL LSQVHRIRAL VLLSRFLDLG PWAVYLSLSI GIFPYVLKL LQSPAPELKP ILVFIWARIM SIDYKNTQSE LIKEKGYMYF VTVLVPDWGV NGMSATNGSA MINSGNPLT MTASQNINGP SSRYYERQQG NRTSNLGHNN LPFYHSNDTT DEQKAMAVFV L ASFVRNFP LGQKNCFSLE LVNKLCFYID NSEIPLLRQW CVILLGLLFA DNPLNRFVCM NT GAVEILL KSLKDPVPEV RTASIFALKH FISGFQDAEV ILRLQQEFEE QYQQLHSQLQ HLQ NQSHLQ QQQSQQQQQH LEQQQMKIEK QIRHCQVMQN QLEVIDLRKL KRQEIGNLIS ILPL INDGS SLVRKELVVY FSHIVSRYSN FFIVVVFNDL LEEIKLLEKS DINTRNTSDK YSVSQ GSIF YTVWKSLLIL AEDPFLENKE LSKQVIDYIL LELSAHKELG GPFAVMEKFL LKRSSK AHQ TGKFGFNSSQ VQFVKSSLRS FSPNERVDNN AFKKEQQQHD PKISHPMRTS LAKLFQS LG FSESNSDSDT QSSNTSMKSH TSKKGPSGLY LLNGNNNIYP TAETPRFRKH TEPLQLPL N SSFLDYSREY FQEPQMKKQE ADEPGSVEYN ARLWRRNRNE TIIQETQGEK KLSIYGNWS KKLISLNNKS QPKLMKFAQF EDQLITADDR STITVFDWEK GKTLSKFSNG TPFGTKVTDL KLINEDDSA LLLTGSSDGV IKIYRDYQDV DTFKIVSAWR GLTDMLLTPR STGLLTEWLQ I RGSLLTTG DVKVIRVWDA HTETVEVDIP AKTSSLITSL TADQLAGNIF VAGFADGSLR VY DRRLDPR DSMIRRWRAG NDKQGVWINN VHLQRGGYRE LVSGATNGVV ELWDIRSEDP VES FVDQNV TSQYGSQQKP TTMTCMQVHE HAPIIATGTK QIKIWTTSGD LLNSFKNSHN NGVT STLAA TGIPKSLSYS STSDAFLSSM AFHPHRMMIA ATNSHDSIVN IYKCEDERID YF |
-分子 #2: Serine/threonine-protein kinase TOR2
| 分子 | 名称: Serine/threonine-protein kinase TOR2 / タイプ: protein_or_peptide / ID: 2 詳細: S. cerevisiae TOROIDs (TORC1 Organized in Inhibited Domains) in the sample are composed of Kog1 (P38873), Lst8 (P41318) and Tor2 (P32600). 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 配列 | 文字列: MNKYINKYTT PPNLLSLRQR AEGKHRTRKK LTHKSHSHDD EMSTTSNTDS NHNGPNDSGR VITGSAGHI GKISFVDSEL DTTFSTLNLI FDKLKSDVPQ ERASGANELS TTLTSLAREV S AEQFQRFS NSLNNKIFEL IHGFTSSEKI GGILAVDTLI SFYLSTEELP ...文字列: MNKYINKYTT PPNLLSLRQR AEGKHRTRKK LTHKSHSHDD EMSTTSNTDS NHNGPNDSGR VITGSAGHI GKISFVDSEL DTTFSTLNLI FDKLKSDVPQ ERASGANELS TTLTSLAREV S AEQFQRFS NSLNNKIFEL IHGFTSSEKI GGILAVDTLI SFYLSTEELP NQTSRLANYL RV LIPSSDI EVMRLAANTL GRLTVPGGTL TSDFVEFEVR TCIDWLTLTA DNNSSSSKLE YRR HAALLI IKALADNSPY LLYPYVNSIL DNIWVPLRDA KLIIRLDAAV ALGKCLTIIQ DRDP ALGKQ WFQRLFQGCT HGLSLNTNDS VHATLLVFRE LLSLKAPYLR DKYDDIYKST MKYKE YKFD VIRREVYAIL PLLAAFDPAI FTKKYLDRIM VHYLRYLKNI DMNAANNSDK PFILVS IGD IAFEVGSSIS PYMTLILDNI REGLRTKFKV RKQFEKDLFY CIGKLACALG PAFAKHL NK DLLNLMLNCP MSDHMQETLM ILNEKIPSLE STVNSRILNL LSISLSGEKF IQSNQYDF N NQFSIEKARK SRNQSFMKKT GESNDDITDA QILIQCFKML QLIHHQYSLT EFVRLITIS YIEHEDSSVR KLAALTSCDL FIKDDICKQT SVHALHSVSE VLSKLLMIAI TDPVAEIRLE ILQHLGSNF DPQLAQPDNL RLLFMALNDE IFGIQLEAIK IIGRLSSVNP AYVVPSLRKT L LELLTQLK FSNMPKKKEE SATLLCTLIN SSDEVAKPYI DPILDVILPK CQDASSAVAS TA LKVLGEL SVVGGKEMTR YLKELMPLII NTFQDQSNSF KRDAALTTLG QLAASSGYVV GPL LDYPEL LGILINILKT ENNPHIRRGT VRLIGILGAL DPYKHREIEV TSNSKSSVEQ NAPS IDIAL LMQGVSPSND EYYPTVVIHN LMKILNDPSL SIHHTAAIQA IMHIFQNLGL RCVSF LDQI IPGIILVMRS CPPSQLDFYF QQLGSLISIV KQHIRPHVEK IYGVIREFFP IIKLQI TII SVIESISKAL EGEFKRFVPE TLTFFLDILE NDQSNKRIVP IRILKSLVTF GPNLEDY SH LIMPIVVRMT EYSAGSLKKI SIITLGRLAK NINLSEMSSR IVQALVRILN NGDRELTK A TMNTLSLLLL QLGTDFVVFV PVINKALLRN RIQHSVYDQL VNKLLNNECL PTNIIFDKE NEVPERKNYE DEMQVTKLPV NQNILKNAWY CSQQKTKEDW QEWIRRLSIQ LLKESPSACL RSCSSLVSV YYPLARELFN ASFSSCWVEL QTSYQEDLIQ ALCKALSSSE NPPEIYQMLL N LVEFMEHD DKPLPIPIHT LGKYAQKCHA FAKALHYKEV EFLEEPKNST IEALISINNQ LH QTDSAIG ILKHAQQHNE LQLKETWYEK LQRWEDALAA YNEKEAAGED SVEVMMGKLR SLY ALGEWE ELSKLASEKW GTAKPEVKKA MAPLAAGAAW GLEQWDEIAQ YTSVMKSQSP DKEF YDAIL CLHRNNFKKA EVHIFNARDL LVTELSALVN ESYNRAYNVV VRAQIIAELE EIIKY KKLP QNSDKRLTMR ETWNTRLLGC QKNIDVWQRI LRVRSLVIKP KEDAQVRIKF ANLCRK SGR MALAKKVLNT LLEETDDPDH PNTAKASPPV VYAQLKYLWA TGLQDEALKQ LINFTSR MA HDLGLDPNNM IAQSVPQQSK RVPRHVEDYT KLLARCFLKQ GEWRVCLQPK WRLSNPDS I LGSYLLATHF DNTWYKAWHN WALANFEVIS MLTSVSKKKQ EGSDASSVTD INEFDNGMI GVNTFDAKEV HYSSNLIHRH VIPAIKGFFH SISLSESSSL QDALRLLTLW FTFGGIPEAT QAMHEGFNL IQIGTWLEVL PQLISRIHQP NQIVSRSLLS LLSDLGKAHP QALVYPLMVA I KSESLSRQ KAALSIIEKM RIHSPVLVDQ AELVSHELIR MAVLWHEQWY EGLDDASRQF FG EHNTEKM FAALEPLYEM LKRGPETLRE ISFQNSFGRD LNDAYEWLMN YKKSKDVSNL NQA WDIYYN VFRKIGKQLP QLQTLELQHV SPKLLSAHDL ELAVPGTRAS GGKPIVKISK FEPV FSVIS SKQRPRKFCI KGSDGKDYKY VLKGHEDIRQ DSLVMQLFGL VNTLLQNDAE CFRRH LDIQ QYPAIPLSPK SGLLGWVPNS DTFHVLIREH REAKKIPLNI EHWVMLQMAP DYDNLT LLQ KVEVFTYALN NTEGQDLYKV LWLKSRSSET WLERRTTYTR SLAVMSMTGY ILGLGDR HP SNLMLDRITG KVIHIDFGDC FEAAILREKF PEKVPFRLTR MLTYAMEVSG IEGSFRIT C ENVMKVLRDN KGSLMAILEA FAFDPLINWG FDLPTKKIEE ETGIQLPVMN ANELLSNGA ITEEEVQRVE NEHKNAIRNA RAMLVLKRIT DKLTGNDIRR FNDLDVPEQV DKLIQQATSV ENLCQHYIG WCPFW |
-分子 #3: Target of rapamycin complex subunit LST8
| 分子 | 名称: Target of rapamycin complex subunit LST8 / タイプ: protein_or_peptide / ID: 3 詳細: S. cerevisiae TOROIDs (TORC1 Organized in Inhibited Domains) in the sample are composed of Kog1 (P38873), Lst8 (P41318) and Tor2 (P32600). 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 配列 | 文字列: MSVILVSAGY DHTIRFWEAL TGVCSRTIQH SDSQVNRLEI TNDKKLLATA GHQNVRLYDI RTTNPNPVA SFEGHRGNVT SVSFQQDNRW MVTSSEDGTI KVWDVRSPSI PRNYKHNAPV N EVVIHPNQ GELISCDRDG NIRIWDLGEN QCTHQLTPED DTSLQSLSMA ...文字列: MSVILVSAGY DHTIRFWEAL TGVCSRTIQH SDSQVNRLEI TNDKKLLATA GHQNVRLYDI RTTNPNPVA SFEGHRGNVT SVSFQQDNRW MVTSSEDGTI KVWDVRSPSI PRNYKHNAPV N EVVIHPNQ GELISCDRDG NIRIWDLGEN QCTHQLTPED DTSLQSLSMA SDGSMLAAAN TK GNCYVWE MPNHTDASHL KPVTKFRAHS TYITRILLSS DVKHLATCSA DHTARVWSID DDF KLETTL DGHQRWVWDC AFSADSAYLV TASSDHYVRL WDLSTREIVR QYGGHHKGAV CVAL NDV |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | らせん対称体再構成法 |
| 試料の集合状態 | filament |
- 試料調製
試料調製
| 濃度 | 0.02 mg/mL | ||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.5 構成要素:
詳細: 50 mM MES-NaOH pH 6.0, 5 mM CHAPS, 600 mM KCl, 0,5 mM DTT, Phosphatase Inhibitor Mix, Complete Protease Inhibitor Cocktail (Roche; 1 tablet/ 50 ml) and 1 mM PMSF) | ||||||||||||||||||||||||
| グリッド | モデル: PELCO Ultrathin Carbon with Lacey Carbon / 前処理 - タイプ: GLOW DISCHARGE | ||||||||||||||||||||||||
| 凍結 | 凍結剤: ETHANE / 装置: HOMEMADE PLUNGER |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / デジタル化 - 画像ごとのフレーム数: 2-16 / 実像数: 4901 / 平均露光時間: 8.0 sec. / 平均電子線量: 20.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: OTHER / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 0.8 µm / 倍率(公称値): 37000 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | プロトコル: RIGID BODY FIT |
|---|
 ムービー
ムービー コントローラー
コントローラー






 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)