+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Stacked stretched Dunaliella PSII | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | green algae / photosystem II / thylakoid / oxygen evolving complex / cryo-EM / stacking / PHOTOSYNTHESIS | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報oxygen evolving activity / photosystem II reaction center / photosystem II / oxidoreductase activity, acting on diphenols and related substances as donors, oxygen as acceptor / photosynthetic electron transport chain / response to herbicide / photosystem II / chlorophyll binding / photosynthetic electron transport in photosystem II / phosphate ion binding ...oxygen evolving activity / photosystem II reaction center / photosystem II / oxidoreductase activity, acting on diphenols and related substances as donors, oxygen as acceptor / photosynthetic electron transport chain / response to herbicide / photosystem II / chlorophyll binding / photosynthetic electron transport in photosystem II / phosphate ion binding / photosynthesis, light reaction / chloroplast thylakoid membrane / : / photosynthesis / electron transfer activity / protein stabilization / iron ion binding / heme binding / metal ion binding 類似検索 - 分子機能 | |||||||||
| 生物種 |  Dunaliella salina (しおひげむし) Dunaliella salina (しおひげむし) | |||||||||
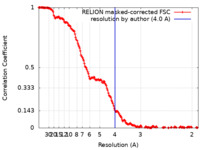
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 4.0 Å | |||||||||
 データ登録者 データ登録者 | Caspy I / Fadeeva M | |||||||||
| 資金援助 |  イスラエル, 2件 イスラエル, 2件
| |||||||||
 引用 引用 |  ジャーナル: Elife / 年: 2023 ジャーナル: Elife / 年: 2023タイトル: Structure of photosystem II reveals conformational flexibility of stacked and unstacked supercomplexes. 著者: Ido Caspy / Maria Fadeeva / Yuval Mazor / Nathan Nelson /   要旨: Photosystem II (PSII) generates an oxidant whose redox potential is high enough to enable water oxidation , a substrate so abundant that it assures a practically unlimited electron source for life on ...Photosystem II (PSII) generates an oxidant whose redox potential is high enough to enable water oxidation , a substrate so abundant that it assures a practically unlimited electron source for life on earth . Our knowledge on the mechanism of water photooxidation was greatly advanced by high-resolution structures of prokaryotic PSII . Here, we show high-resolution cryogenic electron microscopy (cryo-EM) structures of eukaryotic PSII from the green alga at two distinct conformations. The conformers are also present in stacked PSII, exhibiting flexibility that may be relevant to the grana formation in chloroplasts of the green lineage. CP29, one of PSII associated light-harvesting antennae, plays a major role in distinguishing the two conformations of the supercomplex. We also show that the stacked PSII dimer, a form suggested to support the organisation of thylakoid membranes , can appear in many different orientations providing a flexible stacking mechanism for the arrangement of grana stacks in thylakoids. Our findings provide a structural basis for the heterogenous nature of the eukaryotic PSII on multiple levels. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_13455.map.gz emd_13455.map.gz | 392.2 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-13455-v30.xml emd-13455-v30.xml emd-13455.xml emd-13455.xml | 45.5 KB 45.5 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_13455_fsc.xml emd_13455_fsc.xml | 17.1 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_13455.png emd_13455.png | 100.2 KB | ||
| Filedesc metadata |  emd-13455.cif.gz emd-13455.cif.gz | 10.4 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-13455 http://ftp.pdbj.org/pub/emdb/structures/EMD-13455 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-13455 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-13455 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_13455.map.gz / 形式: CCP4 / 大きさ: 421.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_13455.map.gz / 形式: CCP4 / 大きさ: 421.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.96 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
- 試料の構成要素
試料の構成要素
+全体 : Dunaliella salina Photosystem II complex
+超分子 #1: Dunaliella salina Photosystem II complex
+分子 #1: Photosystem II protein D1
+分子 #2: Photosystem II CP47 reaction center protein
+分子 #3: Photosystem II reaction center protein Ycf12
+分子 #4: Photosystem II CP43 reaction center protein
+分子 #5: Photosystem II D2 protein
+分子 #6: Cytochrome b559 subunit alpha
+分子 #7: Cytochrome b559 subunit beta
+分子 #8: Photosystem II reaction center protein H
+分子 #9: Photosystem II reaction center protein I
+分子 #10: Photosystem II reaction center protein J
+分子 #11: Photosystem II reaction center protein K
+分子 #12: Photosystem II reaction center protein L
+分子 #13: Photosystem II reaction center protein M
+分子 #14: PsbO
+分子 #15: PsbP
+分子 #16: Photosystem II reaction center protein T
+分子 #17: PsbW
+分子 #18: PsbX
+分子 #19: PsbZ
+分子 #20: LHCII M3
+分子 #21: LHCII M2
+分子 #22: CP29
+分子 #23: CP26
+分子 #24: LHCII M1
+分子 #25: PsbU
+分子 #26: Photosystem II reaction center protein M
+分子 #27: CP29
+分子 #28: CA-MN4-O5 CLUSTER
+分子 #29: FE (II) ION
+分子 #30: CHLORIDE ION
+分子 #31: CHLOROPHYLL A
+分子 #32: PHEOPHYTIN A
+分子 #33: BETA-CAROTENE
+分子 #34: 1,2-DI-O-ACYL-3-O-[6-DEOXY-6-SULFO-ALPHA-D-GLUCOPYRANOSYL]-SN-GLYCEROL
+分子 #35: 1,2-DISTEAROYL-MONOGALACTOSYL-DIGLYCERIDE
+分子 #36: SODIUM ION
+分子 #37: (1~{S})-3,5,5-trimethyl-4-[(1~{E},3~{E},5~{E},7~{E},9~{E},11~{E},...
+分子 #38: DIACYL GLYCEROL
+分子 #39: GLYCEROL
+分子 #40: DIGALACTOSYL DIACYL GLYCEROL (DGDG)
+分子 #41: 1,2-DIPALMITOYL-PHOSPHATIDYL-GLYCEROLE
+分子 #42: trimethyl-[(2~{R})-1-oxidanyl-1-oxidanylidene-4-[(2~{S})-2-[(1~{S...
+分子 #43: BICARBONATE ION
+分子 #44: 2,3-DIMETHYL-5-(3,7,11,15,19,23,27,31,35-NONAMETHYL-2,6,10,14,18,...
+分子 #45: PROTOPORPHYRIN IX CONTAINING FE
+分子 #46: (3R)-beta,beta-caroten-3-ol
+分子 #47: CHLOROPHYLL B
+分子 #48: (3R,3'R,6S)-4,5-DIDEHYDRO-5,6-DIHYDRO-BETA,BETA-CAROTENE-3,3'-DIOL
+分子 #49: (3S,5R,6S,3'S,5'R,6'S)-5,6,5',6'-DIEPOXY-5,6,5',6'- TETRAHYDRO-BE...
+分子 #50: (1R,3R)-6-{(3E,5E,7E,9E,11E,13E,15E,17E)-18-[(1S,4R,6R)-4-HYDROXY...
+分子 #51: (2S)-3-{[(R)-(2-aminoethoxy)(hydroxy)phosphoryl]oxy}-2-hydroxypro...
+分子 #52: 1,2-DIACYL-GLYCEROL-3-SN-PHOSPHATE
+分子 #53: SPHINGOSINE
+分子 #54: Tripalmitoylglycerol
+分子 #55: DODECYL-BETA-D-MALTOSIDE
+分子 #56: ERGOSTEROL
+分子 #57: PHOSPHATIDYLETHANOLAMINE
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 6.5 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 実像数: 13586 / 平均電子線量: 51.81 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー
















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)