+ Open data
Open data
- Basic information
Basic information
| Entry | Database: EMDB / ID: EMD-13194 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| Title | Cryo-EM structure of human TTYH2 in GDN | |||||||||
 Map data Map data | TTYH2 in glyco diosgenin, full map | |||||||||
 Sample Sample |
| |||||||||
 Keywords Keywords | Membrane protein / lipid metabolism / lipid transport | |||||||||
| Function / homology |  Function and homology information Function and homology informationvolume-sensitive chloride channel activity / L-glutamate transmembrane transport / intracellularly calcium-gated chloride channel activity / chloride channel complex / Stimuli-sensing channels / calcium ion binding / plasma membrane Similarity search - Function | |||||||||
| Biological species |  Homo sapiens (human) Homo sapiens (human) | |||||||||
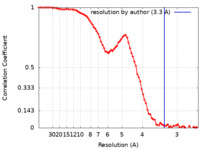
| Method | single particle reconstruction / cryo EM / Resolution: 3.3 Å | |||||||||
 Authors Authors | Sukalskaia A / Straub MS / Sawicka M / Deneka D / Dutzler R | |||||||||
| Funding support |  Switzerland, 1 items Switzerland, 1 items
| |||||||||
 Citation Citation |  Journal: Nat Commun / Year: 2021 Journal: Nat Commun / Year: 2021Title: Cryo-EM structures of the TTYH family reveal a novel architecture for lipid interactions. Authors: Anastasiia Sukalskaia / Monique S Straub / Dawid Deneka / Marta Sawicka / Raimund Dutzler /  Abstract: The Tweety homologs (TTYHs) are members of a conserved family of eukaryotic membrane proteins that are abundant in the brain. The three human paralogs were assigned to function as anion channels that ...The Tweety homologs (TTYHs) are members of a conserved family of eukaryotic membrane proteins that are abundant in the brain. The three human paralogs were assigned to function as anion channels that are either activated by Ca or cell swelling. To uncover their unknown architecture and its relationship to function, we have determined the structures of human TTYH1-3 by cryo-electron microscopy. All structures display equivalent features of a dimeric membrane protein that contains five transmembrane segments and an extended extracellular domain. As none of the proteins shows attributes reminiscent of an anion channel, we revisited functional experiments and did not find any indication of ion conduction. Instead, we find density in an extended hydrophobic pocket contained in the extracellular domain that emerges from the lipid bilayer, which suggests a role of TTYH proteins in the interaction with lipid-like compounds residing in the membrane. | |||||||||
| History |
|
- Structure visualization
Structure visualization
| Movie |
 Movie viewer Movie viewer |
|---|---|
| Structure viewer | EM map:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| Supplemental images |
- Downloads & links
Downloads & links
-EMDB archive
| Map data |  emd_13194.map.gz emd_13194.map.gz | 38 MB |  EMDB map data format EMDB map data format | |
|---|---|---|---|---|
| Header (meta data) |  emd-13194-v30.xml emd-13194-v30.xml emd-13194.xml emd-13194.xml | 15.3 KB 15.3 KB | Display Display |  EMDB header EMDB header |
| FSC (resolution estimation) |  emd_13194_fsc.xml emd_13194_fsc.xml | 10.1 KB | Display |  FSC data file FSC data file |
| Images |  emd_13194.png emd_13194.png | 5.2 MB | ||
| Masks |  emd_13194_msk_1.map emd_13194_msk_1.map | 40.6 MB |  Mask map Mask map | |
| Filedesc metadata |  emd-13194.cif.gz emd-13194.cif.gz | 5.7 KB | ||
| Others |  emd_13194_half_map_1.map.gz emd_13194_half_map_1.map.gz emd_13194_half_map_2.map.gz emd_13194_half_map_2.map.gz | 36.7 MB 36.9 MB | ||
| Archive directory |  http://ftp.pdbj.org/pub/emdb/structures/EMD-13194 http://ftp.pdbj.org/pub/emdb/structures/EMD-13194 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-13194 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-13194 | HTTPS FTP |
-Related structure data
| Related structure data |  7p54MC  7p5cC  7p5jC  7p5mC M: atomic model generated by this map C: citing same article ( |
|---|---|
| Similar structure data |
- Links
Links
| EMDB pages |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- Map
Map
| File |  Download / File: emd_13194.map.gz / Format: CCP4 / Size: 40.6 MB / Type: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) Download / File: emd_13194.map.gz / Format: CCP4 / Size: 40.6 MB / Type: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Annotation | TTYH2 in glyco diosgenin, full map | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Projections & slices | Image control
Images are generated by Spider. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Voxel size | X=Y=Z: 1.302 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Density |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Symmetry | Space group: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Details | EMDB XML:
CCP4 map header:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-Supplemental data
-Mask #1
| File |  emd_13194_msk_1.map emd_13194_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Projections & Slices |
| ||||||||||||
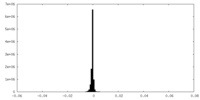
| Density Histograms |
-Half map: TTYH2 in glyco diosgenin, half-map 1
| File | emd_13194_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Annotation | TTYH2 in glyco diosgenin, half-map 1 | ||||||||||||
| Projections & Slices |
| ||||||||||||
| Density Histograms |
-Half map: TTYH2 in glyco diosgenin, half-map 2
| File | emd_13194_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Annotation | TTYH2 in glyco diosgenin, half-map 2 | ||||||||||||
| Projections & Slices |
| ||||||||||||
| Density Histograms |
- Sample components
Sample components
-Entire : TTYH2
| Entire | Name: TTYH2 |
|---|---|
| Components |
|
-Supramolecule #1: TTYH2
| Supramolecule | Name: TTYH2 / type: complex / ID: 1 / Parent: 0 / Macromolecule list: #1 |
|---|---|
| Source (natural) | Organism:  Homo sapiens (human) Homo sapiens (human) |
| Molecular weight | Theoretical: 119 KDa |
-Macromolecule #1: Protein tweety homolog 2
| Macromolecule | Name: Protein tweety homolog 2 / type: protein_or_peptide / ID: 1 / Number of copies: 2 / Enantiomer: LEVO |
|---|---|
| Source (natural) | Organism:  Homo sapiens (human) Homo sapiens (human) |
| Molecular weight | Theoretical: 59.573879 KDa |
| Recombinant expression | Organism:  Homo sapiens (human) Homo sapiens (human) |
| Sequence | String: SQAARVDYIA PWWVVWLHSV PHVGLRLQPV NSTFSPGDES YQESLLFLGL VAAVCLGLNL IFLVAYLVCA CHCRRDDAVQ TKQHHSCCI TWTAVVAGLI CCAAVGVGFY GNSETNDGAY QLMYSLDDAN HTFSGIDALV SGTTQKMKVD LEQHLARLSE I FAARGDYL ...String: SQAARVDYIA PWWVVWLHSV PHVGLRLQPV NSTFSPGDES YQESLLFLGL VAAVCLGLNL IFLVAYLVCA CHCRRDDAVQ TKQHHSCCI TWTAVVAGLI CCAAVGVGFY GNSETNDGAY QLMYSLDDAN HTFSGIDALV SGTTQKMKVD LEQHLARLSE I FAARGDYL QTLKFIQQMA GSVVVQLSGL PVWREVTMEL TKLSDQTGYV EYYRWLSYLL LFILDLVICL IACLGLAKRS KC LLASMLC CGALSLLLSW ASLAADGSAA VATSDFCVAP DTFILNVTEG QISTEVTRYY LYCSQSGSSP FQQTLTTFQR ALT TMQIQV AGLLQFAVPL FSTAEEDLLA IQLLLNSSES SLHQLTAMVD CRGLHKDYLD ALAGICYDGL QGLLYLGLFS FLAA LAFST MICAGPRAWK HFTTRNRDYD DIDDDDPFNP QAWRMAAHSP PRGQLHSFCS YSSGLGSQTS LQPPAQTISN APVSE YMNQ AMLFGRNPRY ENVPLIGRAS PPPTYSPSMR ATYLSVADEH LRHYGNQFPA ALEVLFQ UniProtKB: Protein tweety homolog 2 |
-Macromolecule #3: 2-acetamido-2-deoxy-beta-D-glucopyranose
| Macromolecule | Name: 2-acetamido-2-deoxy-beta-D-glucopyranose / type: ligand / ID: 3 / Number of copies: 4 / Formula: NAG |
|---|---|
| Molecular weight | Theoretical: 221.208 Da |
| Chemical component information |  ChemComp-NAG: |
-Experimental details
-Structure determination
| Method | cryo EM |
|---|---|
 Processing Processing | single particle reconstruction |
| Aggregation state | particle |
- Sample preparation
Sample preparation
| Buffer | pH: 7.5 Component:
| |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Grid | Model: Quantifoil R1.2/1.3 / Material: GOLD / Mesh: 200 | |||||||||||||||
| Vitrification | Cryogen name: ETHANE-PROPANE / Instrument: FEI VITROBOT MARK IV |
- Electron microscopy
Electron microscopy
| Microscope | FEI TITAN KRIOS |
|---|---|
| Image recording | Film or detector model: GATAN K3 BIOQUANTUM (6k x 4k) / Average electron dose: 61.0 e/Å2 |
| Electron beam | Acceleration voltage: 300 kV / Electron source:  FIELD EMISSION GUN FIELD EMISSION GUN |
| Electron optics | Illumination mode: SPOT SCAN / Imaging mode: BRIGHT FIELD |
| Experimental equipment |  Model: Titan Krios / Image courtesy: FEI Company |
 Movie
Movie Controller
Controller














 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)