+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-1319 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Octomeric pyruvate-ferredoxin oxidoreductase from Desulfovibrio vulgaris. | |||||||||
 マップデータ マップデータ | pyrovat-ferredoxin oxidoreductase from Desulfovibrio vulgaris Hildenborough | |||||||||
 試料 試料 |
| |||||||||
| 生物種 |  Desulfovibrio vulgaris str. Hildenborough (バクテリア) Desulfovibrio vulgaris str. Hildenborough (バクテリア) | |||||||||
| 手法 | 単粒子再構成法 / ネガティブ染色法 / 解像度: 17.0 Å | |||||||||
 データ登録者 データ登録者 | Garczarek F / Dong M / Typke D / Witkowska E / Hazen TC / Nogales E / Glaeser R | |||||||||
 引用 引用 |  ジャーナル: J Struct Biol / 年: 2007 ジャーナル: J Struct Biol / 年: 2007タイトル: Octomeric pyruvate-ferredoxin oxidoreductase from Desulfovibrio vulgaris. 著者: Florian Garczarek / Ming Dong / Dieter Typke / H Ewa Witkowska / Terry C Hazen / Eva Nogales / Mark D Biggin / Robert M Glaeser /  要旨: Pyruvate-ferredoxin oxidoreductatse (PFOR) carries out the central step in oxidative decarboxylation of pyruvate to acetyl-CoA. We have purified this enzyme from Desulfovibrio vulgaris Hildenborough ...Pyruvate-ferredoxin oxidoreductatse (PFOR) carries out the central step in oxidative decarboxylation of pyruvate to acetyl-CoA. We have purified this enzyme from Desulfovibrio vulgaris Hildenborough (DvH) as part of a systematic characterization of as many multiprotein complexes as possible for this organism, and the three-dimensional structure of this enzyme has been determined by a combination of electron microscopy (EM), single particle image analysis, homology modeling and computational molecular docking. Our results show that the 1MDa DvH PFOR complex is a homo-octomer, or more precisely, a tetramer of the dimeric form of the related enzyme found in Desulfovibrio africanus (Da), with which it shares a sequence identity of 69%. Our homology model of the DvH PFOR dimer is based on the Da PFOR X-ray structure. Docking of this model into our 17A resolution EM-reconstruction of negatively stained DvH PFOR octomers strongly suggests that the difference in oligomerization state for the two species is due to the insertion of a single valine residue (Val383) within a surface loop of the DvH enzyme. This study demonstrates that the strategy of intermediate resolution EM reconstruction coupled to homology modeling and docking can be powerful enough to infer the functionality of single amino acid residues. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_1319.map.gz emd_1319.map.gz | 14 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-1319-v30.xml emd-1319-v30.xml emd-1319.xml emd-1319.xml | 8.6 KB 8.6 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  1319.gif 1319.gif | 50.3 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-1319 http://ftp.pdbj.org/pub/emdb/structures/EMD-1319 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-1319 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-1319 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_1319_validation.pdf.gz emd_1319_validation.pdf.gz | 221.1 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_1319_full_validation.pdf.gz emd_1319_full_validation.pdf.gz | 220.2 KB | 表示 | |
| XML形式データ |  emd_1319_validation.xml.gz emd_1319_validation.xml.gz | 5.7 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1319 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1319 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1319 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1319 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_1319.map.gz / 形式: CCP4 / 大きさ: 15.3 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_1319.map.gz / 形式: CCP4 / 大きさ: 15.3 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | pyrovat-ferredoxin oxidoreductase from Desulfovibrio vulgaris Hildenborough | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 2.56 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : pyruvate-ferredoxin oxidoreductase from Desulfovibrio vulgaris Hi...
| 全体 | 名称: pyruvate-ferredoxin oxidoreductase from Desulfovibrio vulgaris Hildenborough |
|---|---|
| 要素 |
|
-超分子 #1000: pyruvate-ferredoxin oxidoreductase from Desulfovibrio vulgaris Hi...
| 超分子 | 名称: pyruvate-ferredoxin oxidoreductase from Desulfovibrio vulgaris Hildenborough タイプ: sample / ID: 1000 / 集合状態: homooctomer / Number unique components: 1 |
|---|---|
| 分子量 | 理論値: 1.056 MDa |
-分子 #1: pyruvate-ferredoxin oxidoreductase
| 分子 | 名称: pyruvate-ferredoxin oxidoreductase / タイプ: protein_or_peptide / ID: 1 / Name.synonym: PFOR / コピー数: 8 / 集合状態: homooctomer / 組換発現: Yes |
|---|---|
| 由来(天然) | 生物種:  Desulfovibrio vulgaris str. Hildenborough (バクテリア) Desulfovibrio vulgaris str. Hildenborough (バクテリア)株: ATCC 29579 |
| 分子量 | 実験値: 132 MDa / 理論値: 132 MDa |
| 組換発現 | 生物種:  Desulfovibrio vulgaris str. Hildenborough (バクテリア) Desulfovibrio vulgaris str. Hildenborough (バクテリア) |
-実験情報
-構造解析
| 手法 | ネガティブ染色法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.05 mg/mL |
|---|---|
| 緩衝液 | pH: 7.6 / 詳細: 10mM HEPES, 2mM DTT, 0.01% NP40 |
| 染色 | タイプ: NEGATIVE / 詳細: 5% ammonium molybdate, 1% trehalose, pH7 |
| グリッド | 詳細: 400 mesh copper grid coated with carbon layer |
| 凍結 | 凍結剤: NONE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TECNAI 12 |
|---|---|
| 撮影 | カテゴリ: FILM / フィルム・検出器のモデル: KODAK SO-163 FILM / デジタル化 - スキャナー: OTHER / デジタル化 - サンプリング間隔: 12.7 µm / 実像数: 19 |
| 電子線 | 加速電圧: 120 kV / 電子線源: LAB6 |
| 電子光学系 | 倍率(補正後): 49700 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 6.2 mm / 最大 デフォーカス(公称値): 1.6 µm / 最小 デフォーカス(公称値): 0.6 µm / 倍率(公称値): 50000 |
| 試料ステージ | 試料ホルダー: side entry / 試料ホルダーモデル: OTHER |
- 画像解析
画像解析
| CTF補正 | 詳細: defocus groups |
|---|---|
| 最終 再構成 | 想定した対称性 - 点群: D4 (2回x4回 2面回転対称) 解像度のタイプ: BY AUTHOR / 解像度: 17.0 Å / 解像度の算出法: FSC 0.5 CUT-OFF / ソフトウェア - 名称: EMAN, SPIDER / 使用した粒子像数: 12402 |
 ムービー
ムービー コントローラー
コントローラー



 UCSF Chimera
UCSF Chimera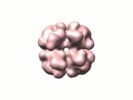






 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)





















